Fylogenetiskt träd
| Del av en serie om |
| Evolutionsbiologi |
|---|

|

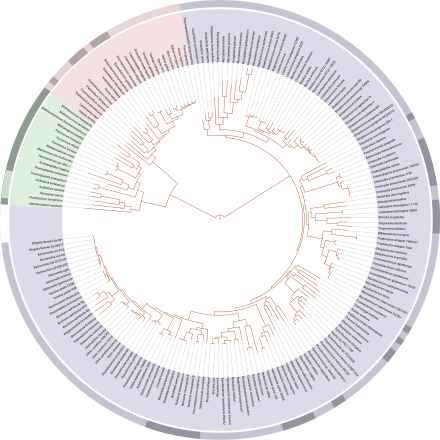
Ett fylogenetiskt träd (även fylogeni eller evolutionärt träd ) är ett grendiagram eller ett träd som visar de evolutionära förhållandena mellan olika biologiska arter eller andra enheter baserat på likheter och skillnader i deras fysiska eller genetiska egenskaper. Allt liv på jorden är en del av ett enda fylogenetiskt träd, vilket indikerar gemensamma anor .
I ett rotat fylogenetiskt träd representerar varje nod med ättlingar den antagna senaste gemensamma förfadern till dessa ättlingar, och kantlängderna i vissa träd kan tolkas som tidsuppskattningar. Varje nod kallas en taxonomisk enhet. Interna noder kallas generellt för hypotetiska taxonomiska enheter, eftersom de inte kan observeras direkt. Träd är användbara inom biologiområden som bioinformatik , systematik och fylogenetik . Orotade träd illustrerar endast släktskapet mellan bladnoderna och kräver inte att den förfäders roten är känd eller antagen.
Historia
Idén om ett livsträd uppstod från uråldriga föreställningar om en stegliknande progression från lägre till högre livsformer ( som i Great Chain of Being ). Tidiga representationer av "förgrenade" fylogenetiska träd inkluderar ett "paleontologiskt diagram" som visar de geologiska förhållandena mellan växter och djur i boken Elementary Geology , av Edward Hitchcock (första upplagan: 1840).
Charles Darwin presenterade ett diagrammatiskt evolutionärt "träd" i sin bok från 1859 On the Origin of Species . Över ett sekel senare använder evolutionsbiologer fortfarande träddiagram för att avbilda evolutionen eftersom sådana diagram effektivt förmedlar konceptet att artbildning sker genom adaptiv och halvslumpmässig uppdelning av linjer.
Termen fylogenetisk , eller fylogeni , härstammar från de två antika grekiska orden φῦλον ( phûlon ), som betyder "ras, härstamning", och γένεσις ( génesis ), som betyder "ursprung, källa".
Egenskaper
Rotat träd

Ett rotat fylogenetiskt träd (se två bilder överst) är ett riktat träd med en unik nod - roten - som motsvarar den (vanligtvis tillskrivna ) senaste gemensamma förfadern till alla enheter vid trädets löv. Rotnoden har ingen föräldernod, utan fungerar som förälder till alla andra noder i trädet. Roten är därför en nod av grad 2, medan andra interna noder har en minimigrad på 3 (där "grad" här syftar på det totala antalet inkommande och utgående flanker).
Den vanligaste metoden för att rota träd är användningen av en okontroversiell utgrupp - tillräckligt nära för att tillåta slutsatser från egenskapersdata eller molekylär sekvensering, men tillräckligt långt för att vara en tydlig utgrupp. En annan metod är mittpunktsrotning, eller ett träd kan också rotas genom att använda en icke-stationär substitutionsmodell .
Orotat träd
Orotade träd illustrerar släktskapet mellan bladnoderna utan att göra antaganden om härkomst. De kräver inte att släktroten är känd eller härledd. Orotade träd kan alltid genereras från rotade genom att helt enkelt utelämna roten. Däremot kräver att man kan sluta sig till roten till ett orotat träd ett sätt att identifiera härkomst. Detta görs normalt genom att inkludera en utgrupp i indata så att roten nödvändigtvis är mellan utgruppen och resten av taxan i trädet, eller genom att införa ytterligare antaganden om de relativa utvecklingshastigheterna på varje gren, såsom en applikation av den molekylära klockhypotesen .
Bifurcating kontra multifurcating
Både rotade och orotade träd kan vara antingen grenade eller flergrenade. Ett rotat förgrenat träd har exakt två avkomlingar som härrör från varje inre nod (det vill säga det bildar ett binärt träd ), och ett orotat förgrenat träd har formen av ett orotat binärt träd , ett fritt träd med exakt tre grannar vid varje inre nod. Däremot kan ett rotat multifurcating-träd ha fler än två barn vid vissa noder och ett orootat multifurcating-träd kan ha mer än tre grannar vid vissa noder.
Märkt kontra omärkt
Både rotade och orotade träd kan vara antingen märkta eller omärkta. Ett märkt träd har specifika värden tilldelade sina löv, medan ett omärkt träd, ibland kallat en trädform, endast definierar en topologi. Vissa sekvensbaserade träd byggda från ett litet genomiskt lokus, såsom Phylotree, har interna noder märkta med antagna förfäders haplotyper.
Räkna upp träd

Antalet möjliga träd för ett givet antal lövnoder beror på den specifika typen av träd, men det finns alltid fler märkta än omärkta träd, fler träd med flera grenar än grenade träd och mer rotade än orotade träd. Den sista distinktionen är den mest biologiskt relevanta; det uppstår eftersom det finns många ställen på ett orotat träd att sätta roten på. För grenade märkta träd är det totala antalet rotade träd:
- displaystyle , representerar antalet lövnoder.
För grenade märkta träd är det totala antalet orotade träd:
- displaystyle .
Bland märkta bifurcating träd är antalet orotade träd med löv lika med antalet rotade träd med löv.
Antalet rotade träd växer snabbt som en funktion av antalet spetsar. För 10 tips finns det fler än möjliga grenade träd, och antalet träd med flera grenar ökar snabbare, med ca. 7 gånger så många av de senare som av de förra.
|
Märkt blad |
Binära orotade träd |
Binära rotade träd |
Multifurcating rotade träd |
Alla möjliga rotade träd |
|---|---|---|---|---|
| 1 | 1 | 1 | 0 | 1 |
| 2 | 1 | 1 | 0 | 1 |
| 3 | 1 | 3 | 1 | 4 |
| 4 | 3 | 15 | 11 | 26 |
| 5 | 15 | 105 | 131 | 236 |
| 6 | 105 | 945 | 1 807 | 2,752 |
| 7 | 945 | 10 395 | 28,813 | 39,208 |
| 8 | 10 395 | 135,135 | 524,897 | 660 032 |
| 9 | 135,135 | 2 027 025 | 10,791,887 | 12,818,912 |
| 10 | 2 027 025 | 34,459,425 | 247,678,399 | 282,137,824 |
Speciella trädtyper
Dendrogram
Ett dendrogram är ett allmänt namn för ett träd, oavsett om det är fylogenetiskt eller inte, och därmed också för den schematiska representationen av ett fylogenetiskt träd.
Kladogram
Ett kladogram representerar bara ett grenmönster; dvs dess grenlängder representerar inte tid eller relativ mängd teckenförändring, och dess interna noder representerar inte förfäder.

Filogram
Ett fylogram är ett fylogenetiskt träd som har grenlängder som är proportionella mot mängden karaktärsförändring.
Ett kronogram är ett fylogenetiskt träd som uttryckligen representerar tiden genom dess grenlängder.
Dahlgrenogram
Ett Dahlgrenogram är ett diagram som representerar ett tvärsnitt av ett fylogenetiskt träd.
Fylogenetiskt nätverk
Ett fylogenetiskt nätverk är strängt taget inte ett träd, utan snarare en mer allmän graf , eller en riktad acyklisk graf i fallet med rotade nätverk. De används för att övervinna några av de begränsningar som träden har.
Spindeldiagram
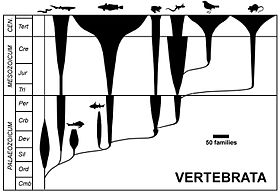
Ett spindeldiagram, eller bubbeldiagram, kallas ofta ett romerogram, efter dess popularisering av den amerikanske paleontologen Alfred Romer . Den representerar taxonomisk mångfald (horisontell bredd) mot geologisk tid (vertikal axel) för att återspegla variationen i överflöd av olika taxa över tiden. Ett spindeldiagram är dock inte ett evolutionärt träd: de taxonomiska spindlarna skymmer de faktiska förhållandena mellan moder-taxonen och dotter-taxonen och har nackdelen att involvera paraphylin av föräldragruppen. Denna typ av diagram används inte längre i den form som ursprungligen föreslagits.
Livets korall
Darwin nämnde också att korallen kan vara en lämpligare metafor än trädet . Faktum är att fylogenetiska koraller är användbara för att skildra tidigare och nuvarande liv, och de har vissa fördelar jämfört med träd (anastomos tillåtna, etc.).
Konstruktion
Fylogenetiska träd sammansatta av ett icke-trivialt antal indatasekvenser konstrueras med hjälp av beräkningsfylogenetiska metoder. Avståndsmatrismetoder som grannsammanfogning eller UPGMA , som beräknar genetiskt avstånd från flera sekvensanpassningar, är enklast att implementera, men åberopar inte en evolutionär modell. Många sekvensanpassningsmetoder som ClustalW skapar också träd genom att använda de enklare algoritmerna (dvs. de som baseras på avstånd) för trädkonstruktion. Maximal sparsamhet är en annan enkel metod för att uppskatta fylogenetiska träd, men innebär en implicit modell av evolution (dvs. sparsamhet). Mer avancerade metoder använder optimalitetskriteriet maximal sannolikhet , ofta inom en Bayesiansk ram , och tillämpar en explicit evolutionsmodell på fylogenetisk träduppskattning. Att identifiera det optimala trädet med många av dessa tekniker är NP-svårt , så heuristiska sök- och optimeringsmetoder används i kombination med trädbetygsfunktioner för att identifiera ett någorlunda bra träd som passar data.
Metoder för trädbyggnad kan bedömas utifrån flera kriterier:
- effektivitet (hur lång tid tar det att beräkna svaret, hur mycket minne behöver det?)
- makt (använder den data på ett bra sätt eller slösas information bort?)
- konsistens (kommer det att konvergera på samma svar upprepade gånger, om varje gång ges olika data för samma modellproblem?)
- robusthet (klarar den väl mot kränkningar av den underliggande modellens antaganden?)
- falsifierbarhet (varnar den oss när den inte är bra att använda, dvs när antaganden kränks?)
Trädbyggnadstekniker har också fått uppmärksamhet hos matematiker. Träd kan också byggas med hjälp av T-teori .
Filformat
Träd kan kodas i ett antal olika format, som alla måste representera den kapslade strukturen i ett träd. De kan eller kanske inte kodar grenlängder och andra funktioner. Standardiserade format är avgörande för att distribuera och dela träd utan att förlita sig på grafikutdata som är svåra att importera till befintlig programvara. Vanligt använda format är
Begränsningar av fylogenetisk analys
Även om fylogenetiska träd producerade på basis av sekvenserade gener eller genomiska data i olika arter kan ge evolutionär insikt, har dessa analyser viktiga begränsningar. Viktigast av allt är att träden som de genererar inte nödvändigtvis är korrekta – de representerar inte nödvändigtvis exakt den evolutionära historien för den inkluderade taxan. Som med alla vetenskapliga resultat är de föremål för förfalskning genom ytterligare studier (t.ex. insamling av ytterligare data, analys av befintliga data med förbättrade metoder). De data som de baseras på kan vara bullriga ; analysen kan förväxlas av genetisk rekombination , horisontell genöverföring , hybridisering mellan arter som inte var närmaste grannar på trädet innan hybridisering äger rum, konvergent evolution och konserverade sekvenser .
Det finns också problem med att basera en analys på en enskild typ av karaktär, såsom en enskild gen eller ett protein eller bara på morfologisk analys, eftersom sådana träd som är konstruerade från en annan orelaterade datakälla ofta skiljer sig från den första, och därför krävs stor försiktighet. att sluta sig till fylogenetiska samband mellan arter. Detta är mest sant för genetiskt material som är föremål för lateral genöverföring och rekombination , där olika haplotypblock kan ha olika historia. I dessa typer av analyser är utdataträdet för en fylogenetisk analys av en enskild gen en uppskattning av genens fylogeni (dvs. ett genträd) och inte fylogenin för taxa (dvs. artträd) som dessa karaktärer togs från , men helst bör båda vara mycket nära. Av denna anledning använder seriösa fylogenetiska studier i allmänhet en kombination av gener som kommer från olika genomiska källor (t.ex. från mitokondriella eller plastider kontra kärngenom), eller gener som skulle förväntas utvecklas under olika selektiva regimer, så att homoplasi ( falskt homologi ) skulle sannolikt inte vara ett resultat av naturligt urval.
När utdöda arter ingår som terminalnoder i en analys (snarare än att till exempel begränsa interna noder), anses de inte representera direkta förfäder till någon existerande art. Utdöda arter innehåller vanligtvis inte högkvalitativt DNA .
Utbudet av användbara DNA-material har utökats med framsteg inom extraktions- och sekvenseringsteknologier. Utveckling av teknologier som kan härleda sekvenser från mindre fragment, eller från rumsliga mönster av DNA-nedbrytningsprodukter, skulle ytterligare utöka intervallet av DNA som anses vara användbart.
Filogenetiska träd kan också härledas från en rad andra datatyper, inklusive morfologi, närvaron eller frånvaron av särskilda typer av gener, insättnings- och raderingshändelser – och alla andra observationer som tros innehålla en evolutionär signal.
Fylogenetiska nätverk används när förgrenade träd inte är lämpliga, på grund av dessa komplikationer som tyder på en mer retikulerad evolutionär historia av de provtagna organismerna.
Se även
- Clade
- Kladistik
- Beräkningsfylogenetik
- Evolutionsbiologi
- Evolutionär taxonomi
- Generaliserad trädanpassning
- Lista över fylogenetik programvara
- Lista över programvara för fylogenetisk trädvisualisering
- PANDIT , en biologisk databas som täcker proteindomäner
- Fylogenetiska jämförande metoder
- Fylogenetisk försoning
- Taxonomisk rangordning
Vidare läsning
- Schuh, RT och AVZ Brower. 2009. Biologisk systematik: principer och tillämpningar (2nd edn.) ISBN 978-0-8014-4799-0
- Manuel Lima , The Book of Trees: Visualizing Branches of Knowledge , 2014, Princeton Architectural Press, New York.
- MEGA , en gratis programvara för att rita fylogenetiska träd.
- Gontier, N. 2011. "Depicing the Tree of Life: the Philosophical and Historical Roots of Evolutionary Tree Diagrams." Evolution, Education, Outreach 4: 515–538.
externa länkar
Bilder
- Mänsklig Y-kromosom 2002 fylogenetiskt träd
- iTOL: Interactive Tree Of Life
- Det fylogenetiska trädet av artificiella organismer utvecklats på datorer arkiverat 2016-02-22 på Wayback Machine
- Miyamoto och Goodmans fylogram över eutheriska däggdjur
Allmän
- En översikt över olika metoder för trädvisualisering finns på Page, RDM (2011). "Rymd, tid, form: Att se livets träd". Trender inom ekologi och evolution . 27 (2): 113–120. doi : 10.1016/j.tree.2011.12.002 . PMID 22209094 .
- OneZoom: Livets träd – alla levande arter som intuitiv och zoombar fraktalutforskare (responsiv design)
- Discover Life Ett interaktivt träd baserat på US National Science Foundations Assembling the Tree of Life Project
- PhyloCode
- En multipel justering av 139 myosinsekvenser och ett fylogenetiskt träd
- Livets träd webbprojekt
- Fylogenetiska slutsatser om T-REX-servern
- NCBI:s taxonomidatabas [1]
- ETE: A Python Environment for Tree Exploration Detta är ett programmeringsbibliotek för att analysera, manipulera och visualisera fylogenetiska träd. Ref.
- Ett dagligt uppdaterat träd av (sekvenserat) liv Fang, H.; Oates, ME; Pethica, RB; Greenwood, JM; Sardar, AJ; Rackham, OJL; Donoghue, PCJ; Stamatakis, A.; De Lima Morais, DA; Gough, J. (2013). "Ett dagligt uppdaterat träd av (sekvenserat) liv som referens för genomforskning" . Vetenskapliga rapporter . 3 : 2015. Bibcode : 2013NatSR...3E2015F . doi : 10.1038/srep02015 . PMC 6504836 . PMID 23778980 .









