PubMed
 | |
| Kontakt | |
|---|---|
| Forskningscenter | United States National Library of Medicine (NLM) |
| Utgivningsdatum | januari 1996 |
| Tillgång | |
| Hemsida | |
PubMed är en gratis sökmotor som i första hand har tillgång till MEDLINE- databasen med referenser och sammanfattningar om biovetenskap och biomedicinska ämnen. United States National Library of Medicine (NLM) vid National Institutes of Health underhåller databasen som en del av Entrez -systemet för informationshämtning .
Från 1971 till 1997 hade tillgången online till MEDLINE-databasen främst skett genom institutionella faciliteter, såsom universitetsbibliotek . PubMed, som först släpptes i januari 1996, inledde en tid präglad av privat, gratis, hem- och kontorsbaserad MEDLINE-sökning. PubMed-systemet erbjöds gratis för allmänheten från och med juni 1997.
Innehåll
Förutom MEDLINE ger PubMed tillgång till:
- äldre referenser från den tryckta versionen av Index Medicus , tillbaka till 1951 och tidigare
- referenser till vissa tidskrifter innan de indexerades i Index Medicus och MEDLINE, till exempel Science , BMJ och Annals of Surgery
- mycket färska poster i poster för en artikel innan den indexeras med Medical Subject Headings (MeSH) och läggs till MEDLINE
- en samling böcker tillgängliga i fulltext och andra delmängder av NLM-poster
- PMC- citat
- NCBI bokhylla
Många PubMed-poster innehåller länkar till fulltextartiklar, av vilka några är fritt tillgängliga, ofta i PubMed Central och lokala speglar, såsom Europe PubMed Central .
Information om tidskrifterna indexerade i MEDLINE, och tillgängliga via PubMed, finns i NLM-katalogen.
Den 27 januari 2020 har PubMed mer än 30 miljoner citeringar och sammanfattningar som går tillbaka till 1966, selektivt till år 1865 och mycket selektivt till 1809. Från och med samma datum är 20 miljoner av PubMeds poster listade med sina sammanfattningar, och 21,5 miljoner poster har länkar till fulltextversioner (varav 7,5 miljoner artiklar är tillgängliga, fulltext gratis). Under de senaste 10 åren (som slutar 31 december 2019) har i genomsnitt nästan 1 miljon nya rekord lagts till varje år. Ungefär 12 % av journalerna i PubMed motsvarar cancerrelaterade poster, som har vuxit från 6 % på 1950-talet till 16 % 2016. Annan betydande andel av journalerna motsvarar "kemi" (8,69 %), "terapi" (8,39 %) och "infektion" (5 %). [ citat behövs ]
Under 2016 ändrade NLM indexeringssystemet så att publicister direkt kan korrigera stavfel och fel i PubMed-indexerade artiklar.
PubMed har rapporterats inkludera några artiklar publicerade i rovtidskrifter. MEDLINE och PubMeds policyer för urval av tidskrifter för databasinkludering skiljer sig något. Svagheter i kriterierna och procedurerna för indexering av tidskrifter i PubMed Central kan göra att publikationer från rovtidskrifter läcker in i PubMed. National Library of Medicine hade svarat att enskilda tidskriftsartiklar kan inkluderas i PMC för att stödja forskningsfinansiärers policy för allmänhetens tillgång och att rigorösa policyer om tidskrifter och förläggare säkerställer integriteten hos NLMs litteraturdatabaser.
Egenskaper
Hemsidedesign
Ett nytt PubMed-gränssnitt lanserades i oktober 2009 och uppmuntrade användningen av sådana snabba, Google-liknande sökformuleringar; de har också beskrivits som "telegramsökningar". Som standard sorteras resultaten efter Senaste, men detta kan ändras till Bästa matchning, Publiceringsdatum, Första författare, Senaste författare, Journal eller Titel.
PubMeds webbdesign och domän uppdaterades i januari 2020 och blev standard den 15 maj 2020, med de uppdaterade och nya funktionerna. Det kom en kritisk reaktion från många forskare som ofta använder sajten.
PubMed för handdatorer/mobiler
PubMed/MEDLINE kan nås via handhållna enheter, med till exempel alternativet "PICO" (för fokuserade kliniska frågor) skapat av NLM. Ett "PubMed Mobile"-alternativ, som ger tillgång till en mobilvänlig, förenklad PubMed-version, finns också.
Sök
Standardsökning
Enkla sökningar på PubMed kan utföras genom att skriva in nyckelaspekter av ett ämne i PubMeds sökfönster.
PubMed översätter denna första sökformulering och lägger automatiskt till fältnamn, relevanta MeSH-termer (Medical Subject Headings), synonymer, booleska operatorer och "kapslar" de resulterande termerna på lämpligt sätt, vilket förbättrar sökformuleringen avsevärt, särskilt genom att rutinmässigt kombinera (med hjälp av OR) operator) textord och MeSH-termer. [ citat behövs ]
Exemplen som ges i en PubMed-handledning visar hur denna automatiska process fungerar:
Orsaker Sömnvandring översätts som ("etiologi"[Underrubrik] ELLER "etiologi"[Alla fält] ELLER "orsaker"[Alla fält] ELLER "kausalitet"[MeSH Termer] ELLER "kausalitet"[Alla fält]) OCH ( " somnambulism "[MeSH-villkor] ELLER "somnambulism"[All Fields] ELLER ("sleep"[All Fields] AND "walking"[All Fields]) ELLER "sömngång"[All Fields])
Likaså,
soft Attack Aspirin Prevention översätts som ("hjärtinfarkt"[MeSH-termer] ELLER ("hjärtinfarkt"[Alla fält] OCH "infarkt"[Alla fält]) ELLER "hjärtinfarkt"[Alla fält] ELLER ("hjärta"[Alla Fields] AND "attack"[All Fields]) OR "heart attack"[All Fields]) AND ("aspirin"[MeSH Terms] ELLER "aspirin"[All Fields]) AND ("förebyggande och kontroll"[Underrubrik] ELLER ("förebyggande"[Alla fält] OCH "kontroll"[Alla fält]) ELLER "förebyggande och kontroll"[Alla fält] ELLER "förebyggande"[Alla fält])
Omfattande sökning
För optimala sökningar i PubMed är det nödvändigt att förstå dess kärnkomponent, MEDLINE, och särskilt av den MeSH (Medical Subject Headings) kontrollerade vokabulären som används för att indexera MEDLINE-artiklar. De kan också kräva komplexa sökstrategier, användning av fältnamn (taggar), korrekt användning av gränser och andra funktioner; referensbibliotekarier och sökspecialister erbjuder söktjänster.
Sökningen i PubMeds sökfönster rekommenderas endast för sökning av entydiga ämnen eller nya insatser som ännu inte har skapat en MeSH-rubrik, samt för sökning efter kommersiella märken av läkemedel och egennamn. Det är också användbart när det inte finns någon lämplig rubrik eller om deskriptorn representerar en delaspekt. Sökningen med hjälp av tesaurus MeSH är mer exakt och ger färre irrelevanta resultat. Dessutom sparar det nackdelen med fritextsökningen där skillnaderna i stavning, singular/plural eller förkortningar måste beaktas. Å andra sidan kommer inte artiklar som nyligen införlivats i databasen som ännu inte har tilldelats deskriptorer att hittas. För att garantera en uttömmande sökning måste därför en kombination av kontrollerade språkrubriker och fritexttermer användas.
Tidskriftsartikelparametrar
När en tidskriftsartikel indexeras extraheras många artikelparametrar och lagras som strukturerad information. Sådana parametrar är: Artikeltyp (MeSH-termer, t.ex. "Clinical Trial"), Sekundära identifierare, (MeSH-termer), Språk, Land för tidskriften eller publiceringshistorik (e-publiceringsdatum, tryckt tidskrifts publiceringsdatum).
Publikationstyp: Kliniska frågor/systematiska översikter
Parametern Publikationstyp tillåter sökning efter typ av publikation , inklusive rapporter om olika typer av klinisk forskning.
Sekundärt ID
Sedan juli 2005 extraherar MEDLINE-artikelindexeringsprocessen identifierare från artikelsammandraget och placerar dem i ett fält som kallas Secondary Identifier (SI). Det sekundära identifieringsfältet är att lagra accessionsnummer till olika databaser med molekylära sekvensdata, genuttryck eller kemiska föreningar och kliniska prövnings-ID. För kliniska prövningar extraherar PubMed prövnings-ID:n för de två största prövningsregistren: ClinicalTrials.gov (NCT-identifierare) och International Standard Randomized Controlled Trial Number Register (IRCTN-identifierare).
Se även
En referens som bedöms som särskilt relevant kan markeras och "relaterade artiklar" kan identifieras. Om det är relevant kan flera studier väljas ut och relaterade artiklar till dem alla kan genereras (på PubMed eller någon av de andra NCBI Entrez-databaserna) med alternativet 'Hitta relaterad data'. De relaterade artiklarna listas sedan i ordning efter "släktskap". För att skapa dessa listor med relaterade artiklar, jämför PubMed ord från titeln och sammanfattningen av varje citat, såväl som MeSH-rubrikerna som tilldelats, med hjälp av en kraftfull ordviktad algoritm. Funktionen "relaterade artiklar" har bedömts vara så exakt att författarna till en artikel föreslog att den kan användas istället för en fullständig sökning.
Mappning till MeSH
PubMed länkar automatiskt till MeSH-termer och underrubriker. Exempel skulle vara: "dålig andedräkt" länkar till (och inkluderar i sökningen) "halitos", "hjärtinfarkt" till "hjärtinfarkt", "bröstcancer" till "bröstneoplasmer". När så är lämpligt "expanderas dessa MeSH-termer automatiskt", det vill säga inkluderar mer specifika termer. Termer som "omvårdnad" länkas automatiskt till "Omvårdnad [MeSH]" eller "Omvårdnad [Underrubrik]". Den här funktionen kallas Auto Term Mapping och används som standard i fritextsökning men inte exakt frassökning (dvs. omger sökfrågan med dubbla citattecken). Denna funktion gör PubMed-sökningar mer känsliga och undviker falsknegativa (missade) träffar genom att kompensera för mångfalden av medicinsk terminologi.
PubMed tillämpar inte automatisk mappning av termen under följande omständigheter: genom att skriva den citerade frasen (t.ex. "njurallotransplantat"), när den stympas på asterisken (t.ex. njurallograft*), och när man tittar med fältetiketter (t.ex. Cancer [ti]).
Min NCBI
PubMeds valfria anläggning "My NCBI" (med gratis registrering) tillhandahåller verktyg för
- spara sökningar
- filtrering av sökresultat
- ställa in automatiska uppdateringar som skickas via e-post
- spara uppsättningar av referenser som hämtats som en del av en PubMed-sökning
- konfigurera visningsformat eller markera söktermer
och ett brett utbud av andra alternativ. Området "My NCBI" kan nås från vilken dator som helst med webbåtkomst. En tidigare version av "My NCBI" hette "PubMed Cubby".
Länka ut
LinkOut är en NLM-möjlighet för att länka och göra tillgängliga lokala tidskrifter i fulltext. Cirka 3 200 platser (främst akademiska institutioner) deltar i denna NLM-anläggning (i mars 2010), från Aalborg Universitet i Danmark till ZymoGenetics i Seattle. Användare vid dessa institutioner ser sin institutions logotyp i PubMed-sökresultatet (om tidskriften hålls på den institutionen) och kan få tillgång till hela texten. Link out konsolideras med Outside Tool från och med den stora plattformsuppdateringen som kommer sommaren 2019.
PubMed Commons
Under 2016 tillåter PubMed författare av artiklar att kommentera artiklar indexerade av PubMed. Denna funktion testades initialt i ett pilotläge (sedan 2013) och gjordes permanent 2016. I februari 2018 avbröts PubMed Commons på grund av att "användningen har förblivit minimal".
fråga MEDLINE
askMEDLINE, ett frågeverktyg för fritext, naturligt språk för MEDLINE/PubMed, utvecklat av NLM, även lämpligt för handdatorer.
PubMed identifierare
En PMID (PubMed identifierare eller PubMed unik identifierare) är ett unikt heltalsvärde, som börjar på 1 , som tilldelas varje PubMed-post. Ett PMID är inte detsamma som ett PMCID (PubMed Central-identifierare) som är identifieraren för alla verk som publiceras i den kostnadsfria PubMed Central .
Tilldelningen av ett PMID eller PMCID till en publikation säger ingenting om innehållets typ eller kvalitet. PMID:er tilldelas brev till redaktören , redaktionella åsikter, opedkolumner och alla andra artiklar som redaktören väljer att inkludera i tidskriften, såväl som peer-reviewed tidningar. Förekomsten av identifikationsnumret är inte heller ett bevis på att papperen inte har dragits tillbaka för bedrägeri, inkompetens eller tjänstefel. Meddelandet om eventuella korrigeringar av originalpapper kan tilldelas ett PMID.
Varje nummer som skrivs in i PubMed-sökfönstret behandlas som standard som om det vore ett PMID. Därför kan alla referenser i PubMed lokaliseras med hjälp av PMID.
Alternativa gränssnitt
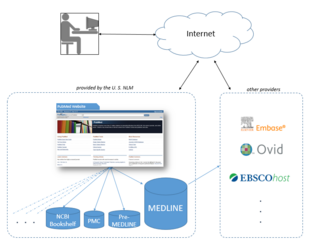
National Library of Medicine hyr ut MEDLINE-informationen till ett antal privata leverantörer som Embase , Ovid , Dialog , EBSCO , Knowledge Finder och många andra kommersiella, icke-kommersiella och akademiska leverantörer. I oktober 2008 hade mer än 500 licenser utfärdats, mer än 200 av dem till leverantörer utanför USA. Eftersom licenser för att använda MEDLINE-data är tillgängliga gratis, ger NLM i själva verket en gratis testplats för ett brett utbud av alternativa gränssnitt och tillägg från tredje part till PubMed, en av mycket få stora, professionellt kurerade databaser som erbjuder detta alternativ.
Lu identifierar ett urval av 28 aktuella och gratis webbaserade PubMed-versioner, som inte kräver någon installation eller registrering, vilka är grupperade i fyra kategorier:
- Rankning av sökresultat, till exempel: eTBLAST ; MedlineRanker; MiSearch;
- Gruppering av resultat efter ämnen, författare, tidskrifter etc., till exempel: Anne O'Tate ; ClusterMed;
- Förbättra semantik och visualisering, till exempel: EBIMed; MedEvi.
- Förbättrat sökgränssnitt och hämtningsupplevelse, till exempel askMEDLINE BabelMeSH; och PubCrawler.
Eftersom de flesta av dessa och andra alternativ huvudsakligen förlitar sig på PubMed/MEDLINE-data som hyrs under licens från NLM/PubMed, har termen "PubMed-derivat" föreslagits. Utan att behöva lagra cirka 90 GB ursprungliga PubMed-dataset, kan vem som helst skriva PubMed-applikationer med hjälp av eutils-applikationsprogramgränssnittet som beskrivs i "The E-utilities In-Depth: Parameters, Syntax and More", av Eric Sayers, PhD. Olika citeringsformatgeneratorer, som tar PMID-nummer som indata, är exempel på webbapplikationer som använder eutils-applikationens programgränssnitt. Exempel på webbsidor inkluderar Citation Generator - Mick Schroeder , Pubmed Citation Generator - Ultrasound of the Week , PMID2cite och Cite this for me .
Data mining av PubMed
i PubMed använder programmeringsmiljöer som Matlab , Python eller R. I dessa fall skrivs frågor av PubMed som kodrader och skickas till PubMed och svaret bearbetas sedan direkt i programmeringsmiljön. Kod kan automatiseras för att systematiskt söka med olika nyckelord som sjukdom, år, organ etc. En färsk publikation (2017) fann att andelen cancerrelaterade poster i PubMed har ökat från 6 % på 1950-talet till 16 % 2016 .
Data som är tillgänglig för PubMed kan speglas lokalt med hjälp av ett inofficiellt verktyg som MEDOC.
Miljontals PubMed-poster förstärker olika öppna datamängder om öppen åtkomst , som Unpaywall . Dataanalysverktyg som Unpaywall Journals används av bibliotek för att hjälpa till med avbokningar av stora affärer : bibliotek kan undvika prenumerationer på material som redan betjänas av omedelbar öppen åtkomst via öppna arkiv som PubMed Central.
Se även
- Europe PubMed Central
- JournalReview.org
- Lista över akademiska databaser och sökmotorer
- PubMed Central
- PubMed centrala Kanada