Ribonukleas H
| ribonukleas H | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Kristallografisk struktur av E. coli RNas HI.
| |||||||||
| Identifierare | |||||||||
| EG nr. | 3.1.26.4 | ||||||||
| CAS-nr. | 9050-76-4 | ||||||||
| Databaser | |||||||||
| IntEnz | IntEnz-vy | ||||||||
| BRENDA | BRENDA inträde | ||||||||
| ExPASy | NiceZyme-vy | ||||||||
| KEGG | KEGG inträde | ||||||||
| MetaCyc | Metabolisk väg | ||||||||
| PRIAM | profil | ||||||||
| PDB- strukturer | RCSB PDB PDBe PDB summa | ||||||||
| Genontologi | AmiGO / QuickGO | ||||||||
| |||||||||
| retroviralt ribonukleas H- | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| identifierare | |||||||||
| EG nr. | 3.1.26.13 | ||||||||
| Databaser | |||||||||
| IntEnz | IntEnz-vy | ||||||||
| BRENDA | BRENDA inträde | ||||||||
| ExPASy | NiceZyme-vy | ||||||||
| KEGG | KEGG inträde | ||||||||
| MetaCyc | Metabolisk väg | ||||||||
| PRIAM | profil | ||||||||
| PDB- strukturer | RCSB PDB PDBe PDB summa | ||||||||
| |||||||||
Ribonukleas H (förkortat RNase H eller RNH ) är en familj av icke - sekvensspecifika endonukleasenzymer som katalyserar klyvningen av RNA i ett RNA/ DNA - substrat via en hydrolytisk mekanism . Medlemmar av RNase H-familjen kan hittas i nästan alla organismer, från bakterier till archaea till eukaryoter .
Familjen är uppdelad i evolutionärt besläktade grupper med något olika substratpreferenser , brett betecknade ribonukleas H1 och H2. Det mänskliga genomet kodar för både H1 och H2. Humant ribonukleas H2 är ett heterotrimert komplex som består av tre subenheter, mutationer i var och en av dem är bland de genetiska orsakerna till en sällsynt sjukdom som kallas Aicardi-Goutières syndrom . En tredje typ, nära besläktad med H2, finns endast i ett fåtal prokaryoter , medan H1 och H2 förekommer i alla livets områden . Dessutom förekommer RNas H1-liknande retrovirala ribonukleas H- domäner i multidomän omvänt transkriptasproteiner , som kodas av retrovirus såsom HIV och krävs för viral replikation.
I eukaryoter är ribonukleas H1 involverad i DNA-replikation av mitokondriella genom . Både H1 och H2 är involverade i genomunderhållsuppgifter såsom bearbetning av R-loopstrukturer .
Klassificering och nomenklatur
Ribonukleas H är en familj av endonukleasenzymer med en delad substratspecificitet för RNA-strängen av RNA - DNA - duplex . Per definition klyver RNaser H RNA-ryggradsfosfodiesterbindningar för att lämna en 3'- hydroxyl- och en 5'- fosfatgrupp . RNaser H har föreslagits som medlemmar av en evolutionärt besläktad superfamilj som omfattar andra nukleaser och nukleinsyrabearbetningsenzymer såsom retrovirala integraser , DNA- transposaser , Holliday junction-resolvaser , Piwi- och Argonaute - proteiner, olika exonukleaser och splitsningsproteinet Prp8 .
RNaser H kan grovt delas in i två undertyper, H1 och H2, som av historiska skäl ges arabiska sifferbeteckningar i eukaryoter och romerska sifferbeteckningar i prokaryoter . Således Escherichia coli RNase HI en homolog av Homo sapiens RNase H1. I E. coli och många andra prokaryoter kodar rnhA -genen för HI och rnhB -genen kodar för HII. En tredje besläktad klass, kallad HIII, förekommer i ett fåtal bakterier och archaea ; det är nära besläktat med prokaryota HII-enzymer.
Strukturera
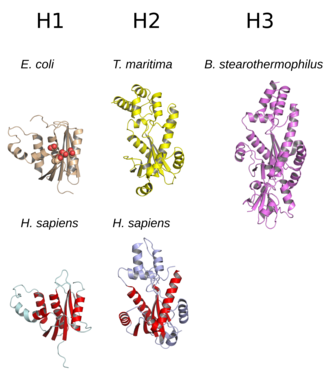
Strukturen av RNase H består vanligtvis av ett 5-strängat β-ark omgivet av en fördelning av α - helixar . Alla RNaser H har ett aktivt ställe centrerat på ett konserverat sekvensmotiv som består av aspartat- och glutamatrester , ofta hänvisat till som DEDD-motivet. Dessa rester interagerar med katalytiskt erforderliga magnesiumjoner .
RNaser H2 är större än H1 och har vanligtvis ytterligare helixar. Enzymernas domänorganisation varierar ; vissa prokaryota och mest eukaryota medlemmar av H1-gruppen har ytterligare en liten domän vid N-terminalen känd som "hybridbindningsdomänen", vilket underlättar bindning till RNA:DNA-hybridduplex och ibland ger ökad processivitet . Medan alla medlemmar av H1-gruppen och de prokaryota medlemmarna av H2-gruppen fungerar som monomerer, är eukaryota H2-enzymer obligatoriska heterotrimerer . Prokaryota HIII-enzymer är medlemmar av den bredare H2-gruppen och delar de flesta strukturella egenskaper med H2, med tillägget av en N-terminal TATA-boxbindande domän . Retrovirala RNas H-domäner som förekommer i multidomän omvänt transkriptasproteiner har strukturer som nära liknar H1-gruppen.
RNaser H1 har studerats omfattande för att utforska sambanden mellan struktur och enzymatisk aktivitet. De används också, särskilt E. coli- homologen, som modellsystem för att studera proteinveckning . Inom H1-gruppen har ett samband identifierats mellan högre substratbindande affinitet och närvaron av strukturella element bestående av en helix och flexibel slinga som ger en större och mer grundläggande substratbindande yta . C-helixen har en spridd taxonomisk fördelning; det finns i E. coli och humana RNase H1-homologer och saknas i HIV RNas H-domänen, men det finns exempel på retrovirala domäner med C-helixar.
Fungera
Ribonukleas H-enzymer klyver fosfodiesterbindningarna av RNA i en dubbelsträngad RNA:DNA-hybrid, vilket lämnar en 3'- hydroxyl- och en 5'- fosfatgrupp på vardera änden av det skurna stället med en två-metalljon-katalysmekanism, där två divalenta katjoner, såsom Mg2+ och Mn2+, deltar direkt i den katalytiska funktionen. Beroende på skillnaderna i deras aminosyrasekvenser, klassificeras dessa RNaser H i typ 1 och typ 2 RNaser H. Typ 1 RNaser H har prokaryota och eukaryota RNaser H1 och retrovirala RNaser H. Typ 2 RNaser H har prokaryota och eukaryota RNaser H2 och bakteriellt RNas H3. Dessa RNaser H existerar i en monomer form, förutom eukaryota RNaser H2, som existerar i en heterotrimer form. RNas H1 och H2 har distinkta substratpreferenser och distinkta men överlappande funktioner i cellen. I prokaryoter och lägre eukaryoter är ingetdera enzymet väsentligt , medan båda tros vara väsentliga i högre eukaryoter. Den kombinerade aktiviteten av både H1- och H2-enzymer är associerad med upprätthållande av genomets stabilitet på grund av enzymernas nedbrytning av RNA-komponenten i R-loopar .
Ribonukleas H1
| Identifierare | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Symbol | RNase H | ||||||||
| Pfam | PF00075 | ||||||||
| Pfam klan | CL0219 | ||||||||
| InterPro | IPR002156 | ||||||||
| PROSITE | PS50879 | ||||||||
| |||||||||
Ribonukleas H1-enzymer kräver minst fyra ribonukleotid -innehållande baspar i ett substrat och kan inte avlägsna en enda ribonukleotid från en sträng som annars är sammansatt av deoxiribonukleotider. Av denna anledning anses det osannolikt att RNase H1-enzymer är involverade i bearbetningen av RNA-primrar från Okazaki-fragment under DNA-replikation . RNas H1 är inte väsentligt i encelliga organismer där det har undersökts; i E. coli ger RNase H1 knockouts en temperaturkänslig fenotyp, och i S. cerevisiae producerar de defekter i stressrespons.
I många eukaryoter, inklusive däggdjur , inkluderar RNase H1-gener en mitokondriell målsökningssekvens , vilket leder till uttryck av isoformer med och utan MTS närvarande. Som ett resultat är RNase H1 lokaliserad till både mitokondrier och kärnan . I knockout-musmodeller är RNase H1-null-mutanter dödliga under embryogenes på grund av defekter i replikerande mitokondrie-DNA . Defekterna i mitokondriell DNA-replikation som induceras av förlust av RNas H1 beror sannolikt på defekter i R-loopbearbetning .
Ribonukleas H2
| Identifierare | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Symbol | RNase HII | ||||||||
| Pfam | PF01351 | ||||||||
| Pfam klan | CL0219 | ||||||||
| InterPro | IPR024567 | ||||||||
| |||||||||
I prokaryoter är RNase H2 enzymatiskt aktivt som ett monomert protein. I eukaryoter är det en obligat heterotrimer som består av en katalytisk subenhet A och strukturella subenheter B och C. Medan A-subenheten är nära homolog med det prokaryota RNas H2, har B- och C-subenheterna inga uppenbara homologer i prokaryoter och är dåligt konserverade vid sekvensnivån även bland eukaryoter . B-subenheten förmedlar protein-proteininteraktioner mellan H2-komplexet och PCNA , vilket lokaliserar H2 till replikationsfoci .
Både prokaryota och eukaryota H2-enzymer kan klyva enstaka ribonukleotider i en sträng. de har dock något annorlunda klyvningsmönster och substratpreferenser: prokaryota enzymer har lägre processivitet och hydrolyserar successiva ribonukleotider mer effektivt än ribonukleotider med en 5'- deoxiribonukleotid, medan eukaryota enzymer är mer processiva och hydrolyserar båda typerna av substrat med liknande effektivitet. Substratspecificiteten för RNase H2 ger den en roll i ribonukleotidexcisionsreparation , avlägsnande av felinkorporerade ribonukleotider från DNA, förutom R-loop- bearbetning. Även om både H1 och H2 är närvarande i däggdjurscellkärnan, är H2 den dominerande källan till RNas H-aktivitet där och är viktig för att upprätthålla genomets stabilitet.
Vissa prokaryoter har en ytterligare gen av H2-typ betecknad RNase HIII i den romerska siffrors nomenklatur som används för de prokaryota generna. HIII-proteiner är närmare besläktade med H2-gruppen genom sekvensidentitet och strukturell likhet, men har substratpreferenser som mer liknar H1. Till skillnad från HI och HII, som båda är vitt spridda bland prokaryoter, finns HIII i endast ett fåtal organismer med en spridd taxonomisk fördelning; det är något vanligare i archaea och finns sällan eller aldrig i samma prokaryota genom som HI.
Mekanism
Det aktiva stället för nästan alla RNaser H innehåller fyra negativt laddade aminosyrarester, kända som DEDD-motivet; ofta finns också ett histidin, t.ex. i HIV-1, människa eller E. coli.
De laddade resterna binder två metalljoner som krävs för katalys; under fysiologiska förhållanden är dessa magnesiumjoner , men mangan stöder vanligtvis också enzymatisk aktivitet, medan kalcium eller hög koncentration av Mg2+ hämmar aktiviteten.
Baserat på experimentella bevis och datorsimuleringar aktiverar enzymet en vattenmolekyl bunden till en av metalljonerna med det konserverade histidinet. Övergångstillståndet är associativt till sin natur och bildar en mellanprodukt med protonerat fosfat och deprotonerad alkoxid som lämnar grupp. Den lämnande gruppen protoneras via glutamatet som har ett förhöjt pKa och sannolikt kommer att protoneras. Mekanismen liknar RNase T och RuvC-subenheten i Cas9- enzymet som båda också använder en histidin- och en tvåmetalljonmekanism.
Mekanismen för frisättningen av den kluvna produkten är fortfarande olöst. Experimentella bevis från tidsupplöst kristallografi och liknande nukleaser pekar på en roll för en tredje jon i reaktionen som rekryteras till det aktiva stället.
I mänsklig biologi
Det mänskliga genomet innehåller fyra gener som kodar för RNas H:
- RNASEH1 , ett exempel på H1 (monomer) subtyp
- RNASEH2A , den katalytiska subenheten av det trimera H2-komplexet
- RNASEH2B , en strukturell subenhet av det trimera H2-komplexet
- RNASEH2C , en strukturell subenhet av det trimera H2-komplexet
Dessutom förekommer genetiskt material av retroviralt ursprung ofta i genomet, vilket återspeglar integreringen av genomen av humana endogena retrovirus . Sådana integrationshändelser resulterar i närvaron av gener som kodar för retroviralt omvänt transkriptas , vilket inkluderar en RNas H-domän. Ett exempel är ERVK6 . Long terminal repeat (LTR) och icke-lång terminal repeat (icke-LTR) retrotransposoner är också vanliga i genomet och inkluderar ofta sina egna RNas H-domäner, med en komplex evolutionär historia.
Roll i sjukdom

I små studier har mutationer i humant RNase H1 associerats med kronisk progressiv extern oftalmoplegi, ett vanligt kännetecken för mitokondriell sjukdom .
Mutationer i någon av de tre RNase H2-subenheterna är väletablerade som orsaker till en sällsynt genetisk störning som kallas Aicardi-Goutières syndrom (AGS), som visar sig som neurologiska och dermatologiska symtom i tidig ålder. Symtomen på AGS påminner mycket om medfödd virusinfektion och är förknippade med olämplig uppreglering av typ I-interferon . AGS kan också orsakas av mutationer i andra gener: TREX1 , SAMHD1 , ADAR och MDA5 /IFIH1, som alla är involverade i nukleinsyrabearbetning. Karakterisering av mutationsfördelning i en AGS-patientpopulation fann 5 % av alla AGS-mutationer i RNASEH2A, 36 % i 2B och 12 % i 2C. Mutationer i 2B har associerats med något mildare neurologisk funktionsnedsättning och med en frånvaro av interferoninducerad genuppreglering som kan detekteras hos patienter med andra AGS-associerade genotyper.
I virus
Två grupper av virus använder omvänd transkription som en del av sina livscykler: retrovirus , som kodar för deras genom i enkelsträngat RNA och replikerar genom en dubbelsträngad DNA-mellanprodukt; och dsDNA-RT-virus , som replikerar sina dubbelsträngade DNA-genom genom en RNA-"pregenom"-mellanprodukt. Patogena exempel inkluderar humant immunbristvirus respektive hepatit B-virus . Båda kodar för stora multifunktionella omvänt transkriptas (RT)-proteiner som innehåller RNas H-domäner.
Retrovirala RT-proteiner från HIV-1 och murint leukemivirus är de bäst studerade medlemmarna i familjen. Retroviral RT är ansvarig för att omvandla virusets enkelsträngade RNA-genom till dubbelsträngat DNA. Denna process kräver tre steg: för det första producerar RNA-beroende DNA-polymerasaktivitet minussträngs- DNA från plussträng-RNA-mallen, vilket genererar en RNA:DNA-hybridmellanprodukt; för det andra förstörs RNA-strängen; och för det tredje DNA-beroende DNA-polymerasaktivitet plussträngs-DNA, vilket genererar dubbelsträngat DNA som slutprodukten. Det andra steget i denna process utförs av en RNas H-domän belägen vid C-terminalen av RT-proteinet.
RNase H utför tre typer av klyvningsåtgärder: ospecifik nedbrytning av plussträngs-RNA-genomet, specifikt avlägsnande av minussträngs- tRNA- primern och avlägsnande av plussträngs-purinrika polypurintrakt (PPT)-primer. RNas H spelar en roll i primingen av plussträngen, men inte i den konventionella metoden för att syntetisera en ny primersekvens. Snarare skapar RNas H en "primer" från PPT som är resistent mot RNas H-klyvning. Genom att ta bort alla baser utom PPT, används PPT som en markör för slutet av U3-regionen av dess långa terminala upprepning .
Eftersom RNas H-aktivitet krävs för viral proliferation, har denna domän ansetts vara ett läkemedelsmål för utveckling av antiretrovirala läkemedel som används vid behandling av HIV/AIDS och andra tillstånd orsakade av retrovirus. Inhibitorer av retroviralt RNas H av flera olika kemotyper har identifierats, av vilka många har en verkningsmekanism baserad på kelering av katjonerna på det aktiva stället. Omvända transkriptashämmare som specifikt hämmar polymerasfunktionen hos RT är i utbredd klinisk användning, men inte hämmare av RNas H-funktionen; det är den enda enzymatiska funktionen som kodas av hiv som ännu inte är föremål för läkemedel i klinisk användning.
Evolution
RNaser H är vitt spridda och förekommer i alla livets domäner . Familjen tillhör en större superfamilj av nukleasenzymer och anses vara evolutionärt gammal. I prokaryota genom är flera RNase H-gener ofta närvarande, men det finns liten korrelation mellan förekomsten av HI-, HII- och HIII-gener och övergripande fylogenetiska förhållanden , vilket tyder på att horisontell genöverföring kan ha spelat en roll för att fastställa fördelningen av dessa enzymer. RNas HI och HIII förekommer sällan eller aldrig i samma prokaryota genom. När en organisms genom innehåller mer än en RNas H-gen, har de ibland betydande skillnader i aktivitetsnivå. Dessa observationer har föreslagits spegla ett evolutionärt mönster som minimerar funktionell redundans bland RNase H-gener. RNas HIII, som är unikt för prokaryoter, har en spridd taxonomisk fördelning och finns i både bakterier och arkéer ; det tros ha avvikit från HII ganska tidigt.
Den evolutionära banan för RNas H2 i eukaryoter, särskilt mekanismen genom vilken eukaryota homologer blev obligata heterotrimerer, är oklar; B- och C-subenheterna har inga uppenbara homologer i prokaryoter.
Ansökningar
Eftersom RNase H specifikt bara bryter ned RNA:t i dubbelsträngade RNA:DNA-hybrider, används det vanligtvis som ett laboratoriereagens inom molekylärbiologi . Renade preparat av E. coli RNas HI och HII är kommersiellt tillgängliga. RNase HI används ofta för att förstöra RNA-mallen efter första-strängs komplementärt DNA (cDNA) syntes genom omvänd transkription . Det kan också användas för att klyva specifika RNA-sekvenser i närvaro av korta komplementära segment av DNA. Mycket känsliga tekniker som ytplasmonresonans kan användas för detektion. RNas HII kan användas för att bryta ned RNA-primerkomponenten i ett Okazaki-fragment eller för att introducera enkelsträngade hack i positioner som innehåller en ribonukleotid. En variant av varmstart-PCR , känd som RNase H-beroende PCR eller rhPCR, har beskrivits med användning av ett termostabilt RNase HII från den hypertermofila arkeonen Pyrococcus abyssi . Det bör noteras att det ribonukleasinhibitorprotein som vanligtvis används som reagens inte är effektivt för att hämma aktiviteten av varken HI eller HII.
Historia
Peter Hausens laboratorium när forskare fann RNA:DNA-hybrid- endonukleasaktivitet i kalvtymus 1969 och gav den namnet "ribonukleas H " för att beteckna dess hybridspecificitet . RNas H-aktivitet upptäcktes därefter i E. coli och i ett prov av onkovirus med RNA-genom under tidiga studier av viral omvänd transkription . Det blev senare klart att kalvtymusextrakt innehöll mer än ett protein med RNas H-aktivitet och att E. coli innehöll två RNas H-gener. Ursprungligen betecknades enzymet som nu är känt som RNase H2 i eukaryoter H1 och vice versa, men namnen på de eukaryota enzymerna byttes ut för att matcha dem i E. coli för att underlätta jämförande analys, vilket ger den moderna nomenklaturen där de prokaryota enzymerna betecknas med romerska siffror och de eukaryota enzymerna med arabiska siffror. Den prokaryota RNas HIII, som rapporterades 1999, var den sista RNas H-subtypen som identifierades.
Att karakterisera eukaryot RNase H2 var historiskt sett en utmaning, delvis på grund av dess låga förekomst. Noggranna ansträngningar vid rening av enzymet antydde att, till skillnad från E. coli RNase H2, det eukaryota enzymet hade flera subenheter. S. cerevisiae -homologen av E. coli- proteinet (det vill säga H2A-subenheten) var lätt identifierbar med bioinformatik när jästgenomet sekvenserades , men motsvarande protein visade sig inte ha enzymatisk aktivitet isolerat. Så småningom isolerades jäst B- och C-subenheterna genom samrening och visade sig vara nödvändiga för enzymatisk aktivitet. Emellertid har jäst B- och C-subenheterna mycket låg sekvensidentitet med sina homologer i andra organismer, och motsvarande humana proteiner identifierades slutgiltigt först efter att mutationer i alla tre visade sig orsaka Aicardi-Goutières syndrom .
externa länkar
- GeneReviews/NCBI/NIH/UW-inlägg om Aicardi-Goutières syndrom
- RNase+H vid US National Library of Medicine Medical Subject Headings (MeSH)

