DnaG
| DNA- | |||||||
|---|---|---|---|---|---|---|---|
| primasidentifierare | |||||||
| Organism | |||||||
| Symbol | dnaG | ||||||
| Alt. symboler | dnaP | ||||||
| Entrez | 947570 | ||||||
| PDB | 1D0Q, 1DD9, 1DDE, 1EQ9, 2R6A, 2R6C | ||||||
| RefSeq (Prot) | NP_417538 | ||||||
| UniProt | P0ABS5 | ||||||
| Övriga uppgifter | |||||||
| EG-nummer | 2.7.7.7 | ||||||
| Kromosom | kromosom: 3,21 - 3,21 Mb | ||||||
| |||||||
DnaG är en bakteriell DNA- primas och kodas av dnaG -genen. Enzymet DnaG, och alla andra DNA-primaser, syntetiserar korta RNA -strängar som kallas oligonukleotider under DNA-replikation . Dessa oligonukleotider är kända som primrar eftersom de fungerar som utgångspunkt för DNA-syntes. DnaG katalyserar syntesen av oligonukleotider som är 10 till 60 nukleotider (den grundläggande enheten av DNA och RNA) långa, men de flesta av de syntetiserade oligonukleotiderna är 11 nukleotider. Dessa RNA-oligonukleotider tjänar som primrar, eller utgångspunkter, för DNA-syntes genom bakteriellt DNA-polymeras III (Pol III). DnaG är viktigt i bakteriell DNA-replikation eftersom DNA-polymeras inte kan initiera syntesen av en DNA-sträng, utan kan bara lägga till nukleotider till en redan existerande sträng. DnaG syntetiserar en enda RNA-primer vid replikationsursprunget . Denna primer tjänar till att primera ledande DNA-syntes. För den andra föräldrasträngen, den eftersläpande strängen , syntetiserar DnaG en RNA-primer med några få kilobaser (kb). Dessa primers tjänar som substrat för syntesen av Okazaki-fragment .
I E. coli associerar DnaG genom icke-kovalenta interaktioner med bakteriellt replikativt helikas DnaB för att utföra sin primasaktivitet, med tre DnaG-primasproteiner som associerar med varje DnaB-helikas för att bilda primosomen . Primaser tenderar att initiera syntes vid specifika tre nukleotidsekvenser på enkelsträngade DNA (ssDNA) mallar och för E. coli DnaG är sekvensen 5'-CTG-3'.
DnaG innehåller tre separata proteindomäner : en zinkbindande domän, en RNA-polymerasdomän och en DnaB-helikasbindande domän. Det finns flera bakterier som använder DNA-primasen DnaG. Några få organismer som har DnaG som DNA-primas är Escherichia coli ( E. coli ), Bacillus stearothermophilus och Mycobacterium tuberculosis (MTB). E. coli DnaG har en molekylvikt på 60 kilodalton (kDa) och innehåller 581 aminosyror .
Fungera
DnaG katalyserar syntesen av oligonukleotider i fem diskreta steg: mallbindning, nukleosidtrifosfatbindning (NTP), initiering, förlängning för att bilda en primer och primeröverföring till DNA-polymeras III. DnaG utför denna katalys nära replikationsgaffeln som bildas av DnaB-helikas under DNA-replikation. DnaG måste komplexbindas med DnaB för att det ska katalysera bildningen av oligonukleotidprimrarna.
Mekanismen för primersyntes av primaser involverar två NTP-bindningsställen på primasproteinet (DnaG). Innan bindning av eventuella NTP:er för att bilda RNA-primern, binder ssDNA-mallsekvensen till DnaG. ssDNA innehåller en igenkänningssekvens med tre nukleotider som rekryterar NTP baserat på Watson-Crick-basparning . Efter bindning av DNA måste DnaG binda två NTP:er för att generera ett enzym-DNA-NTP-NTP-kvartärt komplex. Michaelis-konstanten (km) för NTP:erna varierar beroende på primas och mallar. De två NTP-bindningsställena på DnaG hänvisas till som initieringsstället och förlängningsstället. Initieringsstället är det ställe vid vilket NTP som ska inkorporeras vid 5'-änden av primern binder. Förlängningsstället binder NTP:n som läggs till 3'-änden av primern.
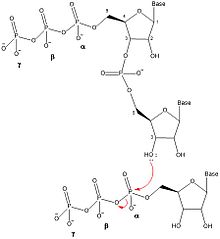
När två nukleotider väl är bundna till primasen katalyserar DnaG bildandet av en dinukleotid genom att bilda en fosfodiesterbindning via dehydreringssyntes mellan 3'-hydroxylen i nukleotiden i initieringsstället och nukleotidens a-fosfat i förlängningsstället. Denna reaktion resulterar i en dinukleotid och brytning av bindningen mellan α- och β-fosfor, vilket frisätter pyrofosfat. Denna reaktion är irreversibel eftersom pyrofosfatet som bildas hydrolyseras till två oorganiska fosfatmolekyler av enzymet oorganiskt pyrofosfatas . Denna dinukleotidsyntesreaktion är samma reaktion som vilket annat enzym som helst som katalyserar bildningen av DNA eller RNA ( DNA Polymerase , RNA Polymerase ), därför måste DnaG alltid syntetisera oligonukleotider i 5'- till 3'-riktningen. I E. coli börjar primrar med en trifosfatadenin-guanin (pppAG)-dinukleotid vid 5'-änden.
För att ytterligare förlängning av dinukleotiden ska ske måste oligonukleotiden flyttas så att 3'-NTP överförs från förlängningsstället till initieringsstället, vilket möjliggör för en annan NTP att binda till förlängningsstället och fästa till 3'-hydroxylen av oligonukleotiden. När en oligonukleotid av lämplig längd har syntetiserats från förlängningssteget av primersyntes, överför DnaG den nyligen syntetiserade primern till DNA-polymeras III för att den ska syntetisera den DNA-ledande strängen eller Okazaki-fragmenten för den eftersläpande strängen. Det hastighetsbegränsande steget för primersyntesen sker efter NTP-bindning men före eller under dinukleotidsyntes.
Strukturera
E. Coli DnaG-primas är ett monomert protein med 581 rester med tre funktionella domäner, enligt proteolysstudier. Det finns en N-terminal zinkbindande domän (resterna 1–110) där en zinkjon är tetraedriskt koordinerad mellan en histidin och tre cysteinrester, vilket spelar en roll för att känna igen sekvensspecifika DNA-bindningsställen. Den centrala domänen (resterna 111–433) visar RNA-polymerasaktiviteter och är platsen för RNA-primersyntes. Den C-terminala domänen (resterna 434–581) är ansvarig för den icke-kovalenta bindningen av DnaG till DnaB-helikasproteinet .
Zinkbindande domän

Den zinkbindande domänen, den domän som är ansvarig för att känna igen sekvensspecifika DNA-bindningsställen, är konserverad över alla virala, bakteriofag-, prokaryota och eukaryota DNA-primaser. Primas zinkbindande domän är en del av underfamiljen av zinkbindande domäner som kallas zinkbandet . Zinkbandsdomäner kännetecknas av två β-hårnålsöglor som bildar den zinkbindande domänen. Typiskt anses zinkbandsdomäner sakna a-helixar , vilket skiljer dem från andra zinkbindande domäner. År 2000 kristalliserades emellertid DnaG:s zinkbindande domän från Bacillus stearothermophilus, vilket avslöjade att domänen bestod av ett femsträngat antiparallellt β-ark intill fyra a-helixar och en 3 10 helix på den c-terminala änden av domänen.
Zinkbindningsstället för B. stearothermophilus består av tre cysteinrester, Cys40, Cys61 och Cys64, och en histidinrest, His43. Cys40 och His43 är belägna på β-hårnålen mellan det andra och tredje β-arket. Cys61 finns på det femte β-arket och Cys64 är på β-hårnålen mellan det fjärde och femte β-arket. Dessa fyra rester koordinerar zinkjonen tetraedriskt. Zinkjonen tros stabilisera slingorna mellan det andra och tredje β-arket samt det fjärde och femte β-arket. Domänen stabiliseras ytterligare av ett antal hydrofoba interaktioner mellan den hydrofoba inre ytan av p-arket som är packad mot de andra och tredje a-spiralerna. Den yttre ytan av p-arket har också många konserverade hydrofoba och basiska rester. Dessa rester är Lys30, Arg34, Lys46, Pro48, Lys56, Ile58, His60 och Phe62.
DNA-bindning
Man tror att funktionen hos den zinkbindande domänen är för sekvensspecifik DNA-igenkänning. DNA-primers gör RNA-primers som sedan används för DNA-syntes. Placeringen av RNA-primrarna är inte slumpmässig, vilket tyder på att de är placerade på specifika DNA-sekvenser. I själva verket har andra DNA-primaser visat sig känna igen triplettsekvenser; den specifika sekvens som känns igen av B. stearothermophilus har ännu inte identifierats. Det har visat sig att om cystinresterna som koordinerar zinkjonen muteras, slutar DNA-primasen att fungera. Detta indikerar att den zinkbindande domänen spelar en roll vid sekvensigenkänning. Dessutom tjänar den hydrofoba ytan på β-arket, såväl som de grundläggande resterna som är klustrade primärt på ena kanten av arket, till att attrahera enkelsträngat DNA, vilket ytterligare underlättar DNA-bindning.
Baserat på tidigare studier av DNA-bindning av DNA-primaser, tros det att DNA binder till den zinkbindande domänen över ytan av β-arket, med de tre nukleotiderna som binder över tre strängar av β-arket. De positivt laddade resterna i arket skulle kunna bilda kontakter med fosfaterna och de aromatiska resterna skulle bilda staplingsinteraktioner med baserna. Detta är modellen för DNA-bindning av den ssDNA-bindande domänen av replikationsprotein A (RPA) . Det är logiskt att anta att B. stearothermophilus zinkbindande domän binder DNA på ett liknande sätt, eftersom resterna som är viktiga för att binda DNA i RPA förekommer i strukturellt ekvivalenta positioner i B. stearothermophilus .
RNA-polymerasdomän

Som namnet antyder är RNA-polymerasdomänen (RNAP) av DnaG ansvarig för att syntetisera RNA-primrarna på det enkelsträngade DNA:t. In vivo kan DnaG syntetisera primerfragment på upp till 60 nukleotider, men in vivo primerfragment är begränsade till cirka 11 nukleotider. Under syntesen av den eftersläpande strängen syntetiserar DnaG mellan 2000 och 3000 primrar med en hastighet av en primer per sekund.
RNAP-domänen av DnaG har tre subdomäner, den N-terminala domänen, som har en blandad α- och β-veckning, den centrala domänen som består av ett 5-strängat β-ark och 6 α-helixar, och slutligen den C-terminala domänen som består av en spiralformad bunt bestående av 3 antiparallella α-helixar. Den centrala domänen utgörs av en del av toprimvecket , ett veck som har observerats i många metallbindande fosfoöverföringsproteiner. Den centrala domänen och den N-terminala domänen bildar en grund klyfta, som utgör det aktiva stället för RNA-kedjeförlängningen i DnaG. Öppningen av klyftan är kantad av flera mycket konserverade basiska rester: Arg146, Arg221 och Lys229. Dessa rester är en del av den elektrostatiskt positiva åsen av den N-terminala subdomänen. Det är denna ås som interagerar med ssDNA:t och hjälper till att styra det in i klyftan, som består av toprimmotivets metallbindande centrum på den centrala subdomänen och de konserverade primasmotiven i den N-terminala domänen. Metallbindningsstället för toprim-domänen är där primern syntetiseras. RNA:DNA-duplexet går sedan ut genom en annan grundläggande depression.
C-terminaldomän

Till skillnad från både de zinkbindande domänerna och RNA-polymerasdomänerna är de C-terminala domänerna av DNA-primaser inte konserverade. I prokaryota primaser är den enda kända funktionen för denna domän att interagera med helikaset, DnaB. Således kallas denna domän för helikasbindande domän (HBD). HBD för DnaG består av två underdomäner: en spiralbunt , C1-subdomänen och en spiralformad hårnål, C2-subdomänen. För var och en av de två till tre DnaG-molekylerna som binder DnaB-hexameren, interagerar C1-subdomänerna av HBD:erna med DnaB vid dess N-terminala domäner på den inre ytan av hexamerringen, medan C2-subdomänerna interagerar med de N-terminala domänerna på den yttre ytan av hexameren.

Tre rester i B. stearothermophilus DnaB har identifierats som viktiga för bildandet av DnaB, DnaG-gränssnittet. Dessa rester inkluderar Tyr88, Ile119 och Ile125. Tyr88 är nära, men kommer inte i kontakt med, HBD för DnaG. Mutation av Tyr88 hämmar bildandet av den N-terminala domänens spiralbunt av DnaB, vilket avbryter kontakterna med HBD av DnaG. Den hexameriska strukturen hos DnaB är verkligen en trimer av dimerer. Både Ile119 och Ile125 är begravda i det N-terminala domändimergränssnittet av DnaB och mutation av dessa rester hämmar bildningen av den hexameriska strukturen och därmed interaktionen med DnaG. En annan rest som har identifierats som spelar en avgörande roll i interaktionen mellan DnaB och DnaG är Glu15. Mutation av Glu15 stör inte bildandet av DnaB, DnaG-komplexet, utan spelar istället en roll för att modulera längden på primrar som syntetiseras av DnaG.
Hämning av DnaG
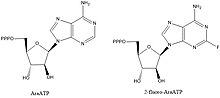
Hämmare av DNA-primaser är värdefulla föreningar för att belysa biokemiska vägar och nyckelinteraktioner, men de är också av intresse som ledande föreningar för att designa läkemedel mot bakteriella sjukdomar. De flesta av föreningarna som är kända för att hämma primaser är nukleotidanaloger såsom AraATP (se Vidarabin ) och 2-fluoro-AraATP. Dessa föreningar kommer ofta att användas som substrat av primasen, men när de väl inkorporerats kan syntes eller förlängning inte längre ske. Till exempel E. coli DnaG att använda 2',3'-dideoxinukleosid 5'-trifosfater (ddNTPs) som substrat, vilka fungerar som kedjeterminatorer på grund av avsaknaden av en 3' hydroxyl för att bilda en fosfodiesterbindning med nästa nukleotid.
Det relativt lilla antalet primashämmare återspeglar sannolikt den inneboende svårigheten med primasanalyser snarare än en brist på potentiella bindningsställen på enzymet. Den korta längden på syntetiserade produkter och den generellt långsamma hastigheten för enzymet jämfört med andra replikationsenzymer gör det svårare att utveckla metoder för screening med hög genomströmning ( HTS). Trots svårigheterna finns det flera kända hämmare av DnaG som inte är NTP-analoger. Doxorubicin och suramin är både DNA- och NTP-kompetitiva hämmare av Mycobacterium Tuberculosis DnaG. Suramin är också känt för att hämma eukaryot DNA-primas genom att konkurrera med GTP, så suramin kommer sannolikt att hämma DnaG via en liknande mekanism.
externa länkar
- dnaG+protein,+E+coli vid US National Library of Medicine Medical Subject Headings (MeSH)
- DnaG+ (Primase) vid US National Library of Medicine Medical Subject Headings (MeSH)