Isocitrat dehydrogenas
| Isocitratdehydrogenas | |||||||||
|---|---|---|---|---|---|---|---|---|---|
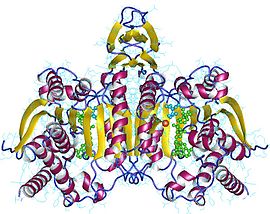
 Kristallografisk struktur av E. coli isocitratdehydrogenas. Det finns tre aktiva sajter. Tre isocitrat, ett isocitrat i bindningsstället för NADP + .
| |||||||||
| Identifierare | |||||||||
| EG nr. | 1.1.1.42 | ||||||||
| CAS-nr. | 9028-48-2 | ||||||||
| Databaser | |||||||||
| IntEnz | IntEnz-vy | ||||||||
| BRENDA | BRENDA inträde | ||||||||
| ExPASy | NiceZyme-vy | ||||||||
| KEGG | KEGG inträde | ||||||||
| MetaCyc | Metabolisk väg | ||||||||
| PRIAM | profil | ||||||||
| PDB- strukturer | RCSB PDB PDBe PDB summa | ||||||||
| Genontologi | AmiGO / QuickGO | ||||||||
| |||||||||
| isocitratdehydrogenas (NAD + ) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identifierare | |||||||||
| EG nr. | 1.1.1.41 | ||||||||
| CAS-nr. | 9001-58-5 | ||||||||
| Databaser | |||||||||
| IntEnz | IntEnz-vy | ||||||||
| BRENDA | BRENDA inträde | ||||||||
| ExPASy | NiceZyme-vy | ||||||||
| KEGG | KEGG inträde | ||||||||
| MetaCyc | Metabolisk väg | ||||||||
| PRIAM | profil | ||||||||
| PDB- strukturer | RCSB PDB PDBe PDB summa | ||||||||
| Genontologi | AmiGO / QuickGO | ||||||||
| |||||||||
| Monomer isocitrat dehydrogenas | |||||||||
|---|---|---|---|---|---|---|---|---|---|
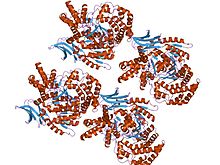 kristallstruktur av det monomera isocitrat dehydrogenas i komplex med isocitrat och mn
| |||||||||
| Identifierare | |||||||||
| Symbol | IDH | ||||||||
| Pfam | PF03971 | ||||||||
| Pfam klan | CL0270 | ||||||||
| InterPro | IPR004436 | ||||||||
| SCOP2 | 1ofg / SCOPe / SUPFAM | ||||||||
| |||||||||
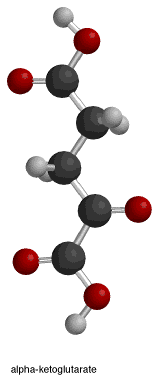
Isocitratdehydrogenas ( IDH ) ( EC 1.1.1.42 ) och ( EC 1.1.1.41 ) är ett enzym som katalyserar den oxidativa dekarboxyleringen av isocitrat och producerar alfa-ketoglutarat (α-ketoglutarat) och CO 2 . Detta är en tvåstegsprocess, som involverar oxidation av isocitrat (en sekundär alkohol ) till oxalosuccinat (en keton ), följt av dekarboxylering av karboxylgruppen beta till ketonen, vilket bildar alfa-ketoglutarat. Hos människor finns IDH i tre isoformer: IDH3 katalyserar det tredje steget i citronsyracykeln samtidigt som det omvandlar NAD + till NADH i mitokondrierna . Isoformerna IDH1 och IDH2 katalyserar samma reaktion utanför sammanhanget av citronsyracykeln och använder NADP + som en kofaktor istället för NAD + . De lokaliseras till cytosolen såväl som mitokondrien och peroxisomen .
Isozymer
Följande är en lista över humana isocitratdehydrogenasisozymer:
NADP + beroende
Varje NADP + -beroende isozym fungerar som en homodimer:
Se även
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
NAD + beroende
Isocitratdehydrogenas 3-isozymet är en heterotetramer som är sammansatt av två alfa-subenheter, en beta-subenhet och en gamma-subenhet:
|
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Strukturera
NAD-IDH består av 3 subenheter, är allosteriskt reglerad och kräver en integrerad Mg 2+ eller Mn 2+ jon. Den närmaste homologen som har en känd struktur är E. coli NADP-beroende IDH, som endast har 2 subenheter och en 13 % identitet och 29 % likhet baserat på aminosyrasekvenserna, vilket gör den olik human IDH och inte lämplig för nära jämförelse. Alla kända NADP-IDH är homodimerer.
De flesta isocitratdehydrogenaser är dimerer, för att vara specifika, homodimerer (två identiska monomersubenheter som bildar en dimer enhet). Vid jämförelse av C. glutamicum och E. coli , monomer respektive dimer, visade sig båda enzymerna "effektivt katalysera identiska reaktioner". C. glutamicum registrerades dock ha tio gånger så mycket aktivitet än E. coli och sju gånger mer affinitiv/specifik för NADP. C. glutamicum gynnade NADP + framför NAD + . När det gäller stabilitet med respons på temperatur, hade båda enzymerna en liknande Tm eller smälttemperatur vid cirka 55 °C till 60 °C. Monomeren C. glutamicum visade emellertid en mer konsekvent stabilitet vid högre temperaturer, vilket förväntades. Dimer E. coli visade stabilitet vid en högre temperatur än normalt på grund av interaktionerna mellan de två monomera subenheterna.
Strukturen av Mycobacterium tuberculosis (Mtb) ICDH-1 bunden med NADPH och Mn(2+) bunden har lösts genom röntgenkristallografi. Det är en homodimer i vilken varje subenhet har ett Rossmann-veck och en gemensam toppdomän av sammankopplade β-ark. Mtb ICDH-1 är mest strukturellt lik den R132H mutanta humana ICDH som finns i glioblastom. I likhet med humant R132H ICDH, katalyserar Mtb ICDH-1 också bildningen av a-hydroxiglutarat.
förordning
IDH-steget i citronsyracykeln är ofta (men inte alltid) en irreversibel reaktion på grund av dess stora negativa förändring i fri energi. Det måste därför regleras noggrant för att undvika utarmning av isocitrat (och därför en ansamling av alfa-ketoglutarat). Reaktionen stimuleras av de enkla mekanismerna för substrattillgänglighet (isocitrat, NAD + eller NADP + , Mg 2+ / Mn 2+ ), produkthämning av NADH (eller NADPH utanför citronsyracykeln) och alfa-ketoglutarat och kompetitiv feedback hämning av ATP . Ett konserverat ncRNA uppströms om icd -genen som kodar för NADP + -beroende isocitratdehydrogenas (IDH) har rapporterats i bakteriegenom, på grund av dess egenskaper liknar detta ncRNA tidigare regulatoriska motiv som kallas riboswitches , icd-II ncRNA-motiv har föreslagits som ett stark kandidat riboswitch.
Katalytiska mekanismer
Isocitratdehydrogenas katalyserar de kemiska reaktionerna :
- Isocitrat + NAD + 2-oxoglutarat + CO 2 + NADH + H +
- Isocitrat + NADP + 2-oxoglutarat + CO 2 + NADPH + H +
Den totala fria energin för denna reaktion är -8,4 kJ/mol.
Steg
Inom citronsyracykeln genomgår isocitrat , framställt från isomeriseringen av citrat, både oxidation och dekarboxylering . Enzymet isocitratdehydrogenas (IDH) håller isocitrat inom sitt aktiva ställe med hjälp av de omgivande aminosyrorna , inklusive arginin , tyrosin , asparagin , serin , treonin och asparaginsyra .
I den medföljande figuren visar den första rutan den övergripande isocitratdehydrogenasreaktionen. De nödvändiga reaktanterna för denna enzymmekanism är isocitrat, NAD + / NADP + och Mn2 + eller Mg2 + . Produkterna från reaktionen är alfa-ketoglutarat , koldioxid och NADH +H + / NADPH +H + . Vattenmolekyler hjälper till att deprotonera syreatomerna i isocitrat.
Den andra rutan i figuren illustrerar steg 1 i reaktionen, vilket är oxidationen av alfa-kolet (C2 här, även kallat alfa-C). I denna process alkoholgruppen i alfa-kolet och det resulterande ensamma elektronparet bildar en ketongrupp på det kolet. NAD + /NADP + fungerar som en elektronaccepterande kofaktor och samlar upp den resulterande hydriden från C2. Oxidationen av alfakolet introducerar ett molekylärt arrangemang där elektroner (i nästa steg) kommer att flöda från den närliggande karboxylgruppen och trycka elektronerna från det dubbelbundna syret upp på själva syreatomen, som samlar en proton från ett närliggande lysin .
Den tredje rutan illustrerar steg 2, som är dekarboxyleringen av oxalosuccinat . I detta steg deprotoneras karboxylgruppens syre av en närliggande tyrosin , och dessa elektroner strömmar ner till C2. Koldioxiden, den lämnande gruppen, lossnar från beta-kolet i isocitrat (C3) och elektronerna strömmar till ketonsyren som är fäst vid alfakolet, ger en negativ laddning till den associerade syreatomen och bildar en alfa-beta-omättad dubbelbindning mellan kol 2 och 3.
Den fjärde och sista rutan illustrerar steg 3, som är mättnaden av den alfa-beta-omättade dubbelbindningen som bildades i föregående steg. Det negativt laddade syret (fäst till alfa-kolet) donerar sina elektroner, omvandlar ketondubbelbindningen och trycker ett annat ensamt par (det som bildar dubbelbindningen mellan alfa- och betakolen) "av" molekylen. Detta ensamma par plockar i sin tur upp en proton från det närliggande tyrosinet. Denna reaktion resulterar i bildningen av alfa-ketoglutarat, NADH + H + /NADPH + H + och CO 2 .
Detaljerad mekanism
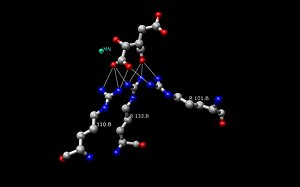
Två aspartataminosyrarester (nedan till vänster) interagerar med två intilliggande vattenmolekyler (w6 och w8) i Mn 2+ isocitrat svin IDH-komplexet för att deprotonera alkoholen från alfa-kolatomen. Oxidationen av alfa-C sker också i denna bild där NAD + accepterar en hydrid som resulterar i oxalosuccinat. Tillsammans med den stereokemiska förändringen sp 3 till sp 2 kring alfa-C finns det en ketongrupp som bildas av alkoholgruppen. Bildandet av denna ketondubbelbindning tillåter resonans att äga rum när elektroner som kommer ner från den lämnande karboxylatgruppen rör sig mot ketonen.
Dekarboxyleringen av oxalosuccinat (under mitten) är ett nyckelsteg i bildningen av alfa-ketoglutarat. I denna reaktion abstraherar det ensamma paret på den intilliggande tyrosinhydroxylen protonen från karboxylgruppen. Denna karboxylgrupp kallas också för beta-subenheten i isocitratmolekylen. Deprotoneringen av karboxylgruppen får det ensamma elektronparet att röra sig nedåt och bilda koldioxid och separera från oxalosuccinat. Elektronerna fortsätter att röra sig mot alfakolet och trycker dubbelbindningselektronerna (som gör ketonen) uppåt för att abstrahera en proton från en intilliggande lysinrest. En alfa-beta-omättad dubbelbindning resulterar mellan kol 2 och tre. Som du kan se på bilden representerar den gröna jonen antingen Mg 2+ eller Mn 2+ , vilket är en kofaktor som är nödvändig för att denna reaktion ska inträffa. Metalljonen bildar ett litet komplex genom joniska interaktioner med syreatomerna på det fjärde och femte kolet (även känd som gammasubenheten av isocitrat).
Efter att koldioxiden har splittrats från oxalosuccinatet i dekarboxyleringssteget (nedan till höger), kommer enolen att tautomerisera till keto från. Bildandet av ketondubbelbindningen startas genom deprotonering av syret från alfakolet (C#2) av samma lysin som protonerade syret i första hand. Det ensamma elektronparet rör sig nedåt och sparkar igång de ensamma paren som gjorde dubbelbindningen. Detta ensamma elektronpar abstraherar en proton från Tyrosin som deprotonerade karboxylgruppen i dekarboxyleringssteget. Anledningen till att vi kan säga att Lys- och Tyr-resterna kommer att vara desamma från föregående steg är att de hjälper till att hålla isocitratmolekylen i enzymets aktiva plats. Dessa två rester kommer att kunna bilda vätebindningar fram och tillbaka så länge de är tillräckligt nära substratet .
Isocitratdehydrogenasenzymet som anges ovan producerar alfa-ketoglutarat, koldioxid och NADH + H + / NADPH + H + . Det finns tre förändringar som inträffade under reaktionen. Oxidationen av kol 2, dekarboxyleringen (förlust av koldioxid) av kol 3 och bildningen av en ketongrupp med en stereokemisk förändring från sp 3 till sp 2 .

Porcin Mitokondriell NADP + -beroende isocitratdehydrogenas komplexbundet med Mn 2+ och isocitrat. Ytvy av den aktiva platsfickan där isocitrat begränsas av polära aminosyror.
|
Aktiv sida
Isocitrate Dehydrogenase (IDH)-enzymstrukturen i Escherichia coli var den första IDH-ortologstrukturen som klargjordes och förstods. Sedan dess Escherichia coli IDH-strukturen använts av de flesta forskare för att göra jämförelser med andra isocitratdehydrogenasenzymer. Det finns mycket detaljerad kunskap om detta bakteriella enzym, och det har visat sig att de flesta isocitratdehydrogenaser är lika i struktur och därför också i funktion. Denna likhet i struktur och funktion ger anledning att tro att strukturerna är bevarade likaväl som aminosyrorna. Därför bör de aktiva platserna bland de flesta prokaryota isocitratdehydrogenasenzymer också bevaras, vilket observeras genom många studier gjorda på prokaryota enzymer. Eukaryota isocitratdehydrogenasenzymer har å andra sidan inte helt upptäckts ännu. Varje dimer av IDH har två aktiva ställen. Varje aktivt ställe binder en NAD + /NADP + -molekyl och en tvåvärd metalljon (Mg2 + ,Mn2 + ). I allmänhet har varje aktivt ställe en konserverad sekvens av aminosyror för varje specifikt bindningsställe. I Desulfotalea psychrophila ( Dp IDH) och svin ( Pc IDH) finns tre substrat bundna till det aktiva stället.
- Isocitrat binder inom det aktiva stället till en konserverad sekvens av cirka åtta aminosyror genom vätebindningar. Dessa syror inkluderar (kan variera i rester men med liknande egenskaper) tyrosin, serin, asparagin, arginin, arginin, arginin, tyrosin och lysin. Deras positioner på ryggraden varierar men de är alla inom ett nära avstånd (dvs Arg131 DpIDH och Arg133 PcIDH, Tyr138 DpIDH och Tyr140 PcIDH).
- Metalljonen (Mg 2+ , Mn 2+ ) binder till tre konserverade aminosyror genom vätebindningar. Dessa aminosyror inkluderar tre aspartatrester.
- NAD + och NADP + binder inom det aktiva stället inom fyra regioner med liknande egenskaper bland IDH-enzymer. Dessa regioner varierar men är runt [250–260], [280–290], [300–330] och [365–380]. Återigen varierar regionerna men närhet till regioner är bevarad.
Klinisk signifikans
Specifika mutationer i isocitratdehydrogenasgenen IDH1 har hittats i flera tumörtyper. Särskilt hjärntumörer inklusive astrocytom , oligodendrogliom och glioblastoma multiforme , med mutationer som finns i nästan alla fall av sekundära glioblastom, som utvecklas från lägre grad av gliom, men sällan i primärt höggradigt glioblastoma multiforme . Patienter vars tumör hade en IDH1-mutation hade längre överlevnad. Dessutom hittades mutationer av IDH2 och IDH1 i upp till 20 % av cytogenetiskt normal akut myeloid leukemi (AML). Dessa mutationer är kända för att producera (D)-2-hydroxiglutarat från alfa-ketoglutarat. (D)-2-hydroxyglutarat ackumuleras till mycket höga koncentrationer vilket hämmar funktionen hos enzymer som är beroende av alfa-ketoglutarat. Detta leder till ett hypermetylerat tillstånd av DNA och histoner, vilket resulterar i olika genuttryck som kan aktivera onkogener och inaktivera tumörsuppressorgener. I slutändan kan detta leda till de typer av cancer som beskrivs ovan. Somatiska mosaikmutationer av denna gen har också hittats associerade med Olliers sjukdom och Maffuccis syndrom . Nya studier har dock också visat att (D)-2-hydroxiglutarat kan omvandlas tillbaka till alfa-ketoglutarat antingen enzymatiskt eller icke-enzymatiskt. Ytterligare studier krävs för att till fullo förstå rollerna av IDH1-mutation (och (D)-2-hydroxiglutarat) i cancer. Ny forskning har belyst cancerframkallande mutationer i isocitratdehydrogenas som kan orsaka ackumulering av metaboliten D-2-hydroxyglutarate (D-2HG). Notarangelo et al. visade att så höga koncentrationer av D-2HG kunde fungera som en direkt hämmare av laktatdehydrogenas i mus-T-celler. Hämning av detta metaboliska enzym förändrade glukosmetabolismen i T-cellerna och hämmade deras proliferation, cytokinproduktion och förmåga att döda målceller.
Se även
externa länkar
- Isocitratdehydrogenas: Månadens RCSB PDB-molekyl arkiverad 2013-12-24 på Wayback Machine
- Översikt över all strukturell information som finns tillgänglig i PDB för UniProt : O75874 (Isocitrate dehydrogenase [NADP] cytoplasma) vid PDBe-KB .
- Översikt över all strukturell information tillgänglig i PDB för UniProt : P48735 (Isocitrate dehydrogenase [NADP], mitokondriell) vid PDBe-KB .
- Översikt över all strukturell information som finns tillgänglig i PDB för UniProt : P50213 (Isocitrate dehydrogenase [NAD] subenhet alfa, mitokondriell) vid PDBe-KB .
- Översikt över all strukturell information tillgänglig i PDB för UniProt : O43837 (Isocitrate dehydrogenase [NAD] subunit beta, mitokondriell) vid PDBe-KB .
- Översikt över all strukturell information tillgänglig i PDB för UniProt : P51553 (Isocitrate dehydrogenase [NAD] subenhet gamma, mitokondriell) vid PDBe-KB .