Haplogrupp A-L1085
| Haplogrupp A-L1085 | |
|---|---|
| Möjlig ursprungstid | 140 000 YBP, 125 000 - 382 000 YBP |
| Möjlig ursprungsort | Central - Nordvästra Afrika |
| Förfader | Homo Y-MRCA |
| Ättlingar | A-V148 (A0), A-P305 (A1) |
| Högsta frekvenser |
Namibia (Tsumkwe San, Nama ) 60–70 % södra Sudan ( Dinka , Shilluk , Nuer ) 33 %–61,5 % Etiopien ( Beta Israel ) 41 %–46 % |
Haplogrupp A-L1085 , även känd som haplogrupp A0-T, är en human Y-DNA-haplogrupp . Det är en del av faderns släktlinje för nästan alla människor som lever idag. SNP L1085 har spelat två roller i populationsgenetik : för det första har de flesta Y-DNA-haplogrupper avvikit från den och; för det andra definierar den den odivergerade basala kladden A-L1085*.
A0-T har två primära grenar: A-V148 (även känd som haplogrupp A0) och haplogrupp A-P305 (haplogrupp A1).
Ursprung

Många förslag på haplogrupp A-L1085s ursprung tyder på att den var associerad med förfädernas befolkning av södra Afrikas jägare-samlare. Detta beror på att haplogroup A-L1085 härstamningar är frekventa bland San-folket .
A-L1085-linjerna i södra Afrika är dock undergrupper av A-linjer som finns i andra delar av Afrika, mestadels bland nilotiska folk men även bland andra afrikaner. Detta tyder på att A-L1085 linjer anlände till södra Afrika från någon annanstans. De två mest basala linjerna av Haplogroup A-L1085, A-V148 och A-P305, har upptäckts i Västafrika, Nordvästra Afrika och Centralafrika. Cruciani et al. 2011 antyder att dessa linjer kan ha dykt upp någonstans mellan Central- och Nordvästra Afrika, även om en sådan tolkning fortfarande är preliminär på grund av den ofullständiga geografiska täckningen av afrikanska y-kromosomer.
Inledande studier rapporterade att Haplogroup A-L1085-linjer uppstod för cirka 60 000 år sedan, vilket var betydligt nyare än TMRCA för mitokondriella DNA-linjer som sammansmälter till mellan 150-200 kya. Cruciani et al. 2011 med stora omstruktureringar av grenar sköt tillbaka roten av Y-kromosomträdet till 142 000 år sedan.
I november 2012, en ny studie av Scozzari et al. förstärkte "hypotesen om ett ursprung i den nordvästra kvadranten av den afrikanska kontinenten för A1b-haplogruppen , och, tillsammans med nya fynd av forntida Y-kromosomlinjer i centrala västra Afrika, tillhandahålla nya bevis angående det geografiska ursprunget för mänsklig MSY mångfald".
Geografisk fördelning
Centralafrika
Haplogrupp A-M13 har observerats i populationer i norra Kamerun (2/9 = 22% Tupuri , 4/28 = 14% Mandara, 2/17 = 12% Fulbe ) och östra DRC (2/9 = 22% Alur , 1 /18 = 6% Hema , 1/47 = 2% Mbuti ).
Haplogrupp A-M91(xA-M31,A-M6,A-M32) har observerats i Bakola -folket i södra Kamerun (3/33 = 9%).
Utan testning för någon undergrupp har haplogrupp A-L1085 observerats i prover från flera populationer i Gabon , inklusive 9 % (3/33) av ett urval av Baka , 3 % (1/36) av ett urval av Ndumu, 2 % (1/46) av ett urval av Duma , 2% (1/57) av ett urval av Nzebi och 2% (1/60) av ett urval av Tsogo .
Östafrika
Haplogrupp A-M13 är vanlig bland södra sudaneser (53 %), särskilt dinka (61,5 %). Haplogrupp A-M13 har också observerats i ett annat urval av en sydsudanesisk befolkning med en frekvens av 45 % (18/40), inklusive 1/40 A-M171. Haplogrupp A har också rapporterats i 14,6 % (7/48) av ett Amhara- prov, 10,3 % (8/78) av ett Oromo -prov, 13,6 % (12/88) av ett annat prov från Etiopien och 41 % av ett prov av Beta Israel (Cruciani et al. 2002), och viktiga procentandelar delas även av Bantus i Kenya (14 %, Luis et al. 2004) och Irak i Tanzania (3/43 = 7,0 % (Luis et al. 2004) till 1/6 = 17 % (Knight et al. 2003)).
Nordafrika
Subclade A1 har observerats hos libyska berber , medan subclade A-M13 har observerats hos cirka 3% av egyptiska män.
Södra Afrika
En studie har hittat haplogrupp A i prover av olika Khoisan -talande grupper med en frekvens som sträcker sig från 10 % till 70 %. Denna speciella haplogrupp hittades inte i ett urval av Hadzabe från Tanzania, en befolkning som ibland grupperades med andra Khoisan-grupper på grund av förekomsten av klickkonsonanter på deras språk, men vars språk grundligt visades vara ett isolat som inte är relaterat till dem som andra språk efter arbete av lingvisten Bonny Sands.
Europa
Haplogrupp A har observerats som A1 hos europeiska män i England. AY-kromosom har även observerats med låg frekvens i Mindre Asien, i Mellanöstern och på vissa Medelhavsöar, bland Egeiska greker, sicilianare (0,2 % av A1a i Capo d'Orlando och 0,5 % av A1b på hela ön) , palestinier , jordanier och jemeniter. Utan att testa för någon undergrupp har haplogrupp A1b observerats i ett urval av greker från Mitilini på den egeiska ön Lesbos och A1b har också observerats på 0,1 % av iberiska judarna . Författarna till en studie har rapporterat att de hittat vad som verkar vara haplogrupp A hos 3,1 % (2/65) av ett urval av cyprioter , även om de inte definitivt har uteslutit möjligheten att någon av dessa individer kan tillhöra haplogrupp B.
Subclade distribution
A-V148* (A0)*)
A-V148 är en av två primära grenar i A0-T.
A-P305* (A1*)
Haplogroup A-P305* är till stor del begränsad till delar av Afrika , även om en handfull fall har rapporterats i Europa och västra Asien .
A-P305 finns med sin högsta andel i Bakola Pygméer (Södra Kamerun ) med 8,3 % och berber från Algeriet med 1,5 % och i Ghana . Kladen uppnår också höga frekvenser i Bushmenjägare -samlarbefolkningen i södra Afrika , tätt följt av många nilotiska grupper i östra Afrika . Haplogrupp A:s äldsta undergrupper finns dock uteslutande i Central - Nordvästra Afrika , där den, och följaktligen den Y-kromosomala Adam, tros ha sitt ursprung för omkring 140 000 år sedan. Kladen har också observerats vid anmärkningsvärda frekvenser i vissa populationer i Etiopien , såväl som vissa pygmégrupper i Centralafrika.
Haplogroup A-L1085 är mindre vanlig bland Niger-Kongo- talare, som till stor del tillhör E1b1a- kladen. Haplogrupp E i allmänhet tros ha sitt ursprung i nordöstra Afrika, och introducerades senare till Västafrika varifrån den spreds för cirka 5 000 år sedan till centrala, södra och sydöstra Afrika med bantuexpansionen . Enligt Wood et al. (2005) och Rosa et al. (2007), förändrade sådana relativt nya befolkningsförflyttningar från Västafrika den redan existerande Y-kromosomala mångfalden i Central-, Syd- och Sydöstra Afrika, och ersatte de tidigare haplogrupperna i dessa områden med de nu dominerande E1b1a-linjerna. Spår av förfäders invånare kan dock observeras idag i dessa regioner via närvaron av Y DNA-haplogrupperna A-M91 och B-M60 som är vanliga i vissa reliktpopulationer, såsom Mbuti -pygméerna och Khoisan .
| Afrika | ||
| Studera befolkning |
Frekv. (i %) |
|
| Tsumkwe San (Namibia) | 66 % | |
| Nama (Namibia) | 64 | |
| Dinka (Sudan) | 62 | |
| Shilluk (Sudan) | 53 | |
| Nuba (Sudan) | 46 | |
| Khoisan | 44 | |
| etiopiska judar | 41 | |
| !Kung /Sekele | ~40 | |
| Borgu (Sudan) | 35 | |
| Nuer (Sudan) | 33 | |
| Päls (Sudan) | 31 | |
| massajer (Kenya) | 27 | |
| Nara (Eritrea) | 20 | |
| Masalit (Sudan) | 19 | |
| Amhara (Etiopien) | ~16 | |
| etiopier | 14 | |
| Bantu (Kenya) | 14 | |
| Mandara (Kamerun) | 14 | |
| Hausa (Sudan) | 13 | |
| Khwe (Sydafrika) | 12 | |
| Fulbe (Kamerun) | 12 | |
| Dama (Namibia) | 11 | |
| Oromo (Etiopien) | 10 | |
| Kunama (Eritrea) | 10 | |
| Sydsemitiska (Etiopien) | 10 | |
| Araber (Egypten) | 3 | |
I ett sammansatt urval av 3551 afrikanska män hade Haplogrupp A en frekvens på 5,4 %. De högsta frekvenserna av haplogrupp A har rapporterats bland Khoisan i södra Afrika, Beta Israel och Nilo-Sahara .
A-M31
Subkladen A-M31 har hittats i cirka 2,8% (8/282) av en pool av sju prover av olika etniska grupper i Guinea-Bissau , särskilt bland Papel-Manjaco-Mancanha (5/64 = 7,8%). En tidigare studie, Gonçalves et al. 2003, rapporterade att man hittade A-M31 i 5,1 % (14/276) av ett prov från Guinea-Bissau och i 0,5 % (1/201) av ett par prover från Cabo Verde . Författarna till en annan studie har rapporterat att haplogrupp A-M31 hittats i 5 % (2/39) av ett prov av Mandinka från Senegambia och 2 % (1/55) av ett prov av Dogon från Mali . Haplogroup A-M31 har också hittats i 3 % (2/64) av ett urval av berber från Marocko och 2,3 % (1/44) av ett urval av ospecificerad etnisk tillhörighet från Mali .
Minst sju män med förfäders ursprung i Yorkshire , England , och som delar det distinkta efternamnet Revis, har identifierats som tillhörande subclade A-M31. Nyhetsrapporter antydde att männen var fenotypiskt "europeiska" och omedvetna om någon afrikansk härkomst. Efterföljande forskning antydde att de delade en gemensam patrilineär förfader på 1700-talet.
A-M6
A-M6 (tidigare A2) finns vanligtvis bland Khoisan-folk. Författarna till en studie har rapporterat att haplogrupp A-M6(xA-P28) hittats i 28 % (8/29) av ett prov av Tsumkwe San och 16 % (5/32) av ett urval av !Kung/Sekele och haplogrupp A-P28 i 17 % (5/29) av ett urval av Tsumkwe San, 9 % (3/32) av ett urval av !Kung /Sekele, 9 % (1/11) av ett urval av Nama , och 6 % (1/18) av ett prov av Dama . Författarna till en annan studie har rapporterat att haplogrupp A-M6 hittats i 15,4 % (6/39) av ett urval av Khoisan-hanar, inklusive 5/39 A-M6(xA-M114) och 1/39 A-M114.
A-M32
Kladden A-M32 (tidigare A3) innehåller de mest folkrika grenarna av haplogrupp A-L1085 och finns främst i östra Afrika och södra Afrika .
A-M28
Subclade A-M28 (tidigare A3a) har endast sällan observerats på Afrikas horn . I 5 % (1/20) av ett blandat urval av talare av sydsemitiska språk från Etiopien, 1,1 % (1/88) av ett urval av etiopier och 0,5 % (1/201) i somalier.
A-M51
Subkladen A-M51 (tidigare A3b1) förekommer vanligast bland Khoisan-folken (6/11 = 55% Nama , 11/39 = 28% Khoisan, 7/32 = 22% ! Kung /Sekele, 6/29 = 21% Tsumkwe San, 1/18 = 6 % Dama). Men det har också hittats med lägre frekvens bland bantufolk i södra Afrika , inklusive 2/28 = 7% Sotho-Tswana , 3/53 = 6% icke-Khoisan sydafrikaner, 4/80 = 5% Xhosa och 1 /29 = 3 % Zulu .
A-M13
Subclade A-M13 (tidigare A3b2) som är vanligt förekommande i Östafrika och norra Kamerun skiljer sig från de som finns i Khoisan-proverna och endast på avstånd besläktad med dem. Detta fynd tyder på en gammal divergens.
I Sudan har haplogrupp A-M13 hittats i 28/53 = 52,8% av södra sudaneser , 13/28 = 46,4% av Nuba i centrala Sudan, 25/90 = 27,8% av västsudaneser , 4/32 = 12,5% av lokala Hausa-folk och 5/216 = 2,3% av norra sudaneser.
I Etiopien har en studie rapporterat att haplogrupp A-M13 hittats i 14,6 % (7/48) av ett prov av Amhara och 10,3 % (8/78) av ett prov av Oromo . En annan studie har rapporterat att haplogrupp A-M118 hittats hos 6,8 % (6/88) och haplogrupp A-M13(xA-M171, A-M118) i 5,7 % (5/88) av ett blandat prov av etiopier, vilket uppgår till totalt av 12,5% (11/88) A-M13.
Haplogrupp A-M13 har också observerats ibland utanför Central- och Östafrika, som i Egeiska regionen i Turkiet (2/30 = 6,7%), jemenitiska judar (1/20 = 5%), Egypten (4/147 = 2,7) %, 3/92 = 3,3%), palestinska araber (2/143 = 1,4%), Sardinien (1/77 = 1,3%, 1/22 = 4,5%), Jordaniens huvudstad , Amman (1/101=1) %) och Oman (1/121 = 0,8%).
Fylogenetik
Fylogenetisk historia
Före 2002 fanns det i den akademiska litteraturen minst sju namnsystem för Y-kromosomens fylogenetiska träd. Detta ledde till stor förvirring. 2002 gick de stora forskargrupperna samman och bildade Y-kromosomkonsortiet (YCC). De publicerade ett gemensamt dokument som skapade ett enda nytt träd som alla gick med på att använda. Senare bildade en grupp medborgarforskare med intresse för populationsgenetik och genetisk genealogi en arbetsgrupp för att skapa ett amatörträd som framför allt syftar till att komma i rätt tid. Tabellen nedan samlar alla dessa verk vid punkten av det landmärke YCC-trädet från 2002. Detta gör att en forskare som granskar äldre publicerad litteratur snabbt kan flytta mellan nomenklaturer.
| YCC 2002/2008 (Shorthand) | (α) | (β) | (γ) | (δ) | (ε) | (ζ) | (η) | YCC 2002 (Longhand) | YCC 2005 (Longhand) | YCC 2008 (Longhand) | YCC 2010r (Longhand) | ISOGG 2006 | ISOGG 2007 | ISOGG 2008 | ISOGG 2009 | ISOGG 2010 | ISOGG 2011 | ISOGG 2012 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A-M31 | 7 | jag | 1A | 1 | – | H1 | A | A1 | A1 | A1 | A1a | A1 | A1 | A1a | A1a | A1a | A1a | A1a |
| A-M6 | 27 | jag | 2 | 3 | – | H1 | A | A2* | A2 | A2 | A2 | A2 | A2 | A2 | A2 | A2 | A2 | A1b1a1a |
| A-M114 | 27 | jag | 2 | 3 | – | H1 | A | A2a | A2a | A2a | A2a | A2a | A2a | A2a | A2a | A2a | A2a | A1b1a1a1a |
| A-P28 | 27 | jag | 2 | 4 | – | H1 | A | A2b | A2b | A2b | A2b | A2b | A2b | A2b | A2b | A2b | A2b | A1b1a1a1b |
| A-M32 | * | * | * | * | * | * | * | * | A3 | A3 | A3 | A3 | A3 | A3 | A3 | A3 | A3 | A1b1b |
| A-M28 | 7 | jag | 1A | 1 | – | H1 | A | A3a | A3a | A3a | A3a | A3a | A3a | A3a | A3a | A3a | A3a | A1b1b1 |
| A-M51 | 7 | jag | 1A | 1 | – | H1 | A | A3b1 | A3b1 | A3b1 | A3b1 | A3b1 | A3b1 | A3b1 | A3b1 | A3b1 | A3b1 | A1b1b2a |
| A-M13 | 7 | jag | 1A | 2 | Eu1 | H1 | A | A3b2* | A3b2 | A3b2 | A3b2 | A3b2 | A3b2 | A3b2 | A3b2 | A3b2 | A3b2 | A1b1b2b |
| A-M171 | 7 | jag | 1A | 2 | Eu1 | H1 | A | A3b2a | A3b2a | A3b2a | A3b2a | A3b2a | A3b2a | A3b2a | A3b2a | A3b2a | A3b2a | tog bort |
| A-M118 | 7 | jag | 1A | 2 | Eu1 | H1 | A | A3b2b | A3b2b | A3b2b | A3b2b | A3b2b | A3b2b | A3b2b | A3b2b | A3b2b | A3b2b | A1b1b2b1 |
Ursprungliga forskningspublikationer
Följande forskargrupper per deras publikationer var representerade i skapandet av YCC-trädet.
- α Jobling och Tyler-Smith 2000 och Kaladjieva 2001
- β Underhill 2000
- γ Hammer 2001
- δ Karafet 2001
- Semino 2000
- ζ Sön 1999
- η Capelli 2001
Cruciani 2011
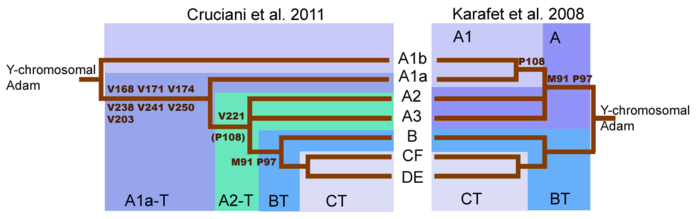
En stor förändring i förståelsen av Haplogruppen Ett träd kom med publiceringen av ( Cruciani 2011 ) . Initial sekvensering av den mänskliga y-kromosomen antydde att den första splittringen i Y-kromosomens släktträd inträffade med M91-mutationen som separerade Haplogrupp A från Haplogrupp BT . Det är dock nu känt att den djupaste splittringen i Y-kromosomträdet finns mellan två tidigare rapporterade undergrupper av Haplogrupp A, snarare än mellan Haplogrupp A och Haplogrupp BT. Subkladerna A1b och A1a-T går nu ner direkt från trädets rot. Omarrangemanget av Y-kromosomens släktträd innebär att linjer som klassificeras som Haplogrupp A inte nödvändigtvis bildar en monofyletisk kladd. Haplogrupp A hänvisar därför till en samling linjer som inte har de markörer som definierar Haplogrupp BT, även om många linjer inom haplogrupp A bara är mycket avlägset släkt.
M91- och P97-mutationerna skiljer Haplogrupp A från Haplogrupp BT . Inom Haplogrupp A-kromosomerna består M91-markören av en sträcka av 8 T-nukleobasenheter . I Haplogroup BT och schimpanskromosomer består denna markör av 9 T-nukleobasenheter . Detta mönster antydde att 9T-sträckan av Haplogroup BT var den förfäders version och att Haplogroup A bildades genom borttagning av en nukleobas .
Men enligt Cruciani et al. 2011 är regionen som omger M91-markören en mutationshotspot benägen för återkommande mutationer. Det är därför möjligt att 8T-sträckan av Haplogrupp A kan vara det förfädliga tillståndet för M91 och 9T för Haplogrupp BT kan vara det härledda tillståndet som uppstod genom en infogning av 1T. Detta skulle förklara varför subkladerna A1b och A1a-T, de djupaste grenarna av Haplogrupp A, båda har 8T-sträckan. Vidare har Cruciani et al. 2011 fastställde att P97-markören, som också används för att identifiera haplogrupp A, hade det förfäderliga tillståndet i haplogrupp A men det härledda tillståndet i Haplogrupp BT .
Filogenetiska träd
Detta fylogenetiska träd av haplogruppsundergrupper är baserat på Y-Chromosome Consortium (YCC) Tree, ISOGG Y-DNA Haplogroup Tree och efterföljande publicerad forskning.
Y-kromosomal Adam
- A0 (tidigare A1b) (P305, V148, V149, V154, V164, V166, V172, V173, V177, V190, V196, V223, V225, V229, V233, V239)
-
A1 (A1a-T enligt Cruciani 2011) (L985, L989, L990, L1002, L1003, L1004, L1009, L1013, L1053, V161, V168, V171, V174, V2803, V2803, V2403, V2803, V2403, V2803, V2803, V2803, V2803, V V250 )
- A1a (M31, P82, V4, V14, V15, V25, V26, V28, V30, V40, V48, V53, V57, V58, V63, V76, V191, V201, V204, V214, V215, V236)
-
A1b (A2-T enligt Cruciani 2011) (P108, V221)
-
A1b1 (L419)
-
A1b1a (V50, V82, V198, V224)
-
A1b1a1 tidigare A2 (M14, M23, L968/M29/P3/PN3, M71, M135, M141, M206, M276/P247, M277/P248, MEH1, P4, P5, P36.1, Sida71, Sida87, Sida87,
-
A1b1a1a (M6, M196)
-
A1b1a1a1 (M212)
- A1b1a1a1a tidigare A2a (M114)
- A1b1a1a1b tidigare A2b (P28)
- A1b1a1a1c tidigare A2c (P262)
-
A1b1a1a1 (M212)
-
A1b1a1a (M6, M196)
-
A1b1a1 tidigare A2 (M14, M23, L968/M29/P3/PN3, M71, M135, M141, M206, M276/P247, M277/P248, MEH1, P4, P5, P36.1, Sida71, Sida87, Sida87,
-
A1b1b tidigare A3 (M32)
- A1b1b1 tidigare A3a (M28, M59)
-
A1b1b2 tidigare A3b (M144, M190, M220, P289)
-
A1b1b2a tidigare A3b1 (M51, P100, P291)
- A1b1b2a1 tidigare A3b1a (P71, P102)
-
A1b1b2b tidigare A3b2 (M13, M127, M202, M219, M305):
- A1b1b2b1 (M118)
-
A1b1b2a tidigare A3b1 (M51, P100, P291)
-
A1b1a (V50, V82, V198, V224)
- BT (M42, M94, M139, M299, M60, M181/Page32, P85, P90, P97, Sida65.1/SRY1532.1/SRY10831.1, V21, V29, V31, V59, V64, V1702, V2, V102, V216, V235)
-
A1b1 (L419)
Se även
Genetik
Y-DNA A underklädes
- A-M114
- A-M118
- A-M13
- A-M171
- A-M28
- A-M31
- A-M32
- A-M51
- A-M6
- A-P28
Y-DNA ryggradsträd
Källor för konverteringstabeller
- Capelli, Cristian; Wilson, James F.; Richards, Martin; Stumpf, Michael PH; et al. (februari 2001). "Ett övervägande inhemskt fadersarv för de austronesisktalande folken i öarna i Sydostasien och Oceanien" . American Journal of Human Genetics . 68 (2): 432–443. doi : 10.1086/318205 .
- Hammer, Michael F.; Karafet, Tatiana M.; Redd, Alan J.; Jarjanazi, Hamdi; et al. (1 juli 2001). "Hierarkiska mönster av global mänsklig Y-kromosommångfald" . Molekylärbiologi och evolution . 18 (7): 1189–1203. doi : 10.1093/oxfordjournals.molbev.a003906 .
- Jobling, Mark A.; Tyler-Smith, Chris (2000), "New uses for new haplotypes", Trends in Genetics , 16 (8): 356–62, doi : 10.1016/S0168-9525(00)02057-6 , PMID 10904265
- Kaladjieva, Luba; Calafell, Francesc; Jobling, Mark A; Angelicheva, Dora; et al. (februari 2001). "Mönster av genetisk mångfald mellan och inom gruppen i Vlax Roma som avslöjats av Y-kromosomer och mitokondriella DNA-linjer" . European Journal of Human Genetics . 9 (2): 97–104. doi : 10.1038/sj.ejhg.5200597 .
- Karafet, Tatiana; Xu, Liping; Du, Ruofu; Wang, William; et al. (september 2001). "Paternal Population History of East Asia: Källor, mönster och mikroevolutionära processer" . American Journal of Human Genetics . 69 (3): 615–628. doi : 10.1086/323299 .
- Semino, O.; Passarino, G; Oefner, PJ; Lin, AA; et al. (2000), "The Genetic Legacy of Paleolithic Homo sapiens sapiens in Extant Europeans: AY Chromosome Perspective", Science , 290 (5494): 1155–9, Bibcode : 2000Sci...290.1155S , doi : 6.419.4190/10.419. 1155 , PMID 11073453
- Su, Bing; Xiao, Junhua; Underhill, Peter; Deka, Ranjan; et al. (december 1999). "Y-kromosombevis för en nordlig migration av moderna människor till östra Asien under den senaste istiden" . American Journal of Human Genetics . 65 (6): 1718–1724. doi : 10.1086/302680 .
- Underhill, Peter A.; Shen, Peidong; Lin, Alice A.; Jin, Li; et al. (november 2000). "Y-kromosomsekvensvariation och mänskliga populationers historia". Naturgenetik . 26 (3): 358–361. doi : 10.1038/81685 .