Katalytiskt intron i grupp I
| Grupp I katalytisk intron | |
|---|---|
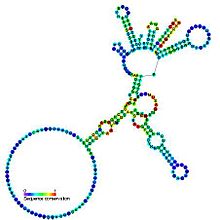 Förutspådd sekundär struktur och sekvenskonservering av Grupp I katalytiska
| |
| intronidentifierare | |
| Symbol | Intron_gpI |
| Rfam | RF00028 |
| Övriga uppgifter | |
| RNA -typ | Intron |
| Domän(er) | Eukaryota ; Bakterier ; Virus |
| GÅ | GO:0000372 |
| SÅ | SO:0000587 |
| PDB- strukturer | PDBe |
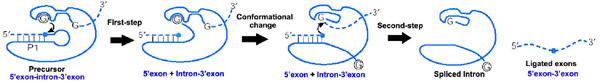
Grupp I-introner är stora självskarvande ribozymer . De katalyserar sin egen excision från mRNA- , tRNA- och rRNA- prekursorer i ett brett spektrum av organismer. Den sekundära kärnstrukturen består av nio parade regioner (P1-P9). Dessa vikas till i huvudsak två domäner – P4-P6-domänen (bildad från staplingen av P5-, P4-, P6- och P6a-helixar) och P3-P9-domänen (bildad från P8-, P3-, P7- och P9-helixarna). Den sekundära strukturpåslaget för denna familj representerar endast denna bevarade kärna. Grupp I- introner har ofta långa öppna läsramar infogade i loopregioner .
Katalys
Splitsning av grupp I-introner bearbetas genom två sekventiella transesterifieringsreaktioner . Den exogena guanosin- eller guanosinnukleotiden ( exoG ) dockar först på det aktiva G-bindningsstället i P7, och dess 3'-OH är inriktat för att attackera fosfodiesterbindningen vid 5'-splitsningsstället i P1, vilket resulterar i en fri 3 '-OH-gruppen vid exonen uppströms och exoG är fäst vid 5'-änden av intronen. Sedan byter intronens terminala G (omega G) exoG och upptar G-bindningsstället för att organisera den andra esteröverföringsreaktionen: 3'-OH-gruppen i exonen uppströms i P1 är inriktad för att attackera 3'-skarvningen plats i P10, vilket leder till ligering av de intilliggande exonerna uppströms och nedströms och frisättning av det katalytiska intronet.
Två-metalljonmekanism som ses i proteinpolymeraser och fosfataser föreslogs användas av grupp I- och grupp II-introner för att bearbeta fosforylöverföringsreaktionerna, vilket otvetydigt bevisades av en högupplöst struktur av Azoarcus grupp I-intron 2006.
Intronvikning
Sedan början av 1990-talet började forskare studera hur grupp I-intron uppnår sin naturliga struktur in vitro , och vissa mekanismer för RNA- veckning har hittills uppskattats. Det är överens om att den tertiära strukturen viks efter bildandet av den sekundära strukturen. Under veckning fylls RNA-molekyler snabbt in i olika veckningsintermediärer, mellanprodukterna som innehåller naturliga interaktioner viks ytterligare in i den naturliga strukturen genom en snabb veckningsväg, medan de som innehåller icke-native intermediärer fångas i metastabila eller stabila icke-native konformationer , och processen för omvandling till den inhemska strukturen sker mycket långsamt. Det är uppenbart att grupp I-introner som skiljer sig åt i uppsättningen av perifera element uppvisar olika potentialer för att komma in i den snabba vikningsvägen. Samtidigt är samverkande sammansättning av den tertiära strukturen viktig för vikningen av den ursprungliga strukturen. Icke desto mindre möter veckning av grupp I-introner in vitro både termodynamiska och kinetiska utmaningar. Ett fåtal RNA-bindande proteiner och chaperoner har visats främja veckningen av grupp I-introner in vitro och i bakterier genom att stabilisera de nativa mellanprodukterna respektive genom att destabilisera de icke-naturliga strukturerna.
Distribution, fylogeni och rörlighet
Grupp I-introner är fördelade i bakterier, lägre eukaryoter och högre växter. Deras förekomst i bakterier verkar dock vara mer sporadisk än i lägre eukaryoter, och de har blivit vanliga i högre växter. Generna som grupp I-introner avbryter skiljer sig markant: De avbryter rRNA- , mRNA- och tRNA -gener i bakteriegenom, såväl som i mitokondriella och kloroplastgenom hos lägre eukaryoter, men invaderar bara rRNA-gener i kärngenomet hos lägre eukaryoter. I högre växter verkar dessa introner vara begränsade till ett fåtal tRNA- och mRNA-gener från kloroplasterna och mitokondrierna.
Grupp I-introner finns också insatta i gener av en mängd olika bakteriofager av grampositiva bakterier . Emellertid är deras distribution i fagen av gramnegativa bakterier huvudsakligen begränsad till T4 , T-even och T7-liknande bakteriofager.
Både intron-tidiga och intron-sena teorier har hittat bevis för att förklara ursprunget till grupp I-introner. Vissa grupp I-introner kodar för homing-endonukleas (HEG), som katalyserar intronmobilitet. Det föreslås att HEG:er flyttar intronen från en plats till en annan, från en organism till en annan och därmed förklarar den breda spridningen av de själviska grupp I-intronerna. Ingen biologisk roll har identifierats för grupp I-introner hittills förutom att splitsning av sig själva från prekursorn för att förhindra döden av värden som de lever av. Ett litet antal grupp I-introner har också visat sig koda för en klass av proteiner som kallas maturaser som underlättar intronsplitsningen .
Se även
- Intron
- Grupp I Intronsekvens- och strukturdatabas
- Splitsplats
- Nukleära introner
- Grupp II-intron
- Grupp III intron
- Twintron
- LtrA
- Cyklisk di-GMP-II riboswitch , där det finns ett exempel på en riboswitch som verkar tillsammans med en grupp I-intron för att reglera uttrycket av en gen
Vidare läsning
- Chauhan, S; Caliskan G; Briber RM; Perez-Salas U; Rangan P; Thirumalai D; Woodson SA (2005). "RNA-tertiära interaktioner förmedlar naturlig kollaps av en bakteriegrupp I-ribozym". J Mol Biol . 353 (5): 1199–1209. doi : 10.1016/j.jmb.2005.09.015 . PMID 16214167 .
- Haugen, P; Simon DM; Bhattacharya D (2005). "The natural history of group I introns". Trender inom genetik . 21 (2): 111–119. doi : 10.1016/j.tig.2004.12.007 . PMID 15661357 .
- Rangan, P; Masquida, B; Westhof E; Woodson SA (2003). "Montering av kärnspiraler och snabb tertiär vikning av en liten bakteriegrupp I-ribozym" . Proc Natl Acad Sci USA . 100 (4): 1574–1579. Bibcode : 2003PNAS..100.1574R . doi : 10.1073/pnas.0337743100 . PMC 149874 . PMID 12574513 .
- Schroeder, R; Barta A; Semrad K (2004). "Strategier för RNA-vikning och montering". Nat Rev Mol Cell Biol . 5 (11): 908–919. doi : 10.1038/nrm1497 . PMID 15520810 . S2CID 22030359 .
- Thirumalai, D; Lee N; Woodson SA; Klimov D (2001). "Tidiga händelser i RNA-veckning". Annu Rev Phys Chem . 52 : 751-762. Bibcode : 2001ARPC...52..751T . doi : 10.1146/annurev.physchem.52.1.751 . PMID 11326079 .
- Treiber, DK; Williamson JR (1999). "Exponera de kinetiska fällorna i RNA-veckning". Curr Opin Struct Biol . 9 (3): 339–345. doi : 10.1016/S0959-440X(99)80045-1 . PMID 10361090 .
- Xiao, M; Leibowitz MJ; Zhang Y (2003). "Samordnad veckning av ett Candida-ribozym till den katalytiskt aktiva strukturen efter en snabb RNA-komprimering" . Nucleic Acids Res . 31 (14): 3901–3908. doi : 10.1093/nar/gkg455 . PMC 165970 . PMID 12853605 .