Avlockningskomplex
MRNA - avkapslingskomplexet är ett proteinkomplex i eukaryota celler som ansvarar för avlägsnande av 5'-kapseln . Det aktiva enzymet i decapping-komplexet är det tvålobade Nudix-familjens enzym Dcp2 , som hydrolyserar 5' cap och frisätter 7mGDP och ett 5'-monofosforylerat mRNA. Detta avkapslade mRNA hämmas för translation och kommer att brytas ned av exonukleaser . Kärnavkapslingskomplexet är bevarat i eukaryoter. Dcp2 aktiveras av Decapping Protein 1 (Dcp1) och i högre eukaryoter förenade av ställningsproteinet VCS. Tillsammans med många andra tillbehörsproteiner samlas avkapslingskomplexet i P-kroppar i cytoplasman .
Syftet med avkapslingskomplexet
mRNA måste brytas ned, annars kommer det att fortsätta flyta runt cellen och skapa oönskade proteiner på måfå. mRNA 5'-kåpan är speciellt utformad för att förhindra att mRNA bryts ned innan den kan användas, och måste därför tas bort så att mRNA-sönderfallsvägen kan ta hand om det.
Avlockningsmekanism
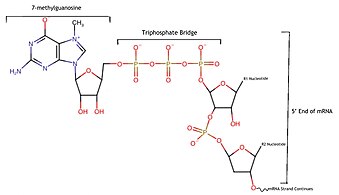
Dcp2 är proteinet som faktiskt avkapslar mRNA, och resten av proteinerna i komplexet förbättrar dess funktion och låter det hydrolysera den kemiska bindningen som fäster mRNA:t till 5'-höljet . Nudix-domänen i Dcp2 hydrolyserar en av bindningarna på trifosfatbryggan som hakar ihop mRNA och 5'-kåpan , vilket gör att 7-metylguanosin- kåpan lossnar och lämnar mRNA:n öppen för nedbrytning av exonukleaserna i cellen.
Struktur av avkapslingskomplexet
Både encelliga och flercelliga organismer behöver avkapsla sitt mRNA för att bli av med det, men olika organismer har lite olika proteiner som utför denna process. Det finns många proteiner som förblir desamma, men flera viktiga skillnader mellan de encelliga ( jäst ) och flercelliga ( metazoaniska ) decapping-komplexen.
Jästtäckningskomplex
I jäst ( S. cerevisiae ) förenas Dcp2 av den decapping-aktivatorn Dcp1, helikaset Dhh1, exonukleaset Xrn1, nonsensförmedlade sönderfallsfaktorer Upf1, Upf2 och Upf3, LSm- komplexet, Pat1 och olika andra proteiner. Dessa proteiner lokaliseras alla till cytoplasmatiska strukturer som kallas P-kroppar . Särskilt i jäst finns det inga translationsfaktorer eller ribosomala proteiner inuti P-kroppar.
Metazoantäckningskomplex
Högre eukaryoter har något olika medlemmar av decapping komplexet. Enzymet Dcp2 är fortfarande den katalytiska subenheten som bildar ett holoenzym med Dcp1 och interagerar med hjälpproteiner såsom Xrn1 , Upf1 , Upf2 , Upf3 , LSm-komplexet och Dhh1-ortologen DDX6 . Proteiner som är unika för växter och däggdjur inkluderar betapropellerproteinet Hedls och förstärkaren av decapping Edc3. Forskare vet hur komplexet fysiskt associerar på grund av immunfällning , medan strukturella detaljer för varje del av komplexet har upptäckts genom att använda röntgenkristallografi i samband med proteinkristallisering . Vart och ett av dessa proteiner bidrar med olika saker till decapping-komplexet, som diskuteras nedan.
Dcp2
Dcp2 , som den huvudsakliga katalysatorn för avkapslingsprocessen, förlitar sig på ett specifikt mönster av aminosyror som kallas en nudix-domän för att anpassa sig till 5'-locket för att hydrolysera det. En nudix- domän skapas genom att packa två beta-ark mellan flera alfaspiraler , kan ha olika längder och storlekar, och används vanligtvis av proteiner för att utföra defosforylering , för att bli av med ett fosfat genom att infoga en vattenmolekyl i bindningen mellan fosfatet och resten av molekylen. I fallet med Dcp2 innehåller det flera glutaminsyrasidokedjor som är negativt laddade under normala cellulära förhållanden, och det är dessa som gör att proteinet kan manipulera vattenmolekyler för att hydrolysera trifosfatbryggan som förbinder 5'-änden av mRNA:t till 7 -metylguanosinlocket . Därför är nudix-domänen det som tillåter Dcp2 att ta bort 5'-locket, vilket resulterar i skapandet av 7mGDP, en 7-metylguanosin med två fosfatgrupper anslutna och en monofosforylerad mRNA-sträng.
Innan nudix-domänen finns en N-terminal regulatorisk domän (NRD), som ytterligare hjälper till att hydrolysera 5'-mRNA-kapseln. Efter nudix-domänen finns ett C-terminalt område som kallas Box B, vilket hjälper till att binda Dcp2 till RNA. Med alla dessa tre huvudmotiv kan Dcp2 hitta, binda fast till och hydrolysera ett 5'-mRNA-lock. Den gör detta antingen genom att känna igen en hårnålsögla i RNA:t inom 10 baspar från locket, vilket kallas ett Dcp2-bindande och decapping-element, eller genom att ett separat protein känner igen ett basparmönster i mRNA:t och direkt rekryterar Dcp2-Dcp1. holoenzym. Tyvärr fungerar Dcp2 långsamt och behöver några andra proteiner för att koordinera med det så att det kan dekapa mRNA i tid.
Dcp1
Dcp1 är en regulatorisk subenhet, den kombineras med Dcp2, vilket skapar ett holoenzym som kan dekapa mRNA ordentligt. Utan Dcp1 är det faktiskt omöjligt för Dcp2 att dekapsla något in vivo , och det fungerar bara otroligt långsamt in vitro , vilket gör bildandet av detta holoenzym till en viktig process vid avkapsling.
Dcp1:s sekundära struktur består av sju betablad och tre alfaspiraler . som går samman för att bilda en V-formad tertiär struktur . De definierande egenskaperna hos Dcp1 är EVH1-domänen och en domän som känner igen prolinrika sekvenser (PRS) på andra proteiner. EVH1-domänen interagerar direkt med den tidigare nämnda NRD av Dcp2 och tros för närvarande direkt hjälpa till med decapping av mRNA, även om hur det gör det är oklart. Domänen som känner igen PRS är gjord av mestadels hydrofoba aminosyror och finns inom klyftan av "V" i Dcp1-strukturen. Det används för att binda till andra proteiner i decapping-komplexet till Dcp1.
PNRC2
PNRC2 fäster till och förstärker effekten av Dcp1 för att uppmuntra decapping, och rekryterar även Upf1 till decapping-komplexet. Den har en prolinrik sekvens som är hydrofob och fäster starkt vid den lika hydrofoba klyftan i Dcp1, och så binder Dcp1 PNRC2 :s prolinrika region, vilket sedan förstärker funktionen av Dcp2 ännu mer. Aktuell forskning tyder på att PNRC2 hjälper till att associera Dcp2 och Dcp1 tillsammans, vilket gör Dcp2-Dcp1 holoenzymet mer stabilt och ökar därför effektiviteten av Dcp2, men de exakta detaljerna om hur det gör det är vaga. Rekryteringen av Upf1 tillåter decapping-komplexet att delta i nonsens-medierat mRNA-sönderfall, vilket gör PNRC2 till ett sätt för Dcp2 att ansluta till den regulatoriska vägen som ansvarar för att förstöra felaktigt transkripterat mRNA.
Upp1-3
Upf1 , Upf2 och Upf3 är proteiner involverade i den regulatoriska vägen för nonsens-medierad mRNA-nedbrytning, och inte den faktiska decappingen av mRNA. Endast Upf1 fäster direkt till decapping-komplexet, medan Upf2 och Upf3 fäster till mRNA, fäster sedan till Upf1 för att underlätta förstörelsen av felaktigt mRNA. Dessa är aktivatorer av komplexet, genom att de kan rikta komplexet mot felaktigt bildat mRNA, men faktiskt inte hjälper till att dekapsla mRNA.
DDX6
DDX6 , en ortolog av Dhh1, förbättrar också effektiviteten hos Dcp2-Dcp1 holoenzymet samtidigt som det hydrolyserar 5'-locket. Det föreslås att, eftersom det är en helikas , är det involverat i att omkonfigurera 5'-änden av mRNA:t för att ge Dcp2 enklare åtkomst till 5'-locket, och att det stimulerar Dcp1 så att det interagerar bättre med Dcp2 när det fästs till resten av avkapslingskomplexet.
Edc3
Edc3 aktiverar ytterligare Dcp2-Dcp1 holoenzymet och gör det möjligt för det att snabbt avkapsla mRNA. Den har en LSm-domän vid sin N-terminal , som interagerar med specifika aminosyramotiv som kallas HLM-fragment som finns på C-terminalen av Dcp1 och tillåter Edc3 att binda till den. En annan viktig del av detta protein är FDF-linkern, som är en lång och ostrukturerad sträcka av aminosyror som binder till DDX6 och stoppar den från att binda direkt till mRNA:t, vilket gör att den istället kan interagera med proteinerna i decapping-komplexet. Den sista anmärkningsvärda domänen är en Yjef-N C-terminal domän som dimeriserar med mRNA och hjälper till att skapa P-kroppar runt platsen för avkapslingskomplexet.
P-kroppar är i huvudsak lagrade klumpar av avkaplat eller undertryckt mRNA blandat med mRNA-nedbrytningsfaktorer, såsom decapping-komplexet och det nonsens-medierade mRNA-sönderfallsmaskineriet, så de är viktiga för den eventuella förstörelsen av mRNA som förändrats av Dcp2. Eftersom Edc3 skapar P-kroppar runt decapping komplexet, blir det lättare för Dcp2 att hitta mRNA 5' caps att hydrolysera, vilket ökar effektiviteten av hela komplexet.
Pat1
Pat1 är ett annat protein som ökar effektiviteten hos decapping-komplexet. Den har tre huvuddomäner. En är nödvändig för att ta bort mRNA och hjälper direkt Dcp2-Dcp1 holoenzymet att göra det. De andra två gör det lättare för proteinet att dekapsla mRNA, men är inte direkt involverade i hydrolysen av fosfatbindningen. Pat1 har många interaktioner med de olika proteinerna i avkapslingskomplexet, och är känt som "byggnadsställningsproteinet" eftersom det samlar allt när det är dags att tappa något. N-terminaldomänen interagerar med DXX6 och för den nära så att den kan aktivera Dcp1, en annan del hjälper till att skapa P-kroppar tillsammans med Edc3, och C-terminaldomänerna fäster Dcp1–Dcp2, Lsm1–7-komplexet och Xrn1 till komplexet .
Xrn1
Xrn1 är ett 5' till 3' exonukleas som bryter ned det just avkapslade mRNA:t. Den riktar sig mot 5'-monofosfatänden av mRNA, vilket är det som blir över när Dcp2 har hydrolyserat locket och tagit bort 7- metylguanosin- locket, tillsammans med två av de tre fosfaterna som fäster locket till mRNA. Den nuvarande teorin är att strukturen av Xrn1 inte tillåter ett capped mRNA att interagera med det eftersom Xrn1 är strukturerat på ett sådant sätt att det finns steriska hinder som fysiskt blockerar proteinet från att interagera med något mRNA som Dcp2 inte redan har avlockat.