Virtuell karyotyp
Virtuell karyotyp är den digitala informationen som återspeglar en karyotyp , resultatet av analysen av korta sekvenser av DNA från specifika loci över hela genomet, som isoleras och räknas upp. Den upptäcker variationer i genomiskt antal kopior vid en högre upplösning för nivå än konventionell karyotypning eller kromosombaserad jämförande genomisk hybridisering (CGH). De huvudsakliga metoderna som används för att skapa virtuella karyotyper är array-jämförande genomisk hybridisering och SNP-arrayer .
Bakgrund
En karyotyp (Fig 1) är det karakteristiska kromosomkomplementet för en eukaryot art . En karyotyp presenteras vanligtvis som en bild av kromosomerna från en enda cell ordnade från störst (kromosom 1) till minsta (kromosom 22), med könskromosomerna (X och Y) sist. Historiskt sett har karyotyper erhållits genom färgning av celler efter att de har stoppats kemiskt under celldelning. Karyotyper har använts i flera decennier för att identifiera kromosomavvikelser i både könsceller och cancerceller. Konventionella karyotyper kan bedöma hela genomet för förändringar i kromosomstruktur och antal, men upplösningen är relativt grov, med en detektionsgräns på 5-10Mb. [ citat behövs ]

Metod
Nyligen har plattformar för att generera högupplösta karyotyper i silico från stört DNA dykt upp, såsom array comparative genomic hybridization (arrayCGH) och SNP-arrays . Konceptuellt är arrayerna sammansatta av hundratals till miljoner prober som är komplementära till en region av intresse i genomet. Det avbrutna DNA:t från testprovet fragmenteras, märks och hybridiseras till arrayen. Hybridiseringssignalintensiteterna för varje prob används av specialiserad mjukvara för att generera ett log2-förhållande mellan test/normal för varje prob på arrayen. [ citat behövs ]
Genom att känna till adressen för varje sond på arrayen och adressen för varje sond i genomet, radar programvaran upp proberna i kromosomal ordning och rekonstruerar genomet i silico (Fig 2 och 3).
Virtuella karyotyper har dramatiskt högre upplösning än konventionell cytogenetik. Den faktiska upplösningen kommer att bero på densiteten av sonder på arrayen. För närvarande Affymetrix SNP6.0 den kommersiellt tillgängliga arrayen med högsta densitet för virtuella karyotypapplikationer. Den innehåller 1,8 miljoner polymorfa och icke-polymorfa markörer för en praktisk upplösning på 10-20 kb - ungefär lika stor som en gen. Detta är ungefär 1000 gånger högre upplösning än karyotyper erhållna från konventionell cytogenetik. [ citat behövs ]
Virtuella karyotyper kan utföras på könslinjeprover för konstitutionella störningar, och kliniska tester är tillgängliga från dussintals CLIA-certifierade laboratorier ( genetests.org ). Virtuell karyotypning kan också göras på färska eller formalinfixerade paraffininbäddade tumörer. CLIA-certifierade laboratorier som erbjuder testning av tumörer inkluderar Creighton Medical Laboratories (färska och paraffininbäddade tumörprover) och CombiMatrix Molecular Diagnostics (färska tumörprover).
Olika plattformar för virtuell karyotyping
Array-baserad karyotypning kan göras med flera olika plattformar, både laboratorieutvecklade och kommersiella. Arrayerna själva kan vara genomomfattande (sonder fördelade över hela genomet) eller riktade (sonder för genomiska regioner som är kända för att vara involverade i en specifik sjukdom) eller en kombination av båda. Vidare kan arrayer som används för karyotypning använda icke-polymorfa prober, polymorfa prober (dvs SNP-innehållande) eller en kombination av båda. Icke-polymorfa prober kan endast ge information om kopienummer, medan SNP-matriser kan ge både kopienummer och LOH-status (loss-of-heterozygosity) i en analys. De sondtyper som används för icke-polymorfa arrayer inkluderar cDNA, BAC-kloner (t.ex. BlueGnome ) och oligonukleotider (t.ex. Agilent , Santa Clara, CA, USA eller Nimblegen , Madison, WI, USA). Kommersiellt tillgängliga oligonukleotid-SNP-matriser kan vara fastfas ( Affymetrix , Santa Clara, CA, USA) eller pärlbaserade ( Illumina , San Diego, CA, USA). Trots mångfalden av plattformar använder de i slutändan alla genomiskt DNA från störda celler för att återskapa en högupplöst karyotyp i silico . Slutprodukten har ännu inte ett konsekvent namn och har kallats virtuell karyotypning, digital karyotypning, molekylär allelokaryotypning och molekylär karyotypning. Andra termer som används för att beskriva de arrayer som används för karyotypning inkluderar SOMA (SNP-oligonukleotidmikroarrayer) och CMA (kromosommikroarray). Vissa anser att alla plattformar är en typ av array-jämförande genomisk hybridisering (arrayCGH), medan andra reserverar den termen för tvåfärgningsmetoder, och ytterligare andra segregerar SNP-matriser eftersom de genererar mer och annan information än tvåfärgade arrayCGH-metoder. [ citat behövs ]
Ansökningar
Upptäcker ändringar av kopienummer
Förändringar i antal kopior kan ses i både könsceller och tumörprover. Ändringar av antal kopior kan detekteras av arrayer med icke-polymorfa prober, såsom arrayCGH, och av SNP-baserade arrayer. Människor är diploida, så ett normalt antal kopior är alltid två för icke-könade kromosomer. [ citat behövs ]
- Borttagningar: En radering är förlust av genetiskt material. Borttagningen kan vara heterozygot (kopiatal 1) eller homozygot (kopiatal 0, nullisomi). Mikrodeletionssyndrom är exempel på konstitutionella störningar på grund av små deletioner i könscellers DNA. Deletioner i tumörceller kan representera inaktiveringen av en tumörsuppressorgen och kan ha diagnostiska, prognostiska eller terapeutiska implikationer.
- Vinster: En ökning av antalet kopior representerar vinsten av genetiskt material. Om vinsten är av bara en ytterligare kopia av ett segment av DNA, kan det kallas en duplicering (Fig 4). Om det finns en extra kopia av en hel kromosom kan den kallas trisomi . Ökningen av antalet kopior i könslinjeprover kan vara sjukdomsassocierade eller kan vara en godartad variant av kopia . När de ses i tumörceller kan de ha diagnostiska, prognostiska eller terapeutiska implikationer.
- Förstärkningar: Tekniskt sett är en förstärkning en typ av kopietalsförstärkning där det finns ett kopietal >10. I samband med cancerbiologi ses amplifieringar ofta i onkogener . Detta kan indikera en sämre prognos, hjälpa till att kategorisera tumören eller indikera läkemedelsberättigande. Ett exempel på läkemedelsberättigande är Her2Neu-amplifiering och Herceptin , och en bild av Her2Neu-amplifiering detekterad genom virtuell karyotypning av SNP-matriser tillhandahålls (fig 5).
Förlust av heterozygositet (LOH), autozygota segment och uniparental disomi
Autozygota segment och uniparental disomi (UPD) är diploida/'kopieringsneutrala' genetiska fynd och kan därför endast detekteras av SNP-baserade arrayer. Både autozygota segment och UPD kommer att visa förlust av heterozygositet (LOH) med ett antal kopior på två genom SNP-array karyotyping. Termen Runs of Homozgygosity (ROH), är en generisk term som kan användas för antingen autozygota segment eller UPD. [ citat behövs ]
- Autozygot segment: Ett autozygott segment är bi-parentalt och ses endast i könslinjen. De är utökade körningar av homozygota markörer i genomet, och de uppstår när ett identiskt haplotypblock ärvs från båda föräldrarna. De kallas också " identiska av descent " (IBD) segment, och de kan användas för homozygositetskartläggning.
- Uniparental disomi: UPD uppstår när båda kopiorna av en gen eller genomisk region ärvs från samma förälder. Detta är uniparentalt, i motsats till autozygota segment som är bi-parentala. När de finns i könscellerna kan de vara ofarliga eller förknippade med sjukdomar, såsom Prader-Willi eller Angelmans syndrom . Också i motsats till autozygositet kan UPD utvecklas i tumörceller, och detta kallas förvärvad UPD eller kopieringsneutral LOH i litteraturen (Fig 6). Förvärvad UPD är ganska vanligt i både hematologiska och solida tumörer och rapporteras utgöra 20 till 80 % av LOH som ses i mänskliga tumörer. Förvärvad UPD kan fungera som den andra träffen i Knudson Two Hit Hypothesis of Tumorigenesis, och kan således vara den biologiska motsvarigheten till en deletion. Eftersom denna typ av lesion inte kan detekteras av arrayCGH, FISH eller konventionell cytogenetik, föredras SNP-baserade arrays för virtuell karyotypning av tumörer.
Figur 7 är en virtuell karyotyp av SNP-array från ett kolorektalt karcinom som visar deletioner, vinster, amplifieringar och förvärvad UPD (kopiera neutral LOH).
Exempel på kliniska cancerapplikationer
En virtuell karyotyp kan genereras från nästan vilken tumör som helst, men den kliniska innebörden av de identifierade genomiska aberrationerna är olika för varje tumörtyp. Den kliniska användbarheten varierar och lämpligheten avgörs bäst av en onkolog eller patolog i samråd med laboratoriechefen för labbet som utför den virtuella karyotypen. Nedan finns exempel på typer av cancer där de kliniska implikationerna av specifika genomiska aberrationer är väl etablerade. Denna lista är representativ, inte uttömmande. Webbplatsen för Cytogenetics Laboratory vid Wisconsin State Laboratory of Hygiene har ytterligare exempel på kliniskt relevanta genetiska förändringar som lätt kan upptäckas genom virtuell karyotypning. [1]
Neuroblastom
Baserat på en serie av 493 neuroblastomprover har det rapporterats att det övergripande genomiska mönstret, som testats med array-baserad karyotyping, är en prediktor för utfallet i neuroblastom:
- Tumörer som uteslutande uppvisar förändringar i antal kopior av hela kromosomer var förknippade med utmärkt överlevnad.
- Tumörer som uppvisade någon form av förändringar i antal segmentella kromosomkopior var förknippade med en hög risk för återfall.
- Inom tumörer som visar segmentella förändringar var ytterligare oberoende prediktorer för minskad total överlevnad MYCN-amplifiering, 1p och 11q deletioner och 1q gain.
Tidigare publikationer kategoriserade neuroblastom i tre huvudsubtyper baserat på cytogenetiska profiler:
- Subtyp 1: gynnsamt neuroblastom med nästan triploidi och en övervägande del av numeriska vinster och förluster, som mest representerar icke-metastaserande NB-stadier 1, 2 och 4S.
- Subtyper 2A och 2B: hittas i ogynnsamt utbrett neuroblastom, steg 3 och 4, med 11q förlust och 17q förstärkning utan MYCN-förstärkning (subtyp 2A) eller med MYCN-förstärkning ofta tillsammans med 1p-deletioner och 17q-förstärkning (subtyp 2B).
Wilms tumör
Tumörspecifik förlust av heterozygositet (LOH) för kromosom 1p och 16q identifierar en undergrupp av Wilms tumörpatienter som har en signifikant ökad risk för återfall och död. LOH för dessa kromosomala regioner kan nu användas som en oberoende prognostisk faktor tillsammans med sjukdomsstadiet för att rikta behandlingens intensitet mot risken för behandlingsmisslyckande.
Njurcellscancer
Renala epiteliala neoplasmer har karakteristiska cytogenetiska avvikelser som kan hjälpa till med klassificeringen. Se även Atlas of Genetics and Cytogenetics in Oncology and Hematology .
- Klarcellscancer: förlust av 3p
- Papillärt karcinom: trisomi 7 och 17
- Kromofob karcinom: hypodiploid med förlust av kromosomerna 1, 2, 6, 10, 13, 17, 21
Array-baserad karyotypning kan användas för att identifiera karakteristiska kromosomavvikelser i njurtumörer med utmanande morfologi. Array-baserad karyotypning fungerar bra på paraffininbäddade tumörer och är mottaglig för rutinmässig klinisk användning.
Dessutom indikerar nyare litteratur att vissa kromosomavvikelser är associerade med resultat i specifika subtyper av njurepiteltumörer. Klarcellsnjurkarcinom: del 9p och del 14q är dåliga prognostiska indikatorer. Papillärt njurcellscancer: duplicering av 1q markerar dödlig progression.
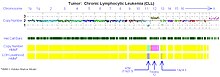
Kronisk lymfatisk leukemi

Array-baserad karyotypning är ett kostnadseffektivt alternativ till FISH för att upptäcka kromosomavvikelser vid kronisk lymfatisk leukemi (KLL). Flera kliniska valideringsstudier har visat >95 % överensstämmelse med standardpanelen CLL FISH. Dessutom har många studier som använder array-baserad karyotypning identifierat "atypiska deletioner" som missats av FISH-standardproberna och förvärvat uniparental disomi vid nyckellokus för prognostisk risk vid KLL.
Fyra huvudsakliga genetiska avvikelser känns igen i CLL-celler som har stor inverkan på sjukdomsbeteende.
- Borttagningar av en del av den korta armen av kromosom 17 (del 17p) som riktar sig mot p53 är särskilt skadliga. Patienter med denna abnormitet har betydligt kortare intervall innan de behöver terapi och en kortare överlevnad. Denna abnormitet finns hos 5–10 % av patienterna med KLL.
- Borttagningar av den långa armen på kromosom 11 (del 11q) är också ogynnsamma men inte i den grad som setts med del 17p. Avvikelsen riktar sig mot ATM-genen och förekommer sällan i KLL (5–10%).
- Trisomi 12, en ytterligare kromosom 12, är ett relativt frekvent fynd som förekommer hos 20–25 % av patienterna och ger en mellanliggande prognos.
- Borttagning av 13q14 (del 13q14) är den vanligaste abnormiteten vid KLL med ungefär 50 % av patienterna med celler som innehåller denna defekt. När del 13q14 ses isolerat har patienterna den bästa prognosen och de flesta kommer att leva många år, till och med decennier, utan behov av terapi.
Multipelt myelom
Avet-Loiseau, et al. i Journal of Clinical Oncology , använde SNP array karyotyping av 192 multipelt myelom (MM) prover för att identifiera genetiska lesioner associerade med prognos, som sedan validerades i en separat kohort (n = 273). I MM gör bristen på en proliferativ klon konventionell cytogenetik informativ i endast ~30% av fallen. FISH-paneler är användbara i MM, men standardpaneler skulle inte upptäcka flera viktiga genetiska abnormiteter som rapporterats i denna studie. [ citat behövs ]
- Virtuell karyotypning identifierade kromosomavvikelser i 98% av MM-fallen
- del(12p13.31) är en oberoende negativ markör
- amp(5q31.1) är en gynnsam markör
- Den prognostiska effekten av amp(5q31.1) åsidosätter hyperdiploidi och identifierar även patienter som har stor nytta av högdosbehandling.
Array-baserad karyotypning kan inte detektera balanserade translokationer, såsom t(4;14) som ses i ~15% av MM. Därför bör FISH för denna translokation också utföras om man använder SNP-matriser för att upptäcka genomomfattande kopienummerförändringar av prognostisk betydelse i MM. [ citat behövs ]
Medulloblastom
Array-baserad karyotypning av 260 medulloblastom av Pfister S, et al. resulterade i följande kliniska undergrupper baserade på cytogenetiska profiler:
- Dålig prognos: förstärkning av 6q eller förstärkning av MYC eller MYCN
- Intermediär: förstärkning på 17q eller en i(17q) utan förstärkning på 6q eller förstärkning av MYC eller MYCN
- Utmärkt prognos: 6q och 17q balanserad eller 6q deletion
Oligodendrogliom
1p/19q-samdeletionen anses vara en "genetisk signatur" av oligodendrogliom . Allelförluster på 1p och 19q, antingen separat eller kombinerade, är vanligare vid klassiska oligodendrogliom än i antingen astrocytom eller oligoastrocytom. I en studie visade klassiska oligodendrogliom 1p förlust i 35 av 42 (83%) fall, 19q förlust i 28 av 39 (72%), och dessa kombinerades i 27 av 39 (69%) fall; det fanns ingen signifikant skillnad i 1p/19q förlust av heterozygositetsstatus mellan låggradiga och anaplastiska oligodendrogliom. 1p/19q co-deletion har korrelerats med både kemosensitivitet och förbättrad prognos vid oligodendrogliom. De flesta större cancerbehandlingscentra kontrollerar rutinmässigt för deletion av 1p/19q som en del av patologirapporten för oligodendrogliom. Statusen för 1p/19q-loci kan detekteras med FISH eller virtuell karyotypning. Virtuell karyotypning har fördelen av att bedöma hela genomet i en analys, såväl som 1p/19q-loci. Detta möjliggör bedömning av andra nyckelloki i glialtumörer, såsom EGFR- och TP53-kopianummerstatus. [ citat behövs ]
Medan den prognostiska relevansen av 1p och 19q deletioner är väl etablerad för anaplastiska oligodendrogliom och blandade oligoastrocytom, är den prognostiska relevansen av deletionerna för låggradiga gliom mer kontroversiell. När det gäller låggradiga gliom, tyder en färsk studie också på att 1p/19q samdeletion kan vara associerad med en (1;19)(q10;p10) translokation som, liksom den kombinerade 1p/19q deletionen, är associerad med överlägsen total överlevnad och progressionsfri överlevnad hos patienter med låggradig gliom. Oligodendrogliom visar endast sällan mutationer i p53-genen, vilket är i motsats till andra gliom. Epidermal tillväxtfaktorreceptoramplifiering och hel 1p/19q-kodletion är ömsesidigt uteslutande och förutsäger helt olika utfall, med EGFR-amplifiering som förutsäger dålig prognos.
Glioblastom
Yin et al. studerade 55 glioblastom- och 6 GBM-cellinjer med användning av SNP-array-karyotypning. Förvärvad UPD identifierades vid 17p i 13/61 fall. En signifikant förkortad överlevnadstid hittades hos patienter med 13q14 (RB) deletion eller 17p13.1 (p53) deletion/förvärvad UPD. Sammantaget tyder dessa resultat på att denna teknik är en snabb, robust och billig metod för att profilera genomomfattande abnormiteter i GBM. Eftersom SNP array karyotyping kan utföras på paraffininbäddade tumörer, är det ett attraktivt alternativ när tumörceller misslyckas med att växa i kultur för metafas cytogenetik eller när önskan om karyotyping uppstår efter att provet har fixerats med formalin. [ citat behövs ]
Vikten av att upptäcka förvärvad UPD (kopiera neutral LOH) i glioblastom: [ citat behövs ]
- Av patienter med 17p abnormitet var ~50% deletioner och ~50% var aUPD
- Både 17p del och 17p UPD var associerade med sämre resultat.
- 9/13 hade homozygota TP53-mutationer som låg bakom 17p UPD.
Dessutom, i fall med osäker grad av morfologi, kan genomisk profilering hjälpa till vid diagnos.
- Samtidig vinst på 7 och förlust av 10 är i huvudsak patognomonisk för GBM
- EGFR-amplifiering, förlust av PTEN (på 10q) och förlust av p16 (på 9p) förekommer nästan uteslutande i glioblastom och kan tillhandahålla medel för att skilja anaplastiskt astrocytom från glioblastom.
Akut lymfatisk leukemi
Cytogenetik , studien av karakteristiska stora förändringar i cancercellers kromosomer , har alltmer blivit erkänd som en viktig prediktor för utfall vid akut lymfoblastisk leukemi ( ALL). OBS: Balanserade translokationer kan inte detekteras genom array-baserad karyotypning (se Begränsningar nedan).
Vissa cytogenetiska subtyper har sämre prognos än andra. Dessa inkluderar:
- En translokation mellan kromosom 9 och 22, känd som Philadelphia-kromosomen , inträffar hos cirka 20 % av vuxna och 5 % i pediatriska fall av ALL.
- En translokation mellan kromosom 4 och 11 sker i cirka 4 % av fallen och är vanligast hos spädbarn under 12 månader.
- Inte alla translokationer av kromosomer har en sämre prognos. Vissa translokationer är relativt gynnsamma. Till exempel är hyperdiploidi (>50 kromosomer) en bra prognostisk faktor.
- Genomomfattande bedömning av förändringar i antal kopior kan göras genom konventionell cytogenetik eller virtuell karyotypning. Virtuell karyotyping av SNP-matriser kan upptäcka ändringar av kopienummer och LOH-status, medan arrayCGH endast kan upptäcka ändringar av kopienummer. Kopieringsneutral LOH (förvärvad uniparental disomi) har rapporterats vid nyckellokus i ALL, såsom CDKN2A-genen vid 9p, som har prognostisk betydelse. Virtuell karyotyping av SNP-matriser kan lätt detektera kopieringsneutral LOH. Array CGH, FISH och konventionell cytogenetik kan inte detektera kopia neutral LOH.
| Cytogenetisk förändring | Riskkategori |
|---|---|
| Philadelphia kromosom | Dålig prognos |
| t(4;11)(q21;q23) | Dålig prognos |
| t(8;14)(q24.1;q32) | Dålig prognos |
| Komplex karyotyp (mer än fyra avvikelser) | Dålig prognos |
| Låg hypodiploidi eller nära triploidi | Dålig prognos |
| Hög hyperdiploidi | Bra prognos |
| del(9p) | Bra prognos |
Korrelation av prognos med benmärgs cytogenetiska fynd vid akut lymfoblastisk leukemi
| Prognos | Cytogenetiska fynd |
|---|---|
| Gynnsam | Hyperdiploidi > 50; t (12;21) |
| Mellanliggande | Hyperdiolodi 47-50; Normal(diploidi); del (6q); Omarrangemang av 8q24 |
| Ogynnsam | Hypodiploidi-nära haploidi; Nära tetraploidi; del (17p); t (9;22); t (11q23) |
Oklassificerad ALL anses ha en mellanliggande prognos.
Myelodysplastiskt syndrom
Myelodysplastiskt syndrom (MDS) har anmärkningsvärd klinisk, morfologisk och genetisk heterogenitet. Cytogenetik spelar en avgörande roll i Världshälsoorganisationens klassificeringsbaserade International Prognostic Scoring System (IPSS) för MDS.
- God prognos: normal karyotyp, isolerad del(5q), isolerad del(20q), -Y
- Dålig prognos: komplexa avvikelser (dvs >=3 avvikelser), −7 eller del(7q)
- Mellanliggande prognos: alla andra abnormiteter, inklusive trisomi 8 och del(11q)
I en jämförelse av metafascytogenetik, FISH-panel och SNP-arraykaryotypning för MDS, fann man att varje teknik gav ett liknande diagnostiskt utbyte. Ingen enskild metod upptäckte alla defekter, och detektionsfrekvensen förbättrades med ~5% när alla tre metoderna användes.
Förvärvad UPD, som inte kan detekteras av FISH eller cytogenetik, har rapporterats vid flera nyckellokus i MDS med användning av SNP-array-karyotypning, inklusive deletion av 7/7q.
Myeloproliferativa neoplasmer/myeloproliferativa störningar
Philadelphia-kromosomnegativa myeloproliferativa neoplasmer (MPN) inklusive polycytemia vera, essentiell trombocytemi och primär myelofibros visar en inneboende tendens till omvandling till leukemi (MPN-blastfas), som åtföljs av förvärv av ytterligare genomiska lesioner. I en studie av 159 fall kunde SNP-arrayanalys fånga praktiskt taget alla cytogenetiska abnormiteter och avslöja ytterligare lesioner med potentiellt viktiga kliniska implikationer. [ citat behövs ]
- Antalet genomiska förändringar var mer än 2 till 3 gånger större i blastfasen som i den kroniska fasen av sjukdomen.
- Deletion av 17p (TP53) var signifikant associerad med tidigare exponering för hydroxiurea såväl som en komplex karyotyp i prover med MPN-blastkris. Både deletion och 17p kopia neutral LOH, var associerade med en komplex karyotyp, en dålig prognostisk markör i myeloida maligniteter. Kopieringsneutral LOH (förvärvad UPD) kan lätt detekteras av SNP-arraykaryotyp, men inte av cytogenetik, FISH eller array-CGH.
- Blastfaspatienter med förlust av kromosomalt material på 7q visade dålig överlevnad. Förlust av 7q är känd för att vara prediktiv för snabb progression och dåligt svar vid AML-behandling. MPN-blastfaspatienter med cytogenetiskt odetekterbar 7q kopia neutral-LOH hade jämförbar överlevnadsgrad som de med 7/7q i sina leukemiceller.
- 9p kopia neutral-LOH med homozygot JAK2-mutation var också kopplad till ett sämre resultat i MPN-blastkris i jämförelse med patienter med antingen heterozygot JAK2V617F eller vildtyp JAK2. I motsats till LOH på 17p var den prognostiska effekten av 9pCNN-LOH oberoende av etablerade riskfaktorer som 7/7q, 5q eller komplex karyotyp.
Kolorektal cancer
Identifiering av biomarkörer vid kolorektal cancer är särskilt viktig för patienter med sjukdomsstadium II, där mindre än 20 % har tumörrecidiv. 18q LOH är en etablerad biomarkör associerad med hög risk för tumörrecidiv vid stadium II tjocktarmscancer. Figur 7 visar en SNP-arraykaryotyp av ett kolorektalt karcinom (vy över hela genomet).
Kolorektalcancer klassificeras i specifika tumörfenotyper baserat på molekylära profiler som kan integreras med resultaten av andra kompletterande tester, såsom mikrosatellitinstabilitetstestning, IHC och KRAS-mutationsstatus:
- Kromosomal instabilitet (CIN) som har allelisk obalans vid ett antal kromosomala loci, inklusive 5q, 8p, 17p och 18q (fig 7).
- Mikrosatellitinstabilitet (MSI) som tenderar att ha diploida karyotyper.
Maligna rhabdoida tumörer
Maligna rhabdoidtumörer är sällsynta, mycket aggressiva neoplasmer som oftast förekommer hos spädbarn och småbarn. På grund av deras heterogena histologiska egenskaper kan diagnos ofta vara svår och felklassificeringar kan förekomma. I dessa tumörer fungerar INI1-genen (SMARCB1) på kromosom 22q som en klassisk tumörsuppressorgen. Inaktivering av INI1 kan ske via deletion, mutation eller förvärvad UPD.
I en nyligen genomförd studie identifierade SNP array karyotyping deletioner eller LOH av 22q i 49/51 rhabdoida tumörer. Av dessa var 14 kopieneutral LOH (eller förvärvad UPD), som kan detekteras genom SNP-arraykaryotypning, men inte av FISH, cytogenetik eller arrayCGH. MLPA upptäckte en enda exon homozygot deletion i ett prov som var under upplösningen för SNP-matrisen. [ citat behövs ]
SNP array karyotyping kan användas för att skilja till exempel ett medulloblastom med en isokromosom 17q från en primär rhabdoidtumör med förlust av 22q11.2. När så anges kan molekylär analys av INI1 med användning av MLPA och direkt sekvensering sedan användas. När de tumörrelaterade förändringarna har hittats kan en analys av könscells-DNA från patienten och föräldrarna göras för att utesluta en ärftlig eller de novo könslinjemutation eller deletion av INI1, så att lämpliga riskbedömningar av återfall kan göras.
Uveal melanom
Den viktigaste genetiska förändringen som är förknippad med dålig prognos vid uvealt melanom är förlust av en hel kopia av kromosom 3 ( Monosomy 3), som är starkt korrelerad med metastatisk spridning. Vinster på kromosom 6 och 8 används ofta för att förfina det prediktiva värdet av Monosomy 3-skärmen, med en vinst på 6p som indikerar en bättre prognos och en vinst på 8q indikerar en sämre prognos i disomi 3-tumörer. I sällsynta fall kan monosomi 3-tumörer duplicera den återstående kopian av kromosomen för att återgå till ett disomiskt tillstånd som kallas isodisomi . Isodisomi 3 är prognostiskt ekvivalent med monosomi 3, och båda kan detekteras genom tester för kromosom 3- förlust av heterozygositet .
Begränsningar
Till skillnad från karyotyper erhållna från konventionell cytogenetik, rekonstrueras virtuella karyotyper av datorprogram med hjälp av signaler erhållna från stört DNA. I huvudsak kommer datorprogrammet att korrigera translokationer när det radar upp signalerna i kromosomordning. Därför kan virtuella karyotyper inte upptäcka balanserade translokationer och inversioner . De kan också bara upptäcka genetiska avvikelser i regioner av genomet som representeras av sonder på arrayen. Dessutom genererar virtuella karyotyper ett relativt antal kopior normaliserat mot ett diploid genom, så tetraploida genom kommer att kondenseras till ett diploid utrymme om inte renormalisering utförs. Renormalisering kräver en extra cellbaserad analys, såsom FISH, om man använder arrayCGH. För karyotyper erhållna från SNP-baserade arrayer kan tetraploidi ofta härledas från upprätthållandet av heterozygositet inom en region med uppenbar kopieantalförlust. Lågnivåmosaicism eller små subkloner kanske inte detekteras av virtuella karyotyper eftersom närvaron av normala celler i provet kommer att dämpa signalen från den onormala klonen. Den exakta punkten för fel, i termer av den minimala andelen neoplastiska celler, kommer att bero på den speciella plattformen och de algoritmer som används. Många program för analys av kopior som används för att generera array-baserade karyotyper kommer att vackla med mindre än 25–30 % tumör/onormala celler i provet. I onkologiska tillämpningar kan dock denna begränsning minimeras genom tumörberikande strategier och programvara optimerad för användning med onkologiprover. Analysalgoritmerna utvecklas snabbt, och vissa är till och med utformade för att trivas med "normal klonkontamination", så det förväntas att denna begränsning kommer att fortsätta att försvinna.
Se även
- DECIPHER , en databas över kromosomal obalans och fenotyp hos människor med hjälp av Ensembl Resources