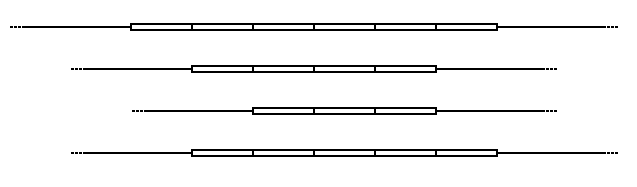
Variabelt antal tandemupprepning
En tandemupprepning med variabelt antal (eller VNTR ) är en plats i ett genom där en kort nukleotidsekvens är organiserad som en tandemupprepning . Dessa kan hittas på många kromosomer och visar ofta variationer i längd (antal upprepningar) mellan individer. Varje variant fungerar som en ärvd allel , vilket gör att de kan användas för personlig eller förälders identifiering. Deras analys är användbar inom genetik och biologiforskning , kriminalteknik och DNA-fingeravtryck .
Struktur och allelvariation
I schemat ovan representerar de rektangulära blocken var och en av de upprepade DNA-sekvenserna vid en speciell VNTR-plats. Upprepningarna är i tandem – dvs de är samlade och orienterade i samma riktning. Individuella upprepningar kan tas bort från (eller läggas till) VNTR via rekombinations- eller replikationsfel , vilket leder till alleler med olika antal upprepningar. Flankerande regioner är segment av icke-repetitiv sekvens (visas här som tunna linjer), vilket gör att VNTR-blocken kan extraheras med restriktionsenzymer och analyseras med RFLP , eller amplifieras med polymeraskedjereaktionstekniken (PCR) och deras storlek bestäms med gelelektrofores .
Används i genetisk analys
VNTRs var en viktig källa för genetiska RFLP-markörer som användes vid länkanalys (kartläggning) av diploida genom. Nu när många genom har sekvenserats har VNTR:er blivit avgörande för kriminaltekniska brottsutredningar, via DNA-fingeravtryck och CODIS -databasen. När de avlägsnas från omgivande DNA med PCR- eller RFLP-metoderna, och deras storlek bestäms genom gelelektrofores eller Southern blotting , producerar de ett mönster av band som är unika för varje individ. När de testas med en grupp oberoende VNTR-markörer är sannolikheten för att två obesläktade individer har samma allelmönster extremt låg. VNTR-analys används också för att studera genetisk mångfald och avelsmönster i populationer av vilda eller tama djur. Som sådan kan VNTRs användas för att särskilja stammar av bakteriella patogener. I detta mikrobiella kriminaltekniska sammanhang kallas sådana analyser vanligtvis Multiple Loci VNTR Analysis eller MLVA .
Arv
Vid analys av VNTR-data kan två grundläggande genetiska principer användas:
- Identitetsmatchning – båda VNTR-allelerna från en specifik plats måste matcha. Om två prover är från samma individ måste de visa samma allelmönster.
- Arvsmatchning – VNTR-allelerna måste följa reglerna för arv. Vid matchning av en individ med sina föräldrar eller barn måste en person ha en allel som matchar en från varje förälder. Om förhållandet är mer avlägset, till exempel en mor- eller farförälder eller ett syskon, måste matchningar överensstämma med graden av släktskap.
Förhållande till andra typer av repetitivt DNA
Repetitivt DNA , som representerar över 40 % av det mänskliga genomet, är ordnat i en förvirrande uppsättning mönster. Upprepningar identifierades först genom extraktion av satellit-DNA , som inte avslöjar hur de är organiserade. Användningen av restriktionsenzymer visade att vissa upprepade block var insprängda i genomet. DNA-sekvensering visade senare att andra upprepningar är klustrade på specifika platser, där tandemupprepningar är vanligare än inverterade upprepningar (vilket kan störa DNA-replikationen). VNTRs är klassen av klustrade tandemupprepningar som uppvisar allelisk variation i sina längder.
Klasser

VNTR:er är en typ av minisatelliter där storleken på den upprepade sekvensen i allmänhet är tio till hundra baspar. Minisatelliter är en typ av DNA- tandemupprepningssekvens , vilket betyder att sekvenserna upprepas en efter en utan andra sekvenser eller nukleotider mellan dem. Minisatelliter kännetecknas av en upprepad sekvens på cirka tio till hundra nukleotider, och antalet gånger som sekvensen upprepas varierar från cirka fem till femtio gånger. Sekvenserna för minisatelliter är större än de för mikrosatelliter , i vilka den upprepade sekvensen i allmänhet är 1 till 6 nukleotider. De två typerna av upprepade sekvenser är båda tandem men specificeras av längden på den upprepade sekvensen. VNTR:er anses därför, eftersom de har upprepade sekvenser på tio till hundra nukleotider där varje upprepning är exakt likadan, som minisatelliter. Men medan alla VNTR:er är minisatelliter, är inte alla minisatelliter VNTR:er. VNTR:er kan variera i antal upprepningar från individ till individ, som där vissa icke-VNTR-minisatelliter har upprepade sekvenser som upprepar samma antal gånger i alla individer som innehåller tandemupprepningarna i deras genom.
Se även
Programvara för MLVA-skrivning
- BioNumerics (med hjälp av BioNumerics MIRU-VNTR-plugin som är speciellt utvecklad för att köra en populär MLVA-analys som genotypar Mycobacterium tuberculosis -stammar)
externa länkar
- Exempel:
- VNTRs – info och animerat exempel
- Databaser:
- Sökverktyg :
- TAPO: En kombinerad metod för identifiering av tandemupprepningar i proteinstrukturer
- Tandem Repeats Finder
- Mreps
- STJÄRNA
- TRED
- TandemSWAN
- Mikrosatellitupprepningssökare
- JSTRING – Java-sökning efter tandemupprepningar i genom
- Phobos – ett tandemupprepningssökverktyg för perfekta och ofullkomliga upprepningar – den maximala mönsterstorleken beror bara på beräkningskraft
- Variable+Number+of+Tandem+Repeats på US National Library of Medicine Medical Subject Headings (MeSH)