Genomredigering utanför målet
Genomredigering utanför målet hänvisar till ospecifika och oavsiktliga genetiska modifieringar som kan uppstå genom användning av konstruerade nukleasteknologier såsom: klustrade, regelbundet mellanrum, korta palindromiska upprepningar ( CRISPR )- Cas9 , transkriptionsaktivatorliknande effektornukleaser ( TALEN ), meganukleaser . och zinkfingernukleaser (ZFN). Dessa verktyg använder olika mekanismer för att binda en förutbestämd sekvens av DNA ("mål"), som de klyver (eller "klipper"), vilket skapar ett dubbelsträngat kromosomavbrott (DSB) som framkallar cellens DNA- reparationsmekanismer (icke-homolog ände). sammanfogning ( NHEJ ) och homolog rekombination ( HR )) och leder till platsspecifika modifieringar. Om dessa komplex inte binder till målet, ofta ett resultat av homologa sekvenser och/eller missmatchningstolerans, kommer de att klyva off-target DSB och orsaka ospecifika genetiska modifieringar. Specifikt består effekter utanför målet av oavsiktliga punktmutationer , deletioner , inversioner av insättningar och translokationer .
Designer nukleassystem som CRISPR-cas9 blir allt populärare forskningsverktyg som ett resultat av deras enkelhet, skalbarhet och överkomliga priser. Med detta sagt är genetiska modifieringar utanför målet frekventa och kan förändra funktionen hos annars intakta gener. Flera studier med tidiga CRISPR-cas9-medel fann att mer än 50 % av RNA-styrda endonukleas-inducerade mutationer inte inträffade på målet. Cas9 " DNA-sekvensen. Emellertid kan målsekvensbindning tolerera felmatchningar upp till flera baspar, vilket innebär att det ofta finns tusentals möjliga bindningsställen som utgör flera experimentella och säkerhetsproblem. Inom forskningssfären kan effekter utanför målet förväxla variabler i biologiska studier som leder till potentiellt vilseledande och icke-reproducerbara resultat. Inom den kliniska sfären är de största problemen kring störningen av viktiga kodande regioner som leder till genotoxiska effekter som cancer. Följaktligen är förbättringen av specificiteten hos genomredigeringsverktyg och upptäckten av effekter utanför målet snabbt framskridande forskningsområden. Sådan forskning inbegriper designers nukleasutveckling och upptäckt, beräkningsprogram för förutsägelse och databaser och sekvensering med hög genomströmning för att minska och förutse mutationsförekomst. Många designade nukleasverktyg är fortfarande i sin relativa linda och när deras molekylära egenskaper och in vivo -beteenden blir bättre förstådda kommer de att bli allt mer exakta och förutsägbara.
Mekanismer
CRISPR-Cas9-systemet fungerar som det adaptiva immunsystemet i bakterier och arkéer. När ett virus infekterar bakterierna, införlivar detta system segment av det virala DNA:t i bakteriegenomet. Vid en andra invasion riktar transkript från dessa sekvenser en nukleasaktivitet till dess komplementära sekvens i det invaderande viruset för att förstöra det.
För att extrapolera denna metod till eukaryoter för att utveckla en genredigeringsmetod krävs ett Cas9-protein, ett igenkänningssekvens-RNA och ett transaktiverande RNA. Fusionen av både igenkänningssekvensspecificiteten CRISPR RNA (crRNA) och transaktiverande RNA (tracrRNA) används vanligen i experiment och kallas ett enda guide-RNA (sgRNA). Den utför båda funktionerna: de första 20 nukleotiderna i sgRNA:t är komplementära till DNA-målsekvensen (cr-funktion), medan nukleotiderna som följer är en del av ett protospacer-angränsande motiv (PAM; tracr-funktion).
Off-targeting nukleasbindning härrör från en partiell men tillräcklig matchning till målsekvensen. Bindningsmekanismer utanför målet kan grupperas i två huvudformer: basfelmatchningstolerans och utbuktning.
Basens oöverensstämmelsetolerans
Även om Cas9-specificiteten tros kontrolleras av 20nt sgRNA och PAM, är mutationer utanför målet fortfarande vanliga och kan förekomma med så många som 3-5 baspar felmatchningar (av 20) mellan sgRNA och mål-DNA-sekvensen. Vidare kan sekundära sgRNA-strukturer också påverka klyvning av ställen på och utanför målet. Som nämnts ovan består sgRNA av en sekvens (~20nt) som är komplementär till målsekvenserna och denna följs av en PAM-sekvens som aktiverar endonukleasaktiviteten . Medan det visades att 10-12 nt intill PAM (kallad "frösekvens") var tillräckligt för Cas9-specificitet, Wu et al. visade att i en katalytiskt död Cas9 krävs endast 1-5 baspar av frösekvens för specificitet. Detta bevisades senare också av andra studier. Cas9-proteinbindningen påverkas ytterligare av ett antal mekanismer:
- Frösekvensen bestämmer frekvensen av ett frö plus PAM i genomet och kontrollerar den effektiva koncentrationen av Cas9 sgRNA-komplex.
- Uracilrika frön har sannolikt låga sgRNA-nivåer och ökar specificiteten eftersom flera uraciler i sekvensen kan introducera avslutning av sgRNA- transkriptionen .
- Felmatchningar i 5'-änden av crRNA:t tolereras mer eftersom det viktiga stället skulle ligga intill PAM-matrisen. Enkla och dubbla felmatchningar tolereras också baserat på hur den placeras.
- I en färsk studie, Ren et al. observerade en koppling mellan mutageneseffektivitet och GC-innehåll i sgRNA. Minst 4-6 bp intill PAM krävs för en bra redigering.
- Medan man väljer ett gRNA föredras guanin framför cytosin som den första basen i fröet intill PAM, cytosin som den första i 5' och adenin i mitten av sekvensen. Denna design är baserad på stabilitet kopplad till bildandet av G-quadruplex .
- En ChIP utfördes av Kim et al. visar att tillägg av en renad Cas9 tillsammans med sgRNA orsakade låga off-måleffekter, vilket betyder att det finns fler faktorer som orsakar dessa effekter.
Det är viktigt att notera att DNA-metylering av CpG-ställen minskar effektiviteten av bindning av Cas9 och andra faktorer i celler. Därför finns det en epigenetisk länk som kommer att utforskas mer för framtiden för epigenomredigering.
Variationer inom PAM-sekvensen kan också påverka sgRNA-aktivitet, vilket i sin tur påverkar själva sgRNA. I vanliga Cas9-system är PAM-motivet 5' NGG 3', där N representerar någon av de fyra DNA-nukleotiderna. Kravet på PAM-sekvensen kan orsaka specificitetsproblem eftersom vissa regioner kommer att ha en tillgänglig målsekvens för att göra en önskad genetisk modifiering. En rapport angav att 99,96% av platser som tidigare antagits vara unika Cas9-mål i mänskliga exoner kan ha potentiella off-target-effekter innehållande NAG eller NGG PAM och en enda basfelmatchning i frösekvensen.
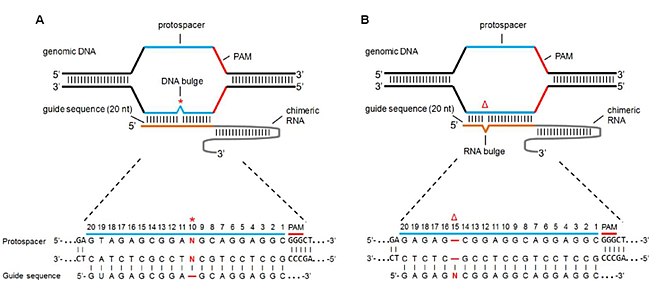
Bulge missmatch
Både ställen utanför mål med saknade baser (eller deletioner) och ställen utanför mål med extra baser (eller insättningar) som kallas RNA-bulge respektive DNA-bulge, har effekter på Cas9-specificitet och klyvningsaktivitet. Lin et al. efterliknade dessa utbuktningar genom att lägga till och ta bort baser från sgRNA-sekvensen så att en basdeletion i sgRNA:t skulle ge en RNA-utbuktning och en basinsättning skulle ge en DNA-utbuktning. Genom att studera mutationshastigheterna via NHEJ kom de fram till följande resultat:
- Vid rena DNA-utbuktningar tolererades mutationerna väl (dvs. Cas9-klyvningsaktivitet var fortfarande utbredd). Regionerna med utbuktningstolerans inkluderade sju baser från PAM och 5'- och 3'-ändarna av frösekvensen. Detta resulterade i liknande eller något högre (i vissa fall) mutationer jämfört med noll utbuktningar.
- I fallet med rena RNA-utbuktningar inducerades högre Cas9-aktivitet vid många positioner jämfört med DNA-utbuktningar. Denna egenskap tillskrevs det faktum att RNA är mer flexibelt än DNA och därför har en mindre bindningsstraff med RNA-utbuktning, vilket resulterar i en högre tolerans och högre off target-mutationer.
- Högre GC-innehåll i sgRNA-sekvensen resulterade i en högre tolerans och därmed en högre mutationshastighet utanför målet.
- Utbuktningar på 2bp-5bp var slående mer toleranta och mutationsinducerande än en enda 2bp utbuktning.
Förbättringar
Metoder för att öka specificiteten
Det allmänt använda Streptococcus pyogenes Cas9 (SpCas9) nukleaset är effektivt, men det inducerar oönskade mutationer utanför målet vid höga frekvenser. Flera ingenjörs- och screeningmetoder har beskrivits i ett försök att minska genomomfattande mutationer utanför målet inklusive nukleasmutation, protospacer adjacent motiv (PAM) sekvensmodifiering, guide RNA (gRNA) trunkering och ny nukleasupptäckt. Till exempel, 2013, Fu et al. rapporterade att genom att trunkera gRNA från <20 bp i längd till 17 eller 18 bp ökade målspecificiteten för nukleaset upp till 5 000 gånger och felparningsförekomster över 3 baser inträffade sällan, om någonsin.
Cas9 nickases
SpCas9-nukleaset kan också muteras på en mängd olika sätt för att förbättra specificitet och kontroll. Nukleasdomäner kan muteras oberoende av varandra till vad som kallas Cas9-nickaser. Dessa nukleaser har en aktiv och inaktiv nukleasdomän som resulterar i ett komplex som utför enkelsträngsklyvning. Cas9 nickaser kan användas i tandem (känd som parade nickaser), som utför två enkelsträngs "snitt" på alternativa strängar. Med denna strategi måste båda Cas9-nickaserna samlokalisera, binda och klyva sitt mål, vilket drastiskt minskar sannolikheten för indelar utanför målet . Dessutom har DSB:erna som klyvs av parade nickaser långa överhäng istället för trubbiga ändar som ger förbättrad kontroll av riktade insättningar.
Fok1-dCas9 och dimeriseringsnukleaser
Eftersom monomera nukleaser ofta involverar höga nivåer av effekter utanför målet, är dimerisering en attraktiv strategi. I ett dimersystem måste båda nukleaserna binda till sina individuella mål eller "halvplatser" och sedan interagera och dimerisera för att initiera klyvning vilket kraftigt minskar sannolikheten för effekter utanför målet. En metod som innehåller tillförlitligheten hos dimeriseringsberoende FokI -nukleasdomäner, som används i ZFNs och TALENs, med enkelheten hos CRISPR-cas9 har utvecklats. FokI-nukleaset hittades ursprungligen i Flavobacterium okeanokoites och kommer endast att klyva DNA vid dimeriseringsaktivering. I grund och botten fusionerade forskarna detta nukleas till ett CRISPR-komplex med ett inaktivt Cas9-nukleas (Fok1-dCas9). gRNA:t dirigerar CRISPR-komplexet till målstället men "snittet" görs av dimeriserat Fok1. Det uppskattas att Fok1-dCas9-strategin minskar detekterbara effekter utanför målet med 10 000 gånger, vilket gör den effektiv för applikationer som kräver mycket exakt och specifik genomredigering.
Nukleasmutation
Förutom ett gRNA-mål kräver Cas9 bindning till en specifik 2-6 nukleotidsekvens PAM. I vanliga SpCas9-system är PAM-motivet 5' NGG 3', där N representerar någon av de fyra DNA-nukleotiderna. Kravet på PAM-sekvensen kan orsaka specificitetsbegränsningar eftersom vissa regioner inte kommer att ha en tillgänglig målsekvens för att göra en önskad genetisk modifiering. PAM-sekvensen kan redigeras till icke-kanoniska NAG- och NGA-motiv som inte bara förbättrar specificiteten utan också minskar effekter utanför målet. En D1135E-mutant verkar förändra PAM-specificiteter. D1135E-mutanten minskar effekter utanför målet och ökar specificiteten hos SpCas9. En ytterligare variant, SpCas9-HF1, resulterar också i gynnsamma förbättringar av Cas9 specificitet. Flera kombinationer av substitutioner kända för att bilda icke-specifika DNA-kontakter (N497A, R661A, Q695A och Q926A) har identifierats. En fyrdubbel substitution av dessa rester (senare kallad SpCas9-HF1), har extremt låga nivåer av effekter utanför målet, vilket detekteras av GUIDE-seq experiment. Varianter som SpCas9-HF1 och D1135E och andra liknande kan kombineras, testas och enkelt läggas till befintliga SpCas9-vektorer för att minska frekvensen av mutationer utanför målet. Dessutom kan många av de tekniska strategierna som anges ovan kombineras för att skapa allt mer robusta och tillförlitliga RNA-styrda nukleasredigeringsverktyg. Riktad evolution kan också användas för att minska nukleasaktivitet på särskilda målsekvenser, vilket leder till varianter som SpartaCas (innehållande mutationer D23A, T67L, Y128V och D1251G i förhållande till vildtyp SpCas9).
CRISPRi och CRISPRa
CRISPR-interferens ( CRISPRi ) och CRISPR-aktivering ( CRISPRa ) har också utvecklats. Dessa system kan exakt ändra gentranskription på DNA-nivå utan att orsaka irreversibla genetiska förändringar. Genom att direkt verka på DNA är de i allmänhet mer specifika och förutsägbara jämfört med RNAi . Även om CRISPRi/a inte kan ersätta genomredigering i alla experiment, kan de fungera som effektiva alternativ i vissa fall. CRISPRi och CRISPRa använder ett deaktiverat Cas9 (dCas9) enzym som inte kan skära DNA, men som kan leverera transkriptionella aktivatorer och repressorer för att modulera önskat genuttryck med hög precision. För närvarande är effekterna utanför målet av CRISPRi minimala och visar en minskad respons och känslighet för enbasfelmatchningar. Viktigt är att när ospecifika effekter oundvikligen uppstår är de reversibla, tidsberoende och mindre skadliga än DNA-redigering, vilket gör dem till effektiva alternativ som kan begränsa bördan utanför målet när det är möjligt. CRISPR-cas13b, med hjälp av ett typ IV CRISPR-Cas-system (i motsats till den vanliga typ II) kan rikta in sig på och redigera specifika RNA-sekvenser. En sådan RNA-redigeringsplattform har förmågan att specifikt redigera mRNA, och därför proteintranslation, utan att förändra DNA:t. Den representerar en lovande teknologi som om den lyckades skulle minska bördan av irreversibla mutationer utanför målet.
Upptäckt
Även om man kan vidta försiktiga åtgärder för att undvika off-target mutationer, och även om man lyckas, måste en bekräftande screening göras för att screena för oavsiktliga mutationer. För närvarande finns det gott om partiska och opartiska metoder för en sådan screening och endast två in vitro- metoder. Alla dessa listas nedan:
Målinriktad, exom- och helgenomsekvensering
Vid normal riktad sekvensering kommer den partiska metoden att ge resultat endast för det avsedda området för fångst, vilket hindrar sökningen eftersom inga oväntade mutationer kommer upp på skärmen. Även om det är enkelt och billigt, blir det tidskrävande och dyrt när fler målwebbplatser läggs till. Exomsekvensering använder exomfångning för att förvärva de proteinkodande regionerna i genomet. Den är opartisk, men den kommer inte att ge målmutationer i den icke-kodande regionen av genomet. Vid sekvensering av hela genomet screenas hela genomet för off-målmutationer. För närvarande är denna metod dyr och liksom exomsekvensering kräver hela genomet också ett referensgenom för att dra slutsatser.
VÄLSIGNA
BLESS är det enklaste sättet att upptäcka och kvantifiera mutationer utanför målet genom att screena för DSB i genomet. Denna metod förlitar sig på direkt in situ -avbrott märkningsanrikning på streptavidin. BLESS utvecklades 2013 och utförs genom att ligera DSB-ändarna med biotin, dvs biotinylering. Detta följs av separation/uppsamling av nämnda ligerade ändar med användning av streptavidin. En länkad sekvens läggs till de biotinylerade sekvenserna och denna slutliga blandning sekvenseras sedan för att ge positionen för mutationen utanför målet. Eftersom BLESS är opartisk till sin natur, ger BLESS information om mutationsstället i genomet snarare än de proteiner som är involverade eller associerade med DSB. Emellertid kan BLESS endast upptäcka mutationer vid tidpunkten för experimentet och inte de som bildades tidigare och reparerades.
LAM-HTGTS
Linear Amplification Mediated - High Throughput Genome Wide Translocation Sequencing, eller LAM-HTGTS, är en metod utvecklad för att spåra translokationshändelser orsakade av sammankoppling mellan DSB:er. Utvecklad för att detektera off-target mutationer från TALEN och CRISPR-Cas9, är denna teknik baserad på DNA-reparation genom ändsammanfogning i DSB. När nukleaset väl tillsätts fortsätter det att producera mutationer på och utanför målet. Tillsammans med detta finns en betessekvens som också klyvs. Därför, om en annan DSB uppträder på en annan kromosom än betessekvenskromosomen, sammanfogas de båda vilket leder till en translokation. Eftersom betessekvensen är känd amplifieras denna translokerade sekvens med primrar. Om det inte finns någon translokation finns det ett restriktionsställe inom vilket klyvs för att förhindra amplifiering av endast betessekvensen. Det amplifierade DNA:t sekvenseras sedan för att studera stora genomiska omarrangemang på grund av mutationer utanför målet. En nackdel är att den förlitar sig på samtidig närvaro av bete och en annan DSB.
GUIDE-Sekv
Ett annat tillvägagångssätt för att hitta mutationer utanför målet på grund av nukleasaktivitet är GUIDE-Seq- metoden. GUIDE-seq eller Genome Wide Unbiased Identification of DSBs Enabled by Sequencing är baserad på inkorporeringen av dubbelsträngade oligodeoxinukleotider (dsODN) i DSB:er via NHEJ. Dess amplifiering följs av sekvensering. Eftersom två primrar kommer att användas för att sekvensera dsODNs kommer regionerna som flankerar DSB tillsammans med DSB att amplifieras. På så sätt möjliggör kartläggning av mutationen utanför målet. Denna teknik har använts för att identifiera alla tidigare kända platser utanför målet såväl som nya med så låga frekvenser som 0,03 %. Precis som BLESS kan dock GUIDE-seq endast upptäcka DSB:er som finns vid studietillfället.
Digenome-Seq
En av de nuvarande in vitro- metoderna, Digenome-Seq använder Cas9s egenskap att klyva genomet för att få en opartisk profil av hela genomet. I denna metod läggs Cas9 till gDNA och eftereffekterna studeras med hjälp av sekvensering med hög genomströmning. Eftersom fragmenten bildas på grund av samma nukleas, kan ändarna av dessa fragment kartläggas i linje. Två stora fördelar är att den kan användas för att studera upp till 10 gRNA på en gång och kan identifiera mål till så låga frekvenser som 0,01%. Den största fördelen är dock att denna metod är in vitro , dvs. DSB:erna som introducerats av Cas9 kommer inte att bearbetas av DNA-reparationsmaskineriet (till skillnad från BLESS och GUIDE-seq) och kommer därför att inkludera alla möjliga off-target mutanter. Det kan dock leda till ett stort antal falska positiva resultat också.
CIRKEL-Sekv
Det senaste tillägget till in vitro -metoderna för att detektera off-target mutationer är CIRCLE-seq. Licensierad av Beacon genomics (tillsammans med GUIDE-seq), syftar CIRCLE-seq till att ta bort nackdelarna med Digenome-seq såsom behovet av en stor provstorlek och läsdjup (~400 miljoner läsningar) och den höga bakgrunden som gör identifiering av lågfrekventa klyvningshändelser svårare. Den antar en restriktionsenzymoberoende strategi för att skapa och välja omvandling av slumpmässigt klippt DNA. Vid klyvning bildar mål-DNA en stamslinga till vilken adaptrar kan läggas till för sekvensering. Även om detta visade sig möjligt, gav den andra möjligheten en dubbelt hög skillnad i upptäckt/. I det andra fallet är sekvensen klyvd med Cas9 och när den klyvs igen på halva platsen finns ett cirkulärt snitt tillgängligt (vilket är anledningen till namnet CIRCLE-seq). Nästan alla platser som identifierats genom cirkularisering innehåller både linjära detekterade platser och nyare, vilket tyder på att CIRCLE-seq inte förargar mellan avbrott och även erhåller starka lågfrekventa avbrott. Det hjälper vidare att sekvensera brytstället från båda sidor av klyvningen jämfört med andra metoder som endast har en avläst sida.
Streckkodade bibliotek av mål
Nukleaser såsom Cas9 kan också utmanas in vitro av randomiserade bibliotek av mål. Adapterligering för att kvantifiera kluvna och oklyvda biblioteksmedlemmar möjliggör opartisk mätning av ett nukleas specificitetsprofil. Mätning av klyvning av streckkodade målbibliotek (BLT) med SpCas9 indikerade att specificitetsprofiler var guidespecifika och beror på guidesekvensen såväl som själva nukleaset. Opartiska specificitetsprofiler baserade på varje särskilt Cas9-gRNA-komplex kan sedan användas för att bygga guidespecifika prediktiva modeller för in vitro- klyvning.
Betydelse
Genterapi
För att genredigeringstekniker ska kunna ta steget mot säker och utbredd användning på kliniken, måste graden av modifiering utanför målet göras föråldrad. Säkerheten för genterapibehandling är ytterst oroande, särskilt under kliniska prövningar när modifieringar utanför målet kan blockera den fortsatta utvecklingen av en kandidatprodukt. Det kanske mest kända exemplet på modern genterapi är CAR-T-terapi, som används för behandling av B-cellslymfom . För att begränsa hastigheten för klyvning utanför målet använder terapin en mycket specifik och finjusterad TALEN, som har visat sig ha liten eller ingen bakgrundsinteraktion utanför målet. CAR-T- immunterapi är ett ex vivo- förfarande, vilket innebär att patientens immunceller (i detta fall T-celler ) extraheras och redigeras med hjälp av designernukleaser. Medan TALEN-systemutveckling är dyr och tidskrävande, har forskning och tekniska modifieringar drastiskt begränsat deras frekvens av interaktion utanför målet. Patienter som får behandlingen övervakas dock fortfarande ofta och kommer att göra det under de kommande 15 åren så att effekter utanför målet och immunogena svar kan analyseras och tas i beaktande när nya genterapier förs in i klinisk prövning.
CCR5 ZFN-modifierade autologa T-hjälparförsök
En klinisk fas I/II- studie inkluderade 12 patienter med förvärvat immunbristsyndrom (AIDS) för att testa säkerheten och effektiviteten av att administrera ZFN-modifierade autologa T-hjälparceller. Genom riktade raderingar inaktiverar den anpassade ZFN-genen CC kemokinreceptor 5 ( CCR5 ), som kodar för en co-receptor som används av HIV-viruset för att komma in i cellen. Som ett resultat av den höga graden av sekvenshomologi mellan CC-kemokinreceptorer klyver denna ZFN också CCR2 , vilket leder till bort-mål-15 kb deletioner och genomiska omarrangemang. Effekterna av dessa CCR2- modifieringar är fortfarande inte kända, och hittills har det inte rapporterats några biverkningar. Men CCR2 är känt för att ha många kritiska roller i neurala och metabola system.
Gene Drives
Konstruerade gendrivningar med CRISPR-cas9 testas för närvarande och har föreslagits som strategier för att eliminera invasiva arter och sjukdomsvektorer. Genom att genetiskt modifiera en organism för att uttrycka ett endogent sekvensspecifikt endonukleas kan ett mål (såsom en fertilitetsgen) klyvas på den motsatta kromosomen. En DSB vid målet leder till homolog reparation som effektivt gör organismen homozygot för den önskade målsekvensen. Denna strategi, känd som en målsökning, kan undertrycka en population genom att påverka en kritisk gen eller inducera recessiv sterilitet. Men om ett sådant system släpptes ut i naturen skulle CRISPR-cas9-systemet förbli fungerande på obestämd tid. Med varje efterföljande generation skulle mutationer utanför målet bli allt mer sannolika och effekterna av dessa mutationer på en art skulle vara stokastiska. Off-target-mutationer kan inaktivera de undertryckande egenskaperna hos en gendrivning samtidigt som endonukleasuttrycket bibehålls. I en sådan situation skulle det finnas en ökad risk för genflöde mellan målarten och andra arter som sannolikt skulle leda till oönskade resultat.
Kontrovers
Den ökade användningen av genomredigering och dess eventuella översättning till klinisk användning har framkallat kontroverser kring teknologiernas verkliga börda utanför målet.
Schäfer et al. 2017
Den 30 maj 2017 publicerades en tvåsidig korrespondensartikel i Nature Methods som rapporterade ett ovanligt stort antal SNV och indels utanför målet efter sekvensering av möss som tidigare var involverade i ett genreparationsexperiment in vivo . Det tidigare experimentet, avslutat av samma grupp, återställde framgångsrikt synen av blinda musstam ( rd1 ) genom att korrigera Y347X-mutationen i Pde6b -genen med ett CRISPR-cas9-system. Efter att ha slutfört experimentet sekvenserades två genetiskt korrigerade möss av hela genomet och jämfördes med kontroll- och kända genom av musstammar. Mer än 1 600 SNVs och 128 indels upptäcktes, varav 1 397 SNVs och 117 indels delades mellan de två redigerade mössen, vilket tyder på att effekterna utanför målet inte var slumpmässiga. Algoritmer som försökte förutsäga platsen för dessa mutationer utanför målet misslyckades för en överväldigande majoritet av loci. Som jämförelse fann en hel exome-sekvenseringsstudie 2016 19 SNV och 3 indels i 5 redigerade möss, medan Schaefer et al. hittade 115 exoniska SNV: er och 9 indelar i bara 2 redigerade möss. Många experter höll inte med om uppsatsen och kritiserade den genom tidskriftsartiklar och sociala medier, och antydde att ovanliga CRISPR-behandlingar användes i den första uppsatsen och att urvalsstorleken var för låg för signifikans (n=2). Nature Methods har gett ut två redaktionella anteckningar om tidningen. Icke desto mindre visar sig off-target rater konsekvent vara vanligare in vivo jämfört med cellodlingsexperiment och tros vara särskilt vanliga hos människor.