Forensisk DNA-analys
| Del av en serie om |
| kriminalteknisk vetenskap |
|---|
 |
DNA-profilering är fastställandet av en DNA-profil för juridiska och utredningsändamål. DNA-analysmetoder har förändrats flera gånger under åren i takt med att tekniken förbättras och gör att mer information kan fastställas med mindre utgångsmaterial. Modern DNA-analys baseras på statistisk beräkning av sällsyntheten hos den producerade profilen inom en population.
Även om det är mest känt som ett verktyg i rättsmedicinska undersökningar, kan DNA-profilering också användas för icke-kriminaltekniska ändamål som faderskapstestning och mänsklig släktforskning .
Historia
Metoderna för att producera en DNA-profil utvecklades av Alec Jeffreys och hans team 1984.
Metoder
Pensionerade metoder
RFLP-analys
Den första riktiga metoden för DNA-profilering var polymorfismanalys av restriktionsfragmentlängd. Den första användningen av RFLP-analys i rättsmedicinskt ärende var 1985 i Storbritannien. Denna typ av analys använde variabelt antal tandemupprepningar (VNTR) för att skilja mellan individer. VNTR är vanliga i genomet och består av samma DNA-sekvens som upprepas om och om igen. Olika individer kan ha olika antal upprepningar på en specifik plats i genomet. Till exempel kan person A ha fyra medan person B kan ha 5 repetitioner. Skillnaderna visualiserades genom en process som kallas gelelektrofores . Mindre fragment skulle färdas längre genom gelén än större fragment som separerade dem. Dessa skillnader användes för att skilja mellan individer och när flera VNTR-platser kördes tillsammans har RFLP-analys en hög grad av individualiserande kraft.
Processen med RFLP-analys var extremt tidskrävande och på grund av längden på de upprepningar som användes, mellan 9 och 100 baspar, kunde amplifieringsmetoder såsom polymeraskedjereaktionen inte användas . Detta begränsade RFLP till prover som redan hade en större mängd DNA tillgängligt till att börja med och som inte fungerade bra med nedbrutna prover. RFLP-analys var den primära typen av analys som utfördes i de flesta kriminaltekniska laboratorier innan de slutligen pensionerades och ersattes av nyare metoder. Det övergavs helt av FBI 2000 och ersattes med STR-analys.
DQ alfatestning
DQ alfa-testning utvecklades 1991 och var den första kriminaltekniska DNA-tekniken som använde polymeraskedjereaktionen. Denna teknik gjorde det möjligt att använda mycket färre celler än RFLP-analys, vilket gjorde den mer användbar för brottsplatser som inte hade de stora mängder DNA-material som tidigare krävdes. DQ alfa 1- lokuset (eller platsen) var också polymorft och hade flera olika alleler som kunde användas för att begränsa poolen av individer som kunde ha producerat det resultatet och öka sannolikheten för uteslutning.
DQ alpha locus kombinerades med andra loci i ett kommersiellt tillgängligt kit som heter Polymarker 1993. Polymarker var en föregångare till moderna multiplexeringssatser och gjorde att flera olika loci kunde undersökas med en produkt. Även om den var känsligare än RFLP-analys, innehöll Polymarker inte samma diskriminerande kraft som den äldre RFLP-testningen. År 1995 försökte forskare återgå till en VNTR-baserad analys kombinerad med PCR-teknologi som kallas amplifierade fragmentlängdspolymorfismer (AmpFLP).
AmpFLP

AmpFLP var det första försöket att koppla VNTR-analys med PCR för rättsmedicinskt ärendearbete. Denna metod använde kortare VNTR än RFLP-analys, mellan 8 och 16 baspar. De kortare basparstorlekarna av AmpFLP designades för att fungera bättre med amplifieringsprocessen av PCR. Man hoppades att denna teknik skulle möjliggöra RFLP-analysens urskiljande kraft med förmågan att bearbeta prover som har mindre mall-DNA att arbeta med eller som på annat sätt har försämrats. Men endast ett fåtal loci validerades för kriminaltekniska applikationer för att fungera med AmpFLP-analys eftersom kriminaltekniska laboratorier snabbt gick vidare till andra tekniker begränsade dess urskiljningsförmåga för kriminaltekniska prover.
Tekniken användes i slutändan aldrig allmänt även om den fortfarande används i mindre länder på grund av dess lägre kostnad och enklare installation jämfört med nyare metoder. I slutet av 1990-talet började laboratorier gå över till nyare metoder inklusive STR-analys. Dessa använde ännu kortare fragment av DNA och kunde mer tillförlitligt amplifieras med PCR samtidigt som de bibehöll och förbättrade den diskriminerande kraften hos de äldre metoderna.
Aktuella metoder
STR-analys

Kort tandemupprepning (STR)-analys är den primära typen av kriminalteknisk DNA-analys som utförs i moderna DNA-laboratorier. STR-analys bygger på RFLP och AmpFLP som användes tidigare genom att krympa storleken på de upprepade enheterna, till 2 till 6 baspar, och genom att kombinera flera olika loci till en PCR-reaktion. Dessa multiplexerande analyskit kan producera allelvärden för dussintals olika loci genom genomet samtidigt som begränsar hur lång tid det tar att få en fullständig, individualiserande profil. STR-analys har blivit guldstandarden för DNA-profilering och används flitigt i rättsmedicinska tillämpningar.
STR-analys kan också begränsas till endast Y-kromosomen . Y-STR- analys kan användas i fall som involverar faderskap eller vid familjesökning eftersom Y-kromosomen är identisk längs faderlinjen (förutom i fall där en mutation inträffade). Vissa multiplexeringssatser kombinerar både autosomala och Y-STR-loci till ett kit, vilket ytterligare minskar den tid det tar att erhålla en stor mängd data.
För närvarande kräver STR-analys flera celler för att skapa en fullständig DNA-profil. Men vetenskapen närmar sig att skapa en fullständig DNA-profil med hjälp av STR-analys på enstaka celler.
mtDNA-sekvensering
Mitokondriell DNA-sekvensering är en specialiserad teknik som använder det separata mitokondriella DNA som finns i de flesta celler. Detta DNA förs vidare längs moderlinjen och är inte unikt mellan individer. Men på grund av antalet mitokondrier som finns i celler kan mtDNA-analys användas för mycket nedbrutna prover eller prover där STR-analys inte skulle producera tillräckligt med data för att vara användbar. mtDNA finns också på platser där autosomalt DNA skulle saknas, såsom i hårstrån.
På grund av den ökade risken för kontaminering vid hantering av mtDNA är det få laboratorier som bearbetar mitokondriella prover. De som har specialiserade protokoll på plats som ytterligare separerar olika prover från varandra för att undvika korskontaminering.
Snabbt DNA
Rapid DNA är en "swab in-profile out"-teknologi som helt automatiserar hela DNA-extraktionen, amplifieringen och analysprocessen. Snabba DNA-instrument kan gå från en svabb till en DNA-profil på så lite som 90 minuter och eliminerar behovet av utbildade forskare att utföra processen. Dessa instrument undersöks för användning i brottslingsprocessen, vilket gör det möjligt för poliser att få fram DNA-profilen för den gripna personen.
Nyligen antogs Rapid DNA Act of 2017 i USA, som uppmanade FBI att skapa protokoll för implementering av denna teknik i hela landet. För närvarande är DNA som erhållits från dessa instrument inte kvalificerat för uppladdning till nationella DNA-databaser eftersom de inte analyserar tillräckligt många loci för att uppfylla standardtröskeln. Men flera polismyndigheter använder redan Rapid DNA-instrument för att samla in prover från personer som arresterats i deras område. Dessa lokala DNA-databaser är inte föremål för federala eller statliga bestämmelser.
Massivt parallell sekvensering
Även känd som nästa generations sekvensering, massivt parallell sekvensering (MPS) bygger på STR-analys genom att introducera direkt sekvensering av loci. Istället för antalet upprepningar som finns på varje plats, skulle MPS ge vetenskapsmannen den faktiska basparsekvensen. Teoretiskt har MPS förmågan att skilja mellan identiska tvillingar eftersom slumpmässiga punktmutationer skulle ses inom upprepade segment som inte skulle kunna plockas upp av traditionell STR-analys.
Profilsällsynthet
När en DNA-profil används på ett bevismässigt sätt tillhandahålls en matchningsstatistik som förklarar hur sällsynt en profil är inom en population. Specifikt är denna statistik sannolikheten att en person som valts slumpmässigt ur en population skulle ha den specifika DNA-profilen. Det är inte sannolikheten att profilen "matchar" någon . Det finns flera olika metoder för att bestämma denna statistik och var och en används av olika laboratorier baserat på deras erfarenhet och preferenser. Men sannolikhetskvotsberäkningar håller på att bli den föredragna metoden framför de andra två vanligaste metoderna, slumpmässig man inte utesluten och kombinerad sannolikhet för inkludering. Matchningsstatistik är särskilt viktig vid tolkning av blandningar där det finns mer än en bidragsgivare till en DNA-profil. När denna statistik ges i en rättssal eller i en laboratorierapport ges den vanligtvis för de tre vanligaste raserna i det specifika området. Detta beror på att allelfrekvenserna vid olika loci ändrades baserat på individens härkomst. https://strbase.nist.gov/training/6_Mixture-Statistics.pdf
Slumpmässig man inte utesluten
Sannolikheten som produceras med denna metod är sannolikheten att en person som slumpmässigt valts ut från populationen inte kunde uteslutas från de analyserade uppgifterna. Den här typen av matchstatistik är lätt att förklara i en rättssal för individer som inte har någon vetenskaplig bakgrund, men den förlorar också mycket urskiljningsförmåga eftersom den inte tar hänsyn till den misstänktes genotyp. Detta tillvägagångssätt används ofta när provet är försämrat eller innehåller så många bidragsgivare att en enskild profil inte kan fastställas. Det är också användbart för att förklara för lekmän eftersom metoden för att få statistiken är enkel. På grund av dess begränsade särskiljningsförmåga utförs RMNE i allmänhet inte om ingen annan metod kan användas. RMNE rekommenderas inte för användning i data som indikerar att en blandning förekommer.
Kombinerad sannolikhet för inkludering/exkludering
Kombinerad sannolikhet för inkludering eller uteslutning beräknar sannolikheten för att en slumpmässig, obesläktad person skulle vara en bidragsgivare till en DNA-profil eller DNA-blandning. I denna metod bestäms statistik för varje enskilt lokus med hjälp av befolkningsstatistik och kombineras sedan för att få den totala KPI eller CPE. Dessa beräkningar upprepas för alla tillgängliga loci med all tillgänglig data och sedan multipliceras varje värde tillsammans för att få den totala kombinerade sannolikheten för inkludering eller exkludering. Eftersom värdena multipliceras med varandra kan extremt små tal uppnås med hjälp av KPI. KPI eller CPE anses vara en acceptabel statistisk beräkning när en blandning anges. https://www.promega.com/-/media/files/resources/conference-proceedings/ishi-15/parentage-and-mixture-statistics-workshop/generalpopulationstats.pdf?la=en
Exempelberäkning för enskild källprofil
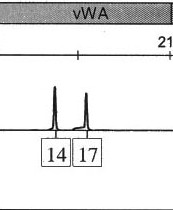
Sannolikheten för att en kaukasisk person har en 16-allel vid vWA = .10204
Sannolikheten för att en kaukasisk person har en 17-allel vid vWA = .26276
Sannolikheten för att en kaukasisk person har både en 14 och en 17 allel (P) = .10204 + .26276 = .3648
Sannolikheten för att alla andra alleler är närvarande (Q) = 1 - P eller 1 - .3648 = .6352
Sannolikhet för uteslutning för vWA = Q 2 + 2Q(1-Q) eller .6352 2 + 2(.6352)(1 - .6352) = .86692096 ≈ 86.69 %
Sannolikhet för inkludering för vWA = 1 - CPE eller 1 - .86692096 = .13307904 ≈ 13,31 %
Exempelberäkning för blandningsprofil
Sannolikheten för att en kaukasisk person har en 14-allel vid vWA = .10204
Sannolikheten för att en kaukasisk person har en 15-allel vid vWA = .11224
Sannolikheten för att en kaukasisk person har en 16-allel vid vWA = .20153
Sannolikheten för att en kaukasisk person har en 19-allel vid vWA = 0,08418
Sannolikheten för att en kaukasisk person har en 14, 15, 16 eller 19 allel ( P) = .10204 + .11224 + .20153 + .08418 = .49999
Sannolikhet för att alla andra alleler är närvarande (Q) = 1 - P eller 1 - .49999 = .50001
Sannolikhet för uteslutning för vWA = Q 2 + 2Q(1-Q) eller .50001 2 + 2(.50001)(1 - .50001) = .7500099999 ≈ 75 %
Sannolikhet för inkludering för vWA = 1 - CPE eller 1 - .7500099999 = .2499900001 ≈ 25 %
Sannolikhetsförhållande
Sannolikhetskvoter (LR) är en jämförelse av två olika sannolikheter för att avgöra vilken som är mer sannolik. När det handlar om en rättegång är LR sannolikheten för åklagarens argument kontra sannolikheten för försvarets argument givet deras utgångsantaganden. I detta scenario är åklagarens sannolikhet ofta lika med 1 eftersom antagandet är att åklagaren inte skulle åtala en misstänkt om de inte var helt säkra (100 %) på att de har rätt person. Sannolikhetskvoter blir vanligare i laboratorier på grund av deras användbarhet för att presentera statistik för data som indikerar flera bidragsgivare samt deras användning i probabilistisk genotypningsprogram som förutsäger de mest sannolika allelkombinationerna givet en uppsättning data.
Nackdelarna med att använda sannolikhetskvoter är att de är mycket svåra att förstå hur analytiker kommit fram till ett specifikt värde och den inblandade matematiken blir mycket komplicerad när mer data introduceras i ekvationerna. För att bekämpa dessa problem i en rättssal har vissa laboratorier satt upp en "verbal skala" som ersätter det faktiska siffervärdet för sannolikhetsförhållandet.