3D Slicer
 | |
| Originalförfattare | Slicer-gemenskapen |
|---|---|
| Stabil frisättning | 5.2.2 / 22 februari 2023
|
| Skrivet i | C++ , Python , Qt |
| Operativ system | Linux , macOS , Windows |
| Storlek | 200 MB |
| Tillgänglig i | engelsk |
| Typ | Vetenskaplig visualisering och bildberäkning |
| Licens | BSD-stil |
| Hemsida | |
3D Slicer ( Slicer ) är ett gratis och öppen källkodsprogram för bildanalys och vetenskaplig visualisering . Slicer används i en mängd olika medicinska tillämpningar, inklusive autism , multipel skleros , systemisk lupus erythematosus , prostatacancer , lungcancer , bröstcancer , schizofreni , ortopedisk biomekanik , KOL , hjärt-kärlsjukdomar och neurokirurgi .
Handla om
3D Slicer är en gratis programvara med öppen källkod (BSD-licens) som är en flexibel, modulär plattform för bildanalys och visualisering. 3D Slicer utökas för att möjliggöra utveckling av både interaktiva och batchbearbetningsverktyg för en mängd olika applikationer.
3D Slicer tillhandahåller bildregistrering , bearbetning av DTI (diffusionstraktografi) , ett gränssnitt till externa enheter för stöd för bildvägledning och GPU -aktiverad volymåtergivning , bland andra funktioner. 3D Slicer har en modulär organisation som tillåter tillägg av ny funktionalitet och ger ett antal generiska funktioner som inte är tillgängliga i konkurrerande verktyg. [ citat behövs ]
De interaktiva visualiseringsmöjligheterna i 3D Slicer inkluderar möjligheten att visa godtyckligt orienterade bildskivor, bygga ytmodeller från bildetiketter och hårdvaruaccelererad volymrendering. [ citat behövs ] 3D Slicer stöder också en rik uppsättning annoteringsfunktioner ( retro- och mätwidgets, anpassade färgkartor). [ citat behövs ]
Slicers funktioner inkluderar:
- Hantera DICOM-bilder och läsa/skriva en mängd andra format
- Interaktiv visualisering av volymetriska Voxel-bilder , polygonala maskor och volymrenderingar
- Manuell redigering
- Fusion och samregistrering av data med hjälp av stela och icke-rigida algoritmer
- Automatisk bildsegmentering
- Analys och visualisering av diffusionstensoravbildningsdata
- Spårning av enheter för bildstyrda procedurer.
Slicer är kompilerad för användning på flera datorplattformar, inklusive Windows , Linux och macOS .
Slicer distribueras under en BSD- stil, gratis, öppen källkodslicens. Licensen har inga begränsningar för användning av programvaran i akademiska eller kommersiella projekt. Det görs dock inga anspråk på att programvaran är användbar för någon speciell uppgift. Det är helt och hållet användarens ansvar att säkerställa efterlevnad av lokala regler och förordningar. Skivmaskinen har inte formellt godkänts för klinisk användning av FDA i USA eller av någon annan tillsynsmyndighet någon annanstans.
Bildgalleri
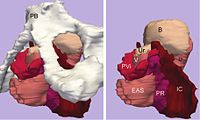
Visualisering av vissa atlasbaserade ROI som motsvarar stora anatomiska fiberkanaler. Atlasen tillhandahölls som en del av en nedladdning av DTI studio .
Historia
Slicer startade som ett masteruppsatsprojekt mellan Surgical Planning Laboratory vid Brigham and Women's Hospital och MIT Artificial Intelligence Laboratory 1998. 3D Slicer version 2 har laddats ner flera tusen gånger. 2007 släpptes en helt förnyad version 3 av Slicer. Nästa stora omstrukturering av Slicer inleddes 2009, vilket övergick Slicers gränssnitt från att använda KWWidgets till Qt . Qt-aktiverad Slicer version 4 släpptes 2011. Från och med 2022 hade Slicer 4 laddats ner över en miljon gånger av användare över hela världen.
Slicer-programvaran har möjliggjort en mängd forskningspublikationer, alla syftade till att förbättra bildanalys.
Detta betydande programvaruprojekt har möjliggjorts genom deltagande av flera storskaliga NIH -finansierade insatser, inklusive NA-MIC, NAC, BIRN , CIMIT, Harvard Catalyst och NCIGT-gemenskaperna. Finansieringsstödet kommer från flera federala finansieringskällor, inklusive NCRR , NIBIB , NIH Roadmap, NCI , NSF och DOD .
Användare
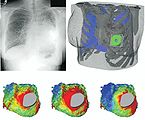
Slicers plattform tillhandahåller funktioner för segmentering, registrering och tredimensionell visualisering av multimodala bilddata, samt avancerade bildanalysalgoritmer för diffusionstensoravbildning , funktionell magnetisk resonanstomografi och bildstyrd strålterapi . Standardbildfilformat stöds, och applikationen integrerar gränssnittsfunktioner till biomedicinsk forskningsprogramvara .
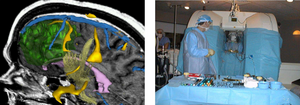
Slicer har använts i en mängd olika klinisk forskning . Inom bildstyrd terapiforskning används Slicer ofta för att konstruera och visualisera samlingar av MRI-data som är tillgängliga pre- och intraoperativt för att möjliggöra inhämtning av rumsliga koordinater för instrumentspårning. Faktum är att Slicer redan har spelat en så avgörande roll i bildstyrd terapi, att den kan anses växa upp vid sidan av det fältet, med över 200 publikationer som refererar till Slicer sedan 1998.
Förutom att producera 3D-modeller från konventionella MRI-bilder, har Slicer också använts för att presentera information som härrör från fMRI (med hjälp av MRI för att bedöma blodflödet i hjärnan relaterat till neural- eller ryggmärgsaktivitet), DTI (med MRT för att mäta den begränsade diffusionen av vatten i avbildad vävnad), och elektrokardiografi . Till exempel tillåter Slicers DTI-paket konvertering och analys av DTI-bilder. Resultaten av sådan analys kan integreras med resultaten från analys av morfologisk MRI, MR- angiogram och fMRI. Andra användningsområden för Slicer inkluderar paleontologi och neurokirurgiplanering.
Det finns en aktiv gemenskap på Slicers diskursserver .
Utvecklare
Slicer Developer Orientation erbjuder resurser för utvecklare som är nya på plattformen. Slicer-utvecklingen koordineras på Slicer Discourse-forumet och en sammanfattning av utvecklingsstatistiken finns tillgänglig på Ohloh.
3D Slicer är byggd på VTK , ett pipelinebaserat grafiskt bibliotek som används flitigt inom vetenskaplig visualisering och ITK , ett ramverk som ofta används för utveckling av bildsegmentering och bildregistrering . I version 4 är kärnapplikationen implementerad i C++ och API:et är tillgängligt via ett Python- omslag för att underlätta snabb, iterativ utveckling och visualisering i den medföljande Python-konsolen. Användargränssnittet är implementerat i Qt och kan utökas med antingen C++ eller Python.
Slicer stöder flera typer av modulär utveckling. Helt interaktiva, anpassade gränssnitt kan skrivas i C++ eller Python. Kommandoradsprogram på valfritt språk kan lindas med en lätt XML- specifikation, från vilken ett grafiskt gränssnitt genereras automatiskt.
För moduler som inte distribueras i Slicer-kärnapplikationen finns ett system tillgängligt för att automatiskt bygga och distribuera för selektiv nedladdning från Slicer. Denna mekanism underlättar inkorporeringen av kod med andra licenskrav än den tillåtande BSD-liknande licensen som används för Slicer-kärnan.
Byggprocessen för Slicer använder CMake för att automatiskt bygga förutsättnings- och valfria bibliotek (exklusive Qt). Kärnutvecklingscykeln inkluderar automatisk testning, såväl som inkrementella och nattliga uppbyggnader på alla plattformar, övervakade med hjälp av en online-instrumentpanel.
Slicers utveckling hanteras främst genom dess GitHub-förråd.
Externa beroenden
Se även
Anteckningar
- 1. ^ För en lista över publikationer som citerar Slicer-användning sedan 1998, besök: slicer.org
externa länkar
- Programvara för datorseende
- Gratis programvara för 3D-grafik
- Gratis DICOM-programvara
- Gratis programvara för sjukvård
- Gratis vetenskap programvara
- Gratis programvara programmerad i C++
- Gratis programvara programmerad i Python
- Fri programvara programmerad i Tcl
- Programvara för bildbehandling
- Vetenskapsprogramvara för Linux
- Vetenskapsprogram som använder Qt
- Programvara som använder VTK