SymE-SymR toxin-antitoxinsystem
| SymR | |
|---|---|
 Konserverad sekundär struktur av SymR RNA.
| |
| Identifierare | |
| Symbol | SymR |
| Rfam | RF01809 |
| Övriga uppgifter | |
| RNA -typ | Antisens-RNA |
| Domän(er) | E coli |
| PDB- strukturer | PDBe |
| SymE-toxin av typ I-toxin-antitoxinsystem | |||||||||
|---|---|---|---|---|---|---|---|---|---|
|
SymE-toxin av typ I-toxin-
| |||||||||
| antitoxinsystemidentifierare | |||||||||
| Symbol | SymE_toxin | ||||||||
| Pfam | PF13957 | ||||||||
| InterPro | IPR020883 | ||||||||
| PROSITE | PS51740 | ||||||||
| |||||||||
| https://swissmodel.expasy.org/repository/uniprot/P39394 | |||||||||
SymE -SymR-toxin-antitoxinsystemet består av ett litet symbiotiskt endonukleastoxin , SymE, och ett icke-kodande RNA- symbiotiskt RNA-antitoxin, SymR, som hämmar SymE-translation. SymE-SymR är ett typ I-toxin-antitoxinsystem och regleras av antitoxinet SymR. SymE-SymR-komplexet tros spela en viktig roll vid återvinning av skadat RNA och DNA . Relationen och motsvarande strukturer för SymE och SymR ger insikt i mekanismen för toxicitet och övergripande roll i prokaryota system.
Upptäckt
SymR var ursprungligen märkt RyjC och är ett 77 nukleotider (nt) RNA med en σ 70 promotor. RyjC befanns överlappa den öppna läsramen yjiW på den motsatta strängen med 6 nt, och karakteriserades som ett antisens-RNA som band den 5'-otranslaterade regionen av yjiW . Ytterligare studier ledde till att både yjiW och RyjC döptes om till SymE (SOS-inducerad yjiW-gen med likhet med MazE ) respektive SymR. Trots likheter med AbrB-superfamiljen har SymE-familjen uteslutande hittats i proteobakterier.
Förhållandet mellan SymE och SymR
SymR- antisens-RNA : t transkriberas 3 nt bakom SymE- startkodonet, vilket är anledningen till att SymR-promotom anses inbäddad i SymE- kodonet . Som ett resultat blockerar SymR RNA- translation av SymE genom antisensbindning, vilket tyder på att detta i slutändan leder till SymR-mRNA-nedbrytning. Aminosyraanalys har kommit fram till att SymE kan ha utvecklats till ett RNA-klyvningsprotein som uppvisar toxinliknande beteende på grund av transkriptionsfaktorer eller antitoxiner . I motsats till andra vanliga toxin-antitoxinsystem är SymR-antitoxinet mer stabilt än SymE-toxinet.
Efter DNA-skada undertrycker SOS -svaret transkription av SymR-RNA, vilket gör att SymE-toxin kan bryta ned potentiellt skadat mRNA tills DNA har reparerats . Omvänt är SymE hårt undertryckt av LexA- repressorbindningsställen, SymR och Lon-proteaset . Dessa tre faktorer finns på flera nivåer där LexA är involverat i transkriptionsnedreglering, SymR RNA är involverat i translationsnedreglering och Lon-proteas är involverat i proteinnedbrytning . Omfattningen av förtrycket på SymE är beroende av den additiva kraften hos LexA, SymR och Lon-proteas. Sammantaget är SymE-syntesen långsam eftersom dess aktivitet är starkt beroende av DNA-reparationsproteiner. I den cellulära miljön mitomycin C DNA vilket leder till ett överuttryck av SymE-mRNA för att initiera DNA-reparation.
Giftighet
Överuttrycket av SymE visade negativa effekter på tillväxten av kolonibildande celler när de testades in vitro . SymE uppvisar sin toxicitet genom att undertrycka global translation i cellen , klyver mRNA på ett liknande sätt som MazF , ett annat toxin. Kvantitativa Northern blot -experiment visade att SymR-RNA finns i celler vid 10 gånger koncentrationen av SymE-mRNA (0,02 fmol μg -1 och 0,2 fmol μg -1 ).
Strukturera
SymE
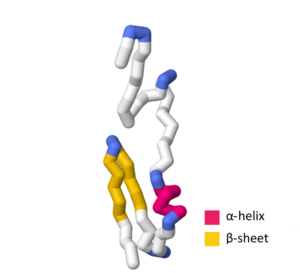
SymE-toxinet består av 113 aminosyror . Vid utvärdering av aminosyrasekvensen och den tertiära strukturen av SymE fann man starka likheter som liknar AbrB -superfamiljen. Denna superfamilj fungerar huvudsakligen som transkriptionsfaktorer eller antitoxiner ; Likheten mellan SymE och den primära sekvensen och tertiära strukturen hos AbrB-superfamiljen tyder dock på att SymE-proteiner upplevde en evolutionär förändring från en transkriptionsfaktor eller antitoxin till ett RNA-associerande protein som uppvisar toxinbeteende. Mellan AbrB-superfamiljens proteinstruktur och SymE-proteinstrukturen finns det flera viktiga hydrofoba rester som är mycket konserverade i -helixen i mitten av proteinet såväl som sträng -1. Trots dessa viktiga likheter uppvisar SymE polära rester som inte finns i den allmänna strukturen för AbrB-superfamiljen, vilket indikerar att dessa rester kan ha en roll i SymE RNA-klyvningsförmågan.
SWISS-MODEL innehåller mer än flera experimentella strukturer och teoretiska homologimodeller som definierar vissa aspekter av SymEs primära sekvens och tertiära struktur . UniProtKB- accessionsnumret P39394 indikerar den allmänna strukturen för SymE-toxinet i Escherichia coli (stam K12) . I den teoretiska modellen för SWISS-MODEL SymE -helixen aminosyrorna G 44, Q 45, W 46 , L 47, E 48, A 49 och A 50. -sträng -1 innehåller aminosyrorna G 55, T 56, A 57, V 58, D 59, V 60, K 61 , V 62, I 67, V 68, L 69, T 70, A 71, Q 72, P 73 och P 74 med -svängen som innehåller M 63, E 64, G 65 och C 66.
SymR
SymR är ett antisens-RNA vilket betyder att dess sekundära struktur har karakteristiska stam-och-loop- element såväl som oparade regioner som flankerar strukturen. Den förutsagda sekundära strukturen av SymR visar upp en slinga som innehåller nukleotidsekvensen CCAG. Denna karakteristiska loop delas med lstR-1 och OhsC RNA-proteinerna och förutsägs vara ett bindningsställe för andra proteiner. För närvarande finns det inga kända filer på RCSB-proteindatabanken eller SWISS-MODEL-förrådet som indikerar en förutspådd tertiär struktur för SymR.
Se även
Vidare läsning
- Buts L, Lah J, Dao-Thi MH, Wyns L, Loris R (december 2005). "Toxin-antitoxinmoduler som hanterare av bakteriell metabolisk stress". Trender inom biokemiska vetenskaper . 30 (12): 672–9. doi : 10.1016/j.tibs.2005.10.004 . PMID 16257530 .
- Gerdes K, Christensen SK, Løbner-Olesen A (maj 2005). "Prokaryot toxin-antitoxin stressrespons loci". Naturrecensioner. Mikrobiologi . 3 (5): 371–82. doi : 10.1038/nrmicro1147 . PMID 15864262 .
- Lewis LK, Harlow GR, Gregg-Jolly LA, Mount DW (augusti 1994). "Identifiering av högaffinitetsbindningsställen för LexA som definierar nya DNA-skada-inducerbara gener i Escherichia coli". Journal of Molecular Biology . 241 (4): 507–23. doi : 10.1006/jmbi.1994.1528 . PMID 8057377 .
- Christensen SK, Pedersen K, Hansen FG, Gerdes K (september 2003). "Toxin-antitoxin loci som stress-respons-element: ChpAK/MazF och ChpBK klyver översatta RNA och motverkas av tmRNA". Journal of Molecular Biology . 332 (4): 809–19. doi : 10.1016/S0022-2836(03)00922-7 . PMID 12972253 .
- Engelberg-Kulka H, Glaser G (1999). "Beroendemoduler och programmerad celldöd och antidöd i bakteriekulturer". Årlig översyn av mikrobiologi . 53 : 43–70. doi : 10.1146/annurev.micro.53.1.43 . PMID 10547685 .
- Cherepanov PP, Wackernagel W (maj 1995). "Genstörning i Escherichia coli: TcR- och KmR-kassetter med möjlighet till Flp-katalyserad excision av antibiotikaresistensdeterminanten". Gene . 158 (1): 9–14. doi : 10.1016/0378-1119(95)00193-A . PMID 7789817 .
- Anantharaman V, Aravind L (2003). "Nya kopplingar i det prokaryota toxin-antitoxin-nätverket: samband med det eukaryota nonsens-medierade RNA-sönderfallssystemet" . Genombiologi . 4 (12): R81. doi : 10.1186/gb-2003-4-12-r81 . PMC 329420 . PMID 14659018 .