Split genteori
Den delade genteorin är en teori om ursprunget till introner , långa icke-kodande sekvenser i eukaryota gener mellan exonerna . Teorin hävdar att slumpmässigheten hos primordiala DNA- sekvenser endast skulle tillåta små (< 600 bp) öppna läsramar (ORF), och att viktiga intronstrukturer och regulatoriska sekvenser härrör från stoppkodoner . I detta introns-första ramverk utvecklades det spliceosomala maskineriet och kärnan på grund av nödvändigheten att förena dessa ORF:er (nu "exoner") till större proteiner, och att intronlösa bakteriegener är mindre uråldriga än de delade eukaryota generna. Teorin har sitt ursprung i Periannan Senapathy .
Teorin ger lösningar på nyckelfrågor som rör den delade genarkitekturen, inklusive delade eukaryota gener, exoner, introner, splitsningsövergångar och förgreningspunkter, baserat på ursprunget till delade gener från slumpmässiga genetiska sekvenser. Det ger också möjliga lösningar på ursprunget till det spliceosomala maskineriet, kärngränsen och den eukaryota cellen.
Denna teori ledde till Shapiro-Senapathy-algoritmen , som tillhandahåller metodiken för att upptäcka splitsningsställena, exonerna och delade generna i eukaryot DNA, och som är huvudmetoden för att upptäcka splitsningsställemutationer i gener som orsakar hundratals sjukdomar.
Split genteori kräver ett separat ursprung för alla eukaryota arter. Det kräver också att de enklare prokaryoterna utvecklats från eukaryoter. Detta motsäger fullständigt den vetenskapliga konsensus om bildandet av eukaryota celler genom endosymbios av bakterier. 1994 skrev Senapathy en bok om denna aspekt av sin teori - The Independent Birth of Organisms. Den föreslog att alla eukaryota genom bildades separat i en primordial pool. Den holländska biologen Gert Korthoff kritiserade teorin genom att ställa olika problem som inte kan förklaras med en teori om oberoende ursprung. Han påpekade att olika eukaryoter behöver omvårdnad och kallade detta "stövelproblemet", eftersom även den första eukaryoten behövde föräldravård. Korthoff noterar att en stor del av eukaryoter är parasiter. Senapathys teori skulle kräva en slump för att förklara deras existens. Senapathys teori kan inte förklara de starka bevisen för vanlig härkomst ( homologi , universell genetisk kod, embryologi , fossilregister .)
Bakgrund

Gener från alla organismer, utom bakterier, består av korta proteinkodande regioner ( exoner ) avbrutna av långa sekvenser ( introner ). När en gen uttrycks kopieras dess DNA-sekvens till en "primär RNA"-sekvens av enzymet RNA-polymeras . Sedan tar "spliceosom"-maskineriet fysiskt bort intronerna från RNA-kopian av genen genom splitsningsprocessen, vilket bara lämnar en sammanhängande serie av exoner, som blir budbärar- RNA (mRNA). Detta mRNA läses nu av ribosomen , som producerar det kodade proteinet. Således, även om introner inte avlägsnas fysiskt från en gen, läses en gens sekvens som om introner inte var närvarande.
Exoner är vanligtvis korta, med en genomsnittlig längd på cirka 120 baser (t.ex. i mänskliga gener). Intronlängder varierar mycket från 10 till 500 000, men exonlängder har en övre gräns på cirka 600 baser i de flesta eukaryoter. Eftersom exoner kodar för proteinsekvenser är de viktiga för cellen, men utgör ändå bara ~2% av sekvenserna. Introner, däremot, utgör 98 % av sekvenserna men verkar ha få avgörande funktioner, förutom förstärkarsekvenser och utvecklingsregulatorer i sällsynta fall.
Tills Philip Sharp och Richard Roberts upptäckte introner i eukaryota gener 1977, trodde man att den kodande sekvensen för alla gener alltid var i en enda sträcka, avgränsad av en enda lång ORF. Upptäckten av introner var en djupgående överraskning, som omedelbart tog upp frågorna om hur, varför och när intronerna kom in i de eukaryota generna.
Det blev snart uppenbart att en typisk eukaryot gen avbröts på många platser av introner, vilket delar upp den kodande sekvensen i många korta exoner. Också överraskande var att intronerna var långa, lika långa som hundratusentals baser. Dessa fynd väckte frågorna om varför många introner förekommer inom en gen (till exempel ~312 introner förekommer i den mänskliga genen TTN), varför de är långa och varför exoner är korta.
| Gen symbol |
Genlängd (baser) |
Längsta intronlängd (baser) |
Antal introner i genen |
|---|---|---|---|
| ROBO2 | 1,743,269 | 1,160,411 | 104 |
| KCNIP4 | 1,220,183 | 1 097 903 | 76 |
| ASIC2 | 1,161,877 | 1 043 911 | 18 |
| NRG1 | 1,128,573 | 956,398 | 177 |
| DPP10 | 1,403,453 | 866 399 | 142 |
Det upptäcktes också att spliceosommaskineriet var stort och komplext med ~300 proteiner och flera SnRNA- molekyler. Frågorna sträckte sig till spliceosomens ursprung. Strax efter upptäckten av introner blev det uppenbart att förbindelserna mellan exoner och introner på båda sidor uppvisade specifika sekvenser som styrde spliceosommaskineriet till den exakta baspositionen för splitsning. Hur och varför dessa skarvövergångssignaler kom till var en annan viktig fråga.
Historia
Upptäckten av introner och den delade genarkitekturen för de eukaryota generna startade en ny era av eukaryotisk biologi. Frågan om varför eukaryota gener hade fragmenterade gener föranledde spekulationer och diskussion nästan omedelbart.
Ford Doolittle publicerade en artikel 1978 där han påstod att de flesta molekylärbiologer antog att det eukaryota genomet uppstod från ett "enklare" och mer "primitivt" prokaryotiskt genom snarare som Escherichia coli . Denna typ av evolution skulle emellertid kräva att introner introduceras i de kodande sekvenserna för bakteriegener. Angående detta krav sa Doolittle: "Det är utomordentligt svårt att föreställa sig hur informationsmässigt irrelevanta sekvenser skulle kunna introduceras i redan existerande strukturella gener utan skadliga effekter." Han uttalade "Jag skulle vilja hävda att det eukaryota genomet, åtminstone i den aspekten av dess struktur som manifesteras som 'gener i bitar', faktiskt är den primitiva ursprungliga formen."
James Darnell uttryckte liknande åsikter 1978. Han sade: "Skillnaderna i biokemin av budbärar-RNA-bildning i eukaryoter jämfört med prokaryoter är så djupgående att de antyder att sekventiell prokaryotisk till eukaryot cellevolution verkar osannolik. De nyligen upptäckta icke-sammanhängande sekvenserna i eukaryot DNA som kodar budbärar-RNA kan återspegla en gammal, snarare än en ny, distribution av information i DNA och att eukaryoter utvecklades oberoende av prokaryoter."
Men i ett uppenbart försök att förena med tanken att RNA föregick DNA i evolutionen, och med konceptet om de tre evolutionära linjerna archea, bakterier och eukarya, avvek både Doolittle och Darnell från sin ursprungliga spekulation i ett gemensamt dokument 1985. De föreslog att förfadern till alla tre grupper av organismer, " progenoten ", hade en gener i bitar, från vilken alla tre linjerna utvecklades. De spekulerade i att det precellulära stadiet hade primitiva RNA-gener som hade introner, som omvänt transkriberades till DNA och bildade progenoten. Bakterier och archea utvecklades från progenoten genom att förlora introner, och "urkaryote" utvecklades från den genom att behålla introner. Senare utvecklades eukaryoten från urkaryoten genom att utveckla en kärna och absorbera mitokondrier från bakterier. Flercelliga organismer utvecklades sedan från eukaryoten.
Dessa författare förutspådde att skillnaderna mellan prokaryoten och eukaryoten var så djupgående att utvecklingen av prokaryot till eukaryot inte var hållbar och hade olika ursprung. Men förutom spekulationerna om att de precellulära RNA-generna måste ha haft introner tog de inte upp nyckelfrågorna om intronursprung. Inga förklaringar beskrev varför exoner var korta och introner var långa, hur splitsningsövergångarna uppstod, vad strukturen och sekvensen av splitsningsövergångarna betydde och varför eukaryota genom var stora.
Ungefär samtidigt som Doolittle och Darnell föreslog att introner i eukaryota gener kunde vara uråldriga, publicerade Colin Blake och Walter Gilbert sina åsikter om introns ursprung oberoende. Enligt deras uppfattning uppstod introner som spacersekvenser som möjliggjorde bekväm rekombination och blandning av exoner som kodade för distinkta funktionella domäner för att utveckla nya gener. Sålunda sattes nya gener samman från exonmoduler som kodade för funktionella domäner, vikningsregioner eller strukturella element från redan existerande gener i arvsmassan hos en förfädlig organism, och utvecklade därmed gener med nya funktioner. De specificerade inte hur exoner eller introner uppstod. Dessutom, även efter många år, visade omfattande analyser av tusentals proteiner och gener att det endast ytterst sällan uppvisar gener det förmodade exon-shuffling-fenomenet. Vidare ifrågasatte molekylärbiologer exon-blandningsförslaget, från en rent evolutionär syn på både metodologiska och konceptuella skäl, och i det långa loppet överlevde inte denna teori.
Hypotes
Ungefär när introner upptäcktes frågade Senapathy hur generna själva kunde ha sitt ursprung. Han förmodade att för att någon gen skulle komma till måste genetiska sekvenser (RNA eller DNA) ha funnits i den prebiotiska miljön. En grundläggande fråga han ställde var hur proteinkodande sekvenser kunde ha sitt ursprung från primordiala DNA-sekvenser vid ursprunget till de första cellerna.
För att svara på detta gjorde han två grundläggande antaganden:
- innan en självreplikerande cell kunde komma till existens, syntetiserades DNA-molekyler i ursoppan genom slumpmässig tillsats av de 4 nukleotiderna utan hjälp av mallar och
- nukleotidsekvenserna som kodar för proteiner valdes från dessa redan existerande slumpmässiga DNA-sekvenser i ursoppan, och inte genom konstruktion från kortare kodande sekvenser.
Han antog också att kodoner måste ha etablerats innan de första genernas ursprung. Om primordialt DNA verkligen innehöll slumpmässiga nukleotidsekvenser, frågade han: Fanns det en övre gräns för kodningssekvenslängder, och i så fall spelade denna gräns en avgörande roll i bildandet av de strukturella egenskaperna hos gener vid genernas ursprung?
Hans logik var följande. Den genomsnittliga längden av proteiner i levande organismer, inklusive de eukaryota och bakteriella organismerna, var ~400 aminosyror. Det fanns dock mycket längre proteiner, till och med längre än 10 000-30 000 aminosyror i både eukaryoter och bakterier. Således existerade den kodande sekvensen av tusentals baser i en enda sträcka i bakteriegener. Däremot existerade den kodande sekvensen för eukaryoter endast i korta segment av exoner på ~120 baser oavsett proteinets längd. Om den kodande sekvensens ORF-längder i slumpmässiga DNA-sekvenser var lika långa som de i bakteriella organismer, var långa, sammanhängande kodande gener möjliga i slumpmässigt DNA. Detta var inte känt, eftersom fördelningen av ORF-längder i en slumpmässig DNA-sekvens aldrig hade studerats.
Eftersom slumpmässiga DNA-sekvenser kunde genereras i datorn, trodde Senapathy att han kunde ställa dessa frågor och utföra sina experiment i silico . Dessutom, när han började studera denna fråga, fanns tillräcklig DNA- och proteinsekvensinformation i databasen National Biomedical Research Foundation (NBRF) i början av 1980-talet.
Testar hypotesen
Ursprunget för introner/splittrade gener

Senapathy analyserade fördelningen av ORF-längderna i datorgenererade slumpmässiga DNA-sekvenser först. Överraskande nog visade denna studie att cirka 200 kodon (600 baser) var den övre gränsen för ORF-längder. Den kortaste ORF (noll bas i längd) var den vanligaste. Vid ökande längder av ORF minskade deras frekvens logaritmiskt och närmade sig noll vid cirka 600 baser. När sannolikheten för ORF-längder i en slumpmässig sekvens plottades, avslöjade det att sannolikheten för ökande längder av ORF:er minskade exponentiellt och avtog vid maximalt cirka 600 baser. Från denna "negativa exponentiella" fördelning av ORF-längder fann man att de flesta ORF:er var mycket kortare än maximum.

Detta fynd var överraskande eftersom den kodande sekvensen för den genomsnittliga proteinlängden på 400 AA:er (med ~1 200 baser av kodande sekvens) och längre proteiner av tusentals AA:er (kräver >10 000 baser av kodande sekvens) inte skulle förekomma i en sträcka slumpmässigt sekvens. Om detta var sant kunde en typisk gen med en sammanhängande kodande sekvens inte ha sitt ursprung i en slumpmässig sekvens. Det enda möjliga sättet att vilken gen som helst skulle kunna härröra från en slumpmässig sekvens var att dela upp den kodande sekvensen i kortare segment och välja dessa segment från korta ORF:er tillgängliga i den slumpmässiga sekvensen, snarare än att öka ORF-längden genom att eliminera på varandra följande stoppkodon. Denna process med att välja korta segment av kodande sekvenser från de tillgängliga ORF:erna för att göra en lång ORF skulle leda till en delad struktur.
Om denna hypotes var sann borde eukaryota DNA-sekvenser återspegla den. När Senapathy plottade fördelningen av ORF-längder i eukaryota DNA-sekvenser, var plottet anmärkningsvärt likt det från slumpmässiga DNA-sekvenser. Denna plot var också en negativ exponentiell fördelning som svansade av vid maximalt cirka 600 baser, som med eukaryota gener, vilket sammanföll exakt med den maximala längden av ORF som observerades i både slumpmässigt DNA och eukaryota DNA-sekvenser.
De splittrade generna härstammar alltså från slumpmässiga DNA-sekvenser genom att välja det bästa av de korta kodande segmenten (exoner) och splitsa dem. De mellanliggande intronsekvenserna var kvarvarande rester av de slumpmässiga sekvenserna och var därför öronmärkta för att avlägsnas av spliceosomen. Dessa fynd tydde på att delade gener kunde ha sitt ursprung från slumpmässiga DNA-sekvenser med exoner och introner som de uppträder i dagens eukaryota organismer. Nobelpristagaren Marshall Nirenberg , som dechiffrerade kodonen, konstaterade att dessa fynd starkt visade att teorin om delad gen för ursprunget till introner och den delade strukturen av gener måste vara giltig.
Blake föreslog Gilbert-Blake-hypotesen 1979 för ursprunget till introner och påstod att Senapathys teori för delad gen på ett omfattande sätt förklarade ursprunget till den delade genstrukturen. Dessutom uppgav han att det förklarade flera nyckelfrågor inklusive ursprunget till skarvningsmekanismen:
Senaste arbete av Senapathy, när det tillämpas på RNA, förklarar på ett omfattande sätt ursprunget till den segregerade formen av RNA i kodande och icke-kodande regioner. Det antyder också varför en splitsningsmekanism utvecklades i början av primordial evolution. Han fann att fördelningen av läsramlängder i en slumpmässig nukleotidsekvens motsvarade exakt den för den observerade fördelningen av eukaryota exonstorlekar. Dessa var avgränsade av regioner som innehöll stoppsignaler, meddelanden om att avsluta konstruktionen av polypeptidkedjan, och var således icke-kodande regioner eller introner. Närvaron av en slumpmässig sekvens var därför tillräcklig för att i den ursprungliga förfadern skapa den segregerade formen av RNA som observerades i den eukaryota genstrukturen. Dessutom visar den slumpmässiga fördelningen också en cutoff vid 600 nukleotider, vilket antyder att den maximala storleken för en tidig polypeptid var 200 rester, återigen som observerats i den maximala storleken på den eukaryota exonen. Sålunda, som svar på evolutionära tryck för att skapa större och mer komplexa gener, sammanfogades RNA-fragmenten med en splitsningsmekanism som tog bort intronerna. Därför verkar den tidiga förekomsten av både introner och RNA-splitsning i eukaryoter vara mycket trolig utifrån en enkel statistisk grund. Dessa resultat stämmer också överens med det linjära förhållandet mellan antalet exoner i genen för ett visst protein och längden på polypeptidkedjan."

Ursprunget till skarvövergångar
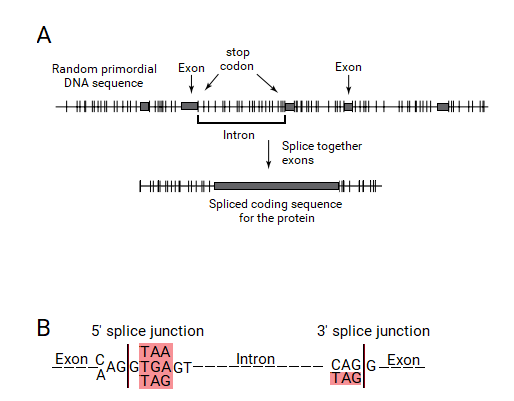
Under split genteorin definieras en exon av en ORF. Det kräver en mekanism för att känna igen en ORF som har sitt ursprung. Eftersom en ORF definieras av en sammanhängande kodande sekvens bunden av stoppkodon, måste dessa stoppkodonändar kännas igen av exon-intron-genigenkänningssystemet. Detta system kunde ha definierat exonerna genom närvaron av ett stoppkodon i ändarna av ORF, som bör inkluderas i ändarna av intronerna och elimineras genom splitsningsprocessen. Således bör intronerna innehålla ett stoppkodon vid sina ändar, vilket skulle vara en del av splitsningsövergångssekvenserna.
Om denna hypotes var sann borde de splittrade generna från dagens levande organismer innehålla stoppkodon exakt i ändarna av introner. När Senapathy testade denna hypotes i splitsningsövergångarna av eukaryota gener, fann han att den stora majoriteten av splitsningsövergångarna innehöll ett stoppkodon i slutet av varje intron, utanför exonerna. Faktum är att dessa stoppkodoner bildade den "kanoniska" GT:AG splitsningssekvensen, med de tre stoppkodonen som uppträdde som en del av de starka konsensussignalerna. Sålunda ledde den grundläggande split-genteorin för ursprunget till introner och den delade genstrukturen till förståelsen att splitsningsförbindelserna härrörde från stoppkodonen.
| Codon |
Antal förekomster i givarsignal |
Antal förekomster i acceptorsignal |
|---|---|---|
| TAA | 370 | 0 |
| TGA | 293 | 0 |
| MÄRKA | 64 | 234 |
| CAG | 7 | 746 |
| Övrig | 297* | 50 |
| Total | 1030 | 1030 |
| *Mer än 70 % är SKATT [TAT = 75; TAC = 59; TGT = 70]. | ||
Sekvensdata för endast cirka 1 000 exon-intron-övergångar var tillgängliga när Senapathy funderade på denna fråga. Han tog data för 1 030 splitsningsövergångssekvenser (donatorer och acceptorer) och räknade kodonerna som förekommer vid var och en av 7-baspositionerna i donatorsignalsekvensen [CAG:GTGAGT] och var och en av de möjliga 2-baspositionerna i acceptorsignalen [CAG:G] från GenBank-databasen. Han fann att stoppkodonen inträffade vid hög frekvens endast vid den 5:e baspositionen i donatorsignalen och den första baspositionen i acceptorsignalen. Dessa positioner är* början av intronen (i själva verket en bas efter starten) och i slutet av intronen, som Senapathy hade förutspått. Kodonantal vid endast dessa positioner visas. Även när kodonen vid dessa positioner inte var stoppkodon började 70 % av dem med de två första baserna av stoppkodonen TA och TG [TAT = 75; TAC = 59; TGT = 70].
Alla tre stoppkodonen (TGA, TAA och TAG) hittades efter en bas (G) i början av introner. Dessa stoppkodon visas i den konsensus kanoniska donatorsplitsningsövergången som AG:GT(A/G)GGT, varvid TAA och TGA är stoppkodonen, och det ytterligare TAG är också närvarande vid denna position. Förutom kodonet CAG hittades endast TAG, som är ett stoppkodon, i ändarna av introner. Den kanoniska acceptorsplitsningsövergången visas som (C/T)AG:GT, där TAG är stoppkodonet. Dessa konsensussekvenser visar tydligt närvaron av stoppkodonen i ändarna av introner som gränsar till exonerna i alla eukaryota gener, vilket ger en stark bekräftelse för teorin om delad gen. Nirenberg uppgav återigen att dessa observationer fullt ut stödde teorin om delad gen för ursprunget för splitsningsövergångssekvenser från stoppkodon.
Strax efter upptäckten av introner av Philip Sharp och Richard Roberts blev det känt att mutationer i skarvkorsningar kunde leda till sjukdomar. Senapati visade att mutationer i stoppkodonbaserna (kanoniska baser) orsakade fler sjukdomar än mutationerna i icke-kanoniska baser.
Grenpunktssekvens (lariat).
Ett mellanstadium i processen för eukaryot RNA-skarvning är bildandet av en lariatstruktur. Den är förankrad vid en adenosinrest i intron mellan 10 och 50 nukleotider uppströms om 3'-splitsningsstället. En kort bevarad sekvens (grenpunktssekvensen) fungerar som igenkänningssignal för platsen för lariatbildning. Under splitsningsprocessen bildar denna konserverade sekvens mot slutet av intronen en lariatstruktur med början av intronen. Det sista steget i splitsningsprocessen inträffar när de två exonerna förenas och intronet frisätts som ett lariat-RNA.
Flera forskare fann grenpunktssekvenserna i olika organismer inklusive jäst, människa, fruktfluga, råtta och växter. Senapathy fann att i alla dessa sekvenser är kodonet som slutar vid grenpunkten adenosin genomgående ett stoppkodon. Vad som är intressant är att två av de tre stoppkodonen (TAA och TGA) förekommer nästan hela tiden vid denna position.
| Organism | Lariat Konsensussekvens |
|---|---|
| Jäst | TAC TAA C |
| Mänskliga betaglobingener | C TGA C C TAA T C TGA T C TAA C CTCAC |
| Drosophila | C TAA T |
| Råttor | C TGA C |
| Växter | (C/T) T(A/G)A (T/C) |
|
Konsekvent närvaro av stoppkodon i grenpunktssignalsekvenser.
Lariat (grenpunkt) sekvenser har identifierats från många olika organismer. Dessa sekvenser visar genomgående att kodonet som slutar på det förgrenade adenosinet är ett stoppkodon, antingen TAA eller TGA, som visas i rött. |
|
Dessa fynd fick Senapathy att föreslå att grenpunktssignalen härrörde från stoppkodon. Upptäckten att två olika stoppkodon (TAA och TGA) förekommer inom lariatsignalen med förgreningspunkten som den tredje basen av stoppkodonen bekräftar detta förslag. Eftersom förgreningspunkten för lariat inträffar vid det sista adeninet i stoppkodonet, är det möjligt att spliceosommaskineriet som uppstod för eliminering av stoppkodonen från den primära RNA-sekvensen skapade en extra stoppkodonsekvenssignal som lariatsekvensen för att underlätta dess skarvningsfunktion.
Det lilla nukleära U2-RNA:t som finns i splitsningskomplex tros underlätta splitsningen genom att interagera med lariatsekvensen. Komplementära sekvenser för både lariatsekvensen och acceptorsignalen finns i ett segment av endast 15 nukleotider i U2 RNA. Vidare har U1 RNA föreslagits att fungera som en guide vid splitsning för att identifiera den exakta donatorsplitsningsövergången genom komplementär basparning. De konserverade regionerna av U1 RNA inkluderar således sekvenser som är komplementära till stoppkodonen. Dessa observationer gjorde det möjligt för Senapathy att förutsäga att stoppkodon hade opererat i ursprunget till inte bara splitsningsövergångssignalerna och lariatsignalen, utan även några små nukleära RNA.
Genreglerande sekvenser
Senapathy föreslog att de reglerande sekvenserna för genuttryck (promotor- och poly-A-additionsställesekvenser) också kunde ha sitt ursprung från stoppkodon. En konserverad sekvens, AATAAA, finns i nästan varje gen en kort bit nedströms från slutet av det proteinkodande meddelandet och fungerar som en signal för tillägg av poly(A) i genens mRNA-kopia. Denna poly(A)-sekvenssignal innehåller ett stoppkodon, TAA. En sekvens kort nedströms från denna signal, som anses vara en del av den fullständiga poly(A)-signalen, innehåller också TAG- och TGA-stoppkodonen.
Eukaryota RNA-polymeras-II-beroende promotorer kan innehålla en TATA-box (konsensussekvens TATAAA), som innehåller stoppkodonet TAA. Bakteriella promotorelement vid ~10 baser uppvisar en TATA-box med en konsensus av TATAAT (som innehåller stoppkodonet TAA), och vid -35 baser uppvisar en konsensus av TTGACA (innehållande stoppkodonet TGA). Således verkar utvecklingen av hela RNA-bearbetningsmekanismen ha påverkats av den alltför frekventa förekomsten av stoppkodon, vilket gör stoppkodonen till fokuspunkterna för RNA-bearbetning.
Stoppkodon är nyckeldelar i varje genetiskt element i den eukaryota genen

| Genetiskt element | Konsensussekvens |
|---|---|
| Promotor | TA TAA T |
| Donator Splice Sequence | CAG:G TAA GT CAG:G TGA GT |
| Acceptor skarvsekvens | (C/T)9... TAG :GT |
| Lariat-sekvens | C TGA C C TAA C |
| Poly-A tilläggswebbplats | TA TAA A |
| Den konsekventa förekomsten av stoppkodon i genetiska element i eukaryota gener. Konsensussekvenserna för de olika genetiska elementen i eukaryota gener visas. Stoppkodonen/stoppkodonen i var och en av dessa sekvenser är färgade i rött. | |
Senapathy upptäckte att stoppkodon förekommer som nyckeldelar i varje genetiskt element i eukaryota gener. Tabellen och figuren visar att nyckeldelarna av kärnpromotorelementen, lariatsignalen, donator- och acceptorsplitsningssignalerna och poly-A-additionssignalen består av ett eller flera stoppkodon. Detta fynd bekräftar påståendet om split-genteorin att den underliggande orsaken till det fullständiga split-genparadigmet är ursprunget till split-gener från slumpmässiga DNA-sekvenser, där slumpmässig distribution av en extremt hög frekvens av stoppkodon användes av naturen för att definiera dessa genetiska element.
Korta exoner/långa introner
Forskning baserad på teorin om split gen belyser andra grundläggande frågor om exoner och introner. Eukaryoternas exoner är i allmänhet korta (mänskliga exoner i genomsnitt ~120 baser och kan vara så korta som 10 baser) och introner är vanligtvis långa (i genomsnitt ~3 000 baser och kan vara flera hundra tusen baser långa), till exempel generna RBFOX1 , CNTNAP2, PTPRD och DLG2. Senapathy gav ett rimligt svar på dessa frågor, den enda förklaringen hittills. Om eukaryota gener härstammar från slumpmässiga DNA-sekvenser måste de matcha längden på ORF från slumpmässiga sekvenser, och bör möjligen vara runt 100 baser (nära medianlängden för ORF i slumpmässig sekvens). Genomsekvenserna för levande organismer uppvisar exakt samma genomsnittliga längder på 120 baser för exoner, och de längsta exonerna på 600 baser (med få undantag), vilket är samma längd som de längsta slumpmässiga ORF:erna.
Om delade gener har sitt ursprung i slumpmässiga DNA-sekvenser, skulle introner vara långa av flera skäl. Stoppkodonen förekommer i kluster som leder till många på varandra följande korta ORF: längre ORF som kan definieras som exoner skulle vara sällsyntare. Dessutom skulle den bästa av de kodande sekvensparametrarna för funktionella proteiner väljas från de långa ORF:erna i slumpmässig sekvens, vilket kan förekomma sällan. Dessutom skulle kombinationen av donator- och acceptorsplitsningsövergångssekvenser inom korta längder av kodande sekvenssegment som skulle definiera exongränser förekomma sällan i en slumpmässig sekvens. Dessa kombinerade skäl skulle göra introns långa jämfört med exoner.
Eukaryota genom
Detta arbete förklarar också varför genom som det mänskliga genomet har miljarder baser, och varför endast en liten del (~2%) kodar för proteiner och andra regulatoriska element. Om delade gener härrörde från slumpmässiga primordiala DNA-sekvenser, skulle de innehålla en betydande mängd DNA som representeras av introner. Vidare skulle ett genom sammansatt från slumpmässigt DNA innehållande delade gener också innefatta intergeniskt slumpmässigt DNA. Genomer som härrörde från slumpmässiga DNA-sekvenser måste alltså vara stora, oavsett organismens komplexitet.
Observationen att flera organismer som löken (~16 miljarder baser) och salamander (~32 miljarder baser) har mycket större genom än människor (~3 miljarder baser) medan organismerna inte är mer komplexa än människor stämmer överens med teorin. Dessutom det faktum att flera organismer med mindre genom har ett liknande antal gener som människan, såsom C. elegans (genomstorlek ~100 miljoner baser, ~19 000 gener) och Arabidopsis thaliana (genomstorlek ~125 miljoner baser, ~25 000 gener) ), stödjer teorin. Teorin förutspår att intronerna i de delade generna i dessa genom kan vara den "reducerade" (eller raderade) formen jämfört med större gener med långa introner, vilket leder till reducerade genom. Faktum är att forskare nyligen föreslagit att dessa mindre genom faktiskt är reducerade genom.
Spliceosomal maskineri och eukaryot kärna
Senapati tog upp ursprunget till det spliceosomala maskineriet som redigerar ut intronerna från RNA-transkript. Om de splittrade generna hade sitt ursprung från slumpmässigt DNA, så skulle intronerna ha blivit en onödig men integrerad del av eukaryota gener tillsammans med splitsningsövergångarna. Det spliceosomala maskineriet skulle krävas för att ta bort dem och för att göra det möjligt för de korta exonerna att linjärt splitsas samman som ett kontinuerligt kodande mRNA som kan översättas till ett komplett protein. Således hävdar teorin om delad gen att spliceosomal maskineri existerar för att ta bort onödiga introner.
Blake säger, "Work by Senapathy, när det tillämpas på RNA, förklarar på ett omfattande sätt ursprunget till den segregerade formen av RNA i kodande och icke-kodande regioner. Det antyder också varför en splitsningsmekanism utvecklades i början av den ursprungliga evolutionen."
Eukaryoter
Senapathy föreslog ett rimligt mekanistiskt och funktionellt skäl till varför den eukaryota kärnan uppstod, en viktig fråga inom biologin. Om transkripten av de splittrade generna och de splitsade mRNA:erna fanns i en cell utan kärna, skulle ribosomerna försöka binda till både det icke-splitsade primära RNA-transkriptet och det splitsade mRNA:t, vilket skulle resultera i kaos. En gräns som skiljer RNA-skarvningsprocessen från mRNA-translationen undviker detta problem. Den nukleära gränsen ger en tydlig separation av den primära RNA-skarvningen och mRNA-translationen.
Dessa undersökningar ledde således till möjligheten att ur-DNA med väsentligen slumpmässig sekvens gav upphov till den komplexa strukturen av de splittrade generna med exoner, introner och splitsningsövergångar. Celler som hyste delade gener måste vara komplexa med en nukleär cytoplasmatisk gräns och måste ha ett spliceosomalt maskineri. Således var det möjligt att den tidigaste cellen var komplex och eukaryot. Överraskande nog visar fynd från omfattande jämförande genomikforskning från flera organismer sedan 2007 överväldigande att de tidigaste organismerna kunde ha varit mycket komplexa och eukaryota, och kunde ha innehållit komplexa proteiner, som förutspåtts av Senapathys teori.
Spliceosomen är en mycket komplex mekanism som innehåller ~200 proteiner och flera SnRNPs . Collins och Penny sa, "Vi börjar med hypotesen att ... spliceosomen har ökat i komplexitet under hela den eukaryotiska evolutionen. Undersökning av fördelningen av spliceosomala komponenter indikerar dock att det inte bara fanns en spliceosom i den eukaryota förfadern utan att den också innehöll de flesta av nyckelkomponenterna som finns i dagens eukaryoter. ... den sista gemensamma förfadern till bevarade eukaryoter verkar visa mycket av den molekylära komplexitet som vi ser idag." Detta tyder på att de tidigaste eukaryota organismerna var komplexa och innehöll sofistikerade gener och proteiner.
Bakteriegener
Gener med oavbrutna kodande sekvenser som är tusentals baser långa - upp till 90 000 baser - som förekommer i många bakteriella organismer var praktiskt taget omöjliga att ha inträffat. De bakteriella generna kunde dock ha sitt ursprung från splittrade gener genom att förlora introner, det enda föreslagna sättet att komma fram till långa kodande sekvenser. Det är också ett bättre [ förtydligande behövs ] sätt än att öka längden på ORF från korta slumpmässiga ORF till långa ORF genom att specifikt ta bort stoppkodonen genom mutation.
| Genstorlek (baser) | Antal gener |
|---|---|
| 5 000 - 10 000 | 3 029 |
| 10 000 - 15 000 | 492 |
| 15 000 - 20 000 | 131 |
| 20 000 - 25 000 | 39 |
| >25 000 | 41 |
| Långa kodande sekvenser förekommer som långa ORF i bakteriegener. Tusentals gener som är längre än 5 000 baser, som kodar för proteiner som är längre än 2 000 aminosyror, finns i många bakteriella genom. De längsta proteinkodande generna är ~90 000 baser långa. Var och en förekommer i en enda sträcka av kodande sekvens (ORF) utan stoppkodon eller introner . | |
Enligt teorin om delad gen kan denna process av intronförlust ha skett från prebiotiskt slumpmässigt DNA. Dessa kontinuerligt kodande gener skulle kunna vara tätt organiserade i bakteriegenomen utan några introner och vara mer strömlinjeformade. Enligt Senapathy skulle den nukleära gränsen som krävdes för en cell som innehåller delade gener inte krävas för en cell som bara innehåller oavbrutna gener. Bakteriecellerna utvecklade alltså ingen kärna. Baserat på teorin om delad gen kan de eukaryota genomen och bakteriegenomen oberoende av varandra ha sitt ursprung från de splittrade generna i primordiala slumpmässiga DNA-sekvenser.
Shapiro-Senapathy algoritm
Senapathy utvecklade algoritmer för att detektera donator- och acceptorsplitsningsställen, exoner och en fullständig delad gen i en genomisk sekvens. Han utvecklade positionsviktmatrismetoden (PWM) baserad på frekvensen av de fyra baserna vid konsensussekvenserna för donatorn och acceptorn i olika organismer för att identifiera splitsningsställena i en given sekvens. Vidare formulerade han den första algoritmen för att hitta exonerna baserat på kravet på exoner att innehålla en donatorsekvens (vid 5'-änden) och en acceptorsekvens (i 3'-änden), och en ORF där exonen ska förekomma , och en annan algoritm för att hitta en fullständig delad gen. Dessa algoritmer är gemensamt kända som Shapiro-Senapathy-algoritmen (S&S).
Denna algoritm hjälper till att identifiera splitsningsmutationer som orsakar sjukdomar och biverkningar av läkemedel. Forskare använde algoritmen för att identifiera mutationer och gener som orsakar cancer, ärftliga sjukdomar, immunbristsjukdomar och neurologiska störningar. Det används alltmer i klinisk praxis och forskning för att hitta mutationer i kända sjukdomsframkallande gener hos patienter och för att upptäcka nya gener som är orsaken till olika sjukdomar. Dessutom används det för att definiera de kryptiska splitsningsställena och härleda de mekanismer genom vilka mutationer kan påverka normal splitsning och leda till olika sjukdomar. Den används också i grundforskning.
Fynd baserade på S&S har påverkat viktiga frågor inom eukaryot biologi och inom humanmedicin.
Bekräftande bevis
Den delade genteorin antyder att strukturella egenskaper hos delade gener som förutsägs från datorsimulerade slumpmässiga sekvenser förekommer i eukaryota delade gener. Detta föds i de flesta kända splittrade gener. Sekvenserna uppvisar en nästan perfekt negativ exponentiell fördelning av ORF-längder. Med sällsynta undantag faller eukaryota genexoner inom det förväntade maximala 600 basen.
Teorin förutspår korrekt att exoner avgränsas av stoppkodon, speciellt vid 3'-ändarna av exoner. Egentligen är de exakt avgränsade starkare vid 3'-ändarna av exoner och mindre starkt vid 5'-ändarna i de flesta kända gener, som förutspåtts. Dessa stoppkodon är de viktigaste funktionella delarna av båda splitsningsförbindelserna. Teorin ger alltså en förklaring till de "konserverade" splitsningsövergångarna i ändarna av exoner och för förlusten av dessa stoppkodon tillsammans med introner när de splitsas ut. Teorin förutspår korrekt att splitsningsövergångar är slumpmässigt fördelade i eukaryota DNA-sekvenser. Teorin förutspår korrekt att splitsningsövergångar som finns i överförings-RNA-gener och ribosomala RNA-gener inte innehåller stoppkodon. Lariatsignalen, en annan sekvens involverad i splitsningsprocessen, innehåller också stoppkodon.
Teorin förutspår korrekt att introner är icke-kodande och att de för det mesta är icke-funktionella. Förutom vissa intronsekvenser inklusive donator- och acceptorsplitsningssignalsekvenser och grenpunktssekvenser, och möjligen intronsplitsningsförstärkare som förekommer i ändarna av introner, som hjälper till att avlägsna introner, saknar den stora majoriteten av introner alla funktioner. . Teorin utesluter inte sällsynta sekvenser inom introner som skulle kunna användas av genomet och cellen, särskilt eftersom intronerna är så långa.
Således bekräftas teorins förutsägelser exakt av huvudelementen i moderna eukaryota genom.
Jämförande analys av moderna genomdata från flera levande organismer fann att egenskaperna hos delade gener spåras tillbaka till de tidigaste organismerna. Dessa organismer kunde ha innehållit de splittrade gener och komplexa proteiner som förekommer i dagens levande organismer.
Studier som använder maximal sannolikhetsanalys fann att de tidigaste eukaryota organismerna innehöll samma gener som moderna organismer med ännu högre introndensitet. Jämförande genomik av många organismer inklusive basala eukaryoter (som anses vara primitiva eukaryota organismer som Amoeboflagellata, Diplomonadida och Parabasalia ) visade att intronrika splitgener åtföljde och spliceosomer från moderna organismer fanns närvarande i deras tidigaste föregångare, och med alla eukaryota cellulära komponenter.
Utvalda publikationer
- Shapiro, Marvin B.; Senapathy, Periannan (1987). "RNA-splitsningsförbindelser av olika klasser av eukaryoter: sekvensstatistik och funktionella implikationer i genuttryck" . Nukleinsyraforskning . 15 (17): 7155–7174. doi : 10.1093/nar/15.17.7155 . PMC 306199 . PMID 3658675 .
- Senapathy, P. (1988). "Möjlig utveckling av splice-junction-signaler i eukaryota gener från stoppkodon" . Proc Natl Acad Sci USA . 85 (4): 1129–33. Bibcode : 1988PNAS...85.1129S . doi : 10.1073/pnas.85.4.1129 . PMC 279719 . PMID 3422483 .
- Senapathy, P; Shapiro, MB; Harris, NL (1990). Splitskopplingar, förgreningspunkter och exoner: sekvensstatistik, identifiering och tillämpningar till genomprojekt . Metoder inom enzymologi . Vol. 183. s. 252–78 . doi : 10.1016/0076-6879(90)83018-5 . ISBN 9780121820848 . PMID 2314278 .
- Harris, NL; Senapathy, P. (1990). "Fördelning och konsensus av grenpunktssignaler i eukaryota gener: en datoriserad statistisk analys" . Nucleic Acids Res . 18 (10): 3015–9. doi : 10.1093/nar/18.10.3015 . PMC 330832 . PMID 2349097 .
- Senapathy, P. (1986). "Ursprunget till eukaryota introner: en hypotes, baserad på kodonfördelningsstatistik i gener, och dess implikationer" . Proc Natl Acad Sci USA . 83 (7): 2133–7. Bibcode : 1986PNAS...83.2133S . doi : 10.1073/pnas.83.7.2133 . PMC 323245 . PMID 3457379 .
- Regulapati, R.; Bhasi, A.; Singh, CK; Senapathy, P. (2008). "Ursprunget till den delade strukturen av spliceosomala gener från slumpmässiga genetiska sekvenser" . PLOS ETT . 3 (10): 10. Bibcode : 2008PLoSO...3.3456R . doi : 10.1371/journal.pone.0003456 . PMC 2565106 . PMID 18941625 .
- Senapathy, P. (1995). "Introner och ursprunget till proteinkodande gener" . Vetenskap . 268 (5215): 1366–7. Bibcode : 1995Sci...268.1366S . doi : 10.1126/science.7761858 . PMID 7761858 .
- ^ a b c d e f g h i j k l m n o p q Senapathy, P. (april 1986). "Ursprunget till eukaryota introner: en hypotes, baserad på kodonfördelningsstatistik i gener, och dess implikationer" . Proceedings of the National Academy of Sciences of the United States of America . 83 (7): 2133–2137. Bibcode : 1986PNAS...83.2133S . doi : 10.1073/pnas.83.7.2133 . ISSN 0027-8424 . PMC 323245 . PMID 3457379 .
- ^ a b c d e f g h i j k l m n o Senapathy, P. (februari 1982). "Möjlig utveckling av splice-junction-signaler i eukaryota gener från stoppkodon" . Proceedings of the National Academy of Sciences of the United States of America . 85 (4): 1129–1133. Bibcode : 1988PNAS...85.1129S . doi : 10.1073/pnas.85.4.1129 . ISSN 0027-8424 . PMC 279719 . PMID 3422483 .
- ^ a b c d e f g h i j Senapathy, P. (1995-06-02). "Introner och ursprunget till proteinkodande gener" . Vetenskap . 268 (5215): 1366–1367, författarsvar 1367–1369. Bibcode : 1995Sci...268.1366S . doi : 10.1126/science.7761858 . ISSN 0036-8075 . PMID 7761858 .
- ^ "Oberoende ursprung och fakta om livet" . wasdarwinwrong.com . Hämtad 2021-07-31 .
- ^ "Oberoende födelse av organismer. Periannan Senapathy. Bokrecension" . wasdarwinwrong.com . Hämtad 2021-07-31 .
-
^
Theobald, Douglas L. (2012). "29+ Evidences for Macroevolution: The Scientific Case for Common Descent" .
{{ citera journal }}: Citera journal kräver|journal=( hjälp ) - ^ Gillies, SD; Morrison, SL; Oi, VT; Tonegawa, S. (juni 1983). "Ett vävnadsspecifikt transkriptionsförstärkarelement är beläget i huvudintronet av en omarrangerad immunglobulin-tungkedjegen". Cell . 33 (3): 717–728. doi : 10.1016/0092-8674(83)90014-4 . ISSN 0092-8674 . PMID 6409417 . S2CID 40313833 .
- ^ Mercola, M.; Wang, XF; Olsen, J.; Calame, K. (1983-08-12). "Transkriptionella förstärkarelement i musimmunoglobulins tunga kedja". Vetenskap . 221 (4611): 663–665. Bibcode : 1983Sci...221..663M . doi : 10.1126/science.6306772 . ISSN 0036-8075 . PMID 6306772 .
- ^ Berk, AJ; Sharp, PA (november 1977). "Stormning och kartläggning av tidiga adenovirus-mRNA genom gelelektrofores av S1-endonukleas-digererade hybrider" . Cell . 12 (3): 721–732. doi : 10.1016/0092-8674(77)90272-0 . ISSN 0092-8674 . PMID 922889 .
- ^ Berget, SM; Moore, C; Sharp, PA (augusti 1977). "Splitsade segment vid 5'-terminalen av adenovirus 2 sent mRNA" . Proceedings of the National Academy of Sciences of the United States of America . 74 (8): 3171–3175. Bibcode : 1977PNAS...74.3171B . doi : 10.1073/pnas.74.8.3171 . ISSN 0027-8424 . PMC 431482 . PMID 269380 .
- ^ Chow, LT; Roberts, JM; Lewis, JB; Mäklare, TR (augusti 1977). "En karta över cytoplasmatiska RNA-transkript från lytiskt adenovirus typ 2, bestämt genom elektronmikroskopi av RNA:DNA-hybrider". Cell . 11 (4): 819–836. doi : 10.1016/0092-8674(77)90294-X . ISSN 0092-8674 . PMID 890740 . S2CID 37967144 .
- ^ "Online Education Kit: 1977: Introns Discovered" . National Human Genome Research Institute (NHGRI) . Hämtad 2019-01-01 .
- ^ Doolittle, W. Ford (13 april 1978). "Gener i bitar: har de någonsin varit tillsammans?" . Naturen . 272 (5654): 581–582. Bibcode : 1978Natur.272..581D . doi : 10.1038/272581a0 . ISSN 1476-4687 . S2CID 4162765 .
- ^ Darnell, JE (1978-12-22). "Implikationer av RNA-RNA-skarvning i utvecklingen av eukaryota celler". Vetenskap . 202 (4374): 1257–1260. doi : 10.1126/science.364651 . ISSN 0036-8075 . PMID 364651 .
- ^ Doolittle, WF; Darnell, JE (1986-03-01). "Spekulationer om evolutionens tidiga förlopp" . Proceedings of the National Academy of Sciences . 83 (5): 1271–1275. Bibcode : 1986PNAS...83.1271D . doi : 10.1073/pnas.83.5.1271 . ISSN 1091-6490 . PMC 323057 . PMID 2419905 .
- ^ a b c Blake, CCF (1985-01-01). Exoner och evolutionen av proteiner . International Review of Cytology . Vol. 93. s. 149–185. doi : 10.1016/S0074-7696(08)61374-1 . ISBN 9780123644930 . ISSN 0074-7696 . PMID 2409042 .
- ^ Gilbert, Walter (februari 1978). "Varför gener i bitar?" . Naturen . 271 (5645): 501. Bibcode : 1978Natur.271..501G . doi : 10.1038/271501a0 . ISSN 1476-4687 . PMID 622185 . S2CID 4216649 .
- ^ Tonegawa, S; Maxam, AM; Tizard, R; Bernard, O; Gilbert, W (mars 1978). "Sekvens av en musgroddlinjegen för en variabel region av en lätt immunglobulinkedja" . Proceedings of the National Academy of Sciences of the United States of America . 75 (3): 1485–1489. Bibcode : 1978PNAS...75.1485T . doi : 10.1073/pnas.75.3.1485 . ISSN 0027-8424 . PMC 411497 . PMID 418414 .
- ^ Feng, DF; Doolittle, RF (1987-01-01). "Rekonstruera utvecklingen av koagulation av ryggradsdjurs blod från ett övervägande av aminosyrasekvenserna av koaguleringsproteiner". Cold Spring Harbor Symposia on Quantitative Biology . 52 : 869-874. doi : 10.1101/SQB.1987.052.01.095 . ISSN 1943-4456 . PMID 3483343 .
- ^ Gibbons, A. (1990-12-07). "Beräkna den ursprungliga familjen - av exoner". Vetenskap . 250 (4986): 1342. Bibcode : 1990Sci...250.1342G . doi : 10.1126/science.1701567 . ISSN 1095-9203 . PMID 1701567 .
- ^ a b c Reva, Oleg; Tümmler, Burkhard (2008). "Tänk stort – jättegener i bakterier" (PDF) . Miljömikrobiologi . 10 (3): 768–777. doi : 10.1111/j.1462-2920.2007.01500.x . hdl : 2263/9009 . ISSN 1462-2920 . PMID 18237309 .
- ^ a b c d e f g Regulapati, Rahul; Singh, Chandan Kumar; Bhasi, Ashwini; Senapathy, Periannan (2008-10-20). "Ursprunget till den delade strukturen av spliceosomala gener från slumpmässiga genetiska sekvenser" . PLOS ETT . 3 (10): e3456. Bibcode : 2008PLoSO...3.3456R . doi : 10.1371/journal.pone.0003456 . ISSN 1932-6203 . PMC 2565106 . PMID 18941625 .
-
^
Information, Reed Business (1986-06-26). Ny vetenskapsman . Reed affärsinformation.
{{ citera bok }}:|first=har ett generiskt namn ( hjälp ) -
^
Information, Reed Business (1988-03-31). Ny vetenskapsman . Reed affärsinformation.
{{ citera bok }}:|first=har ett generiskt namn ( hjälp ) - ^ a b c d Senapathy, Periannan; Harris, Nomi L. (1990-05-25). "Fördelning och konsensus av grenpunktssignaler i eukaryota gener: en datoriserad statistisk analys" . Nukleinsyraforskning . 18 (10): 3015–9. doi : 10.1093/nar/18.10.3015 . ISSN 0305-1048 . PMC 330832 . PMID 2349097 .
- ^ Maier, U.-G.; Brown, JWS; Toloczyki, C.; Feix, G. (januari 1987). "Bindning av en nukleär faktor till en konsensussekvens i den 5'-flankerande regionen av zeingener från majs" . EMBO Journal . 6 (1): 17–22. doi : 10.1002/j.1460-2075.1987.tb04712.x . ISSN 0261-4189 . PMC 553350 . PMID 15981330 .
- ^ Keller, EB; Noon, WA (1985-07-11). "Intronsplitsning: en konserverad intern signal i introner av Drosophila pre-mRNAs" . Nukleinsyraforskning . 13 (13): 4971–4981. doi : 10.1093/nar/13.13.4971 . ISSN 0305-1048 . PMC 321838 . PMID 2410858 .
- ^ BIRNSTIEL, M; BUSSLINGER, M; STRUB, K (juni 1985). "Transkriptionsuppsägning och 3′-bearbetning: slutet är på plats!". Cell . 41 (2): 349–359. doi : 10.1016/s0092-8674(85)80007-6 . ISSN 0092-8674 . PMID 2580642 . S2CID 11999043 .
- ^ Konsortium, Internationell mänsklig genomsekvensering (februari 2001). "Initial sekvensering och analys av det mänskliga genomet" . Naturen . 409 (6822): 860–921. Bibcode : 2001Natur.409..860L . doi : 10.1038/35057062 . ISSN 1476-4687 . PMID 11237011 .
- ^ Zhu, Xiaohong; Zandieh, Ali; Xia, Ashley; Wu, Mitchell; Wu, David; Wen, Meiyuan; Wang, Mei; Venter, Eli; Turner, Russell (2001-02-16). "Sekvensen av det mänskliga genomet" . Vetenskap . 291 (5507): 1304–1351. Bibcode : 2001Sci...291.1304V . doi : 10.1126/science.1058040 . ISSN 1095-9203 . PMID 11181995 .
- ^ Kang, Byoung-Cheorl; Nä, Gyoungju; Lee, Heung-Ryul; Han, Koeun; Purushotham, Preethi M.; Jo, Jinkwan (2017). "Utveckling av en genetisk karta för lök (Allium cepa L.) med hjälp av referensfri genotypning genom sekvensering och SNP-analyser" . Frontiers in Plant Science . 8 : 1606. doi : 10.3389/fpls.2017.01606 . ISSN 1664-462X . PMC 5604068 . PMID 28959273 .
- ^ Smith, Jeramiah J.; Voss, S. Randal; Tsonis, Panagiotis A.; Timoshevskaya, Nataliya Y.; Timoshevskiy, Vladimir A.; Keinath, Melissa C. (2015-11-10). "Initial karakterisering av det stora genomet hos salamandern Ambystoma mexicanum med hjälp av hagelgevär och laserfångande kromosomsekvensering" . Vetenskapliga rapporter . 5 : 16413. Bibcode : 2015NatSR...516413K . doi : 10.1038/srep16413 . ISSN 2045-2322 . PMC 4639759 . PMID 26553646 .
- ^ Venter, JC; Adams, MD; Myers, EW; Li, PW; Mural, RJ; Sutton, GG; Smith, HO; Yandell, M.; Evans, CA (2001-02-16). "Sekvensen av det mänskliga genomet" . Vetenskap . 291 (5507): 1304–1351. Bibcode : 2001Sci...291.1304V . doi : 10.1126/science.1058040 . ISSN 0036-8075 . PMID 11181995 .
- ^ Lander, ES; Linton, LM; Birren, B.; Nusbaum, C.; Zody, MC; Baldwin, J.; Devon, K.; Dewar, K.; Doyle, M. (2001-02-15). "Initial sekvensering och analys av det mänskliga genomet" (PDF) . Naturen . 409 (6822): 860–921. Bibcode : 2001Natur.409..860L . doi : 10.1038/35057062 . ISSN 0028-0836 . PMID 11237011 .
- ^ Consortium*, The C. elegans Sequencing (1998-12-11). "Genome Sequence of the Nematode C. elegans: A Platform for Investigating Biology". Vetenskap . 282 (5396): 2012–2018. Bibcode : 1998Sci...282.2012. . doi : 10.1126/science.282.5396.2012 . ISSN 1095-9203 . PMID 9851916 .
- ^ Arabidopsis Genome Initiative (2000-12-14). "Analys av genomsekvensen för den blommande växten Arabidopsis thaliana" . Naturen . 408 (6814): 796–815. Bibcode : 2000Natur.408..796T . doi : 10.1038/35048692 . ISSN 0028-0836 . PMID 11130711 .
- ^ Bennetzen, Jeffrey L.; Brown, James KM; Devos, Katrien M. (2002-07-01). "Genomstorleksminskning genom olaglig rekombination motverkar genomexpansion i Arabidopsis" . Genomforskning . 12 (7): 1075–1079. doi : 10.1101/gr.132102 . ISSN 1549-5469 . PMC 186626 . PMID 12097344 .
- ^ Kurland, CG; Canbäck, B.; Berg, OG (december 2007). "Ursprunget till moderna proteomer". Biochimie . 89 (12): 1454–1463. doi : 10.1016/j.biochi.2007.09.004 . ISSN 0300-9084 . PMID 17949885 .
- ^ Caetano-Anollés, Gustavo; Caetano-Anollés, Derek (juli 2003). "Ett evolutionärt strukturerat universum av proteinarkitektur" . Genomforskning . 13 (7): 1563–1571. doi : 10.1101/gr.1161903 . ISSN 1088-9051 . PMC 403752 . PMID 12840035 .
- ^ Glansdorff, Nicolas; Xu, Ying; Labedan, Bernard (2008-07-09). "Den sista universella gemensamma förfadern: uppkomst, konstitution och genetiskt arv av en svårfångad föregångare" . Biologi direkt . 3 : 29. doi : 10.1186/1745-6150-3-29 . ISSN 1745-6150 . PMC 2478661 . PMID 18613974 .
- ^ Kurland, CG; Collins, LJ; Penny, D. (2006-05-19). "Genomik och den irreducibla naturen hos eukaryota celler". Vetenskap . 312 (5776): 1011–1014. Bibcode : 2006Sci...312.1011K . doi : 10.1126/science.1121674 . ISSN 1095-9203 . PMID 16709776 . S2CID 30768101 .
- ^ Collins, Lesley; Penny, David (april 2005). "Komplex spliceosomal organisation som är ursprunglig till existerande eukaryoter" . Molekylärbiologi och evolution . 22 (4): 1053–1066. doi : 10.1093/molbev/msi091 . ISSN 0737-4038 . PMID 15659557 .
- ^ Penny, David; Collins, Lesley J.; Daly, Toni K.; Cox, Simon J. (december 2014). "De relativa åldrarna för eukaryoter och akaryoter". Journal of Molecular Evolution . 79 (5–6): 228–239. Bibcode : 2014JMolE..79..228P . doi : 10.1007/s00239-014-9643-y . ISSN 1432-1432 . PMID 25179144 . S2CID 17512331 .
- ^ Fuerst, John A.; Sagulenko, Evgeny (2012-05-04). "Nycklar till eukaryality: Planctomycetes and Ancestral Evolution of Cellular Complexity" . Frontiers in Microbiology . 3 : 167. doi : 10.3389/fmicb.2012.00167 . ISSN 1664-302X . PMC 3343278 . PMID 22586422 .
- ^ Collins, Lesley; Penny, David (april 2005). "Komplex spliceosomal organisation som är ursprunglig till existerande eukaryoter" . Molekylärbiologi och evolution . 22 (4): 1053–1066. doi : 10.1093/molbev/msi091 . ISSN 0737-4038 . PMID 15659557 . [ verifiering behövs ]
- ^ a b c d Shapiro, MB; Senapathy, P. (1987-09-11). "RNA-splitsningsförbindelser av olika klasser av eukaryoter: sekvensstatistik och funktionella implikationer i genuttryck" . Nukleinsyraforskning . 15 (17): 7155–7174. doi : 10.1093/nar/15.17.7155 . ISSN 0305-1048 . PMC 306199 . PMID 3658675 .
- ^ a b c d Senapathy, P.; Shapiro, MB; Harris, NL (1990). Splitskopplingar, förgreningspunkter och exoner: sekvensstatistik, identifiering och tillämpningar till genomprojekt . Metoder inom enzymologi . Vol. 183. s. 252–278. doi : 10.1016/0076-6879(90)83018-5 . ISBN 9780121820848 . ISSN 0076-6879 . PMID 2314278 .
- ^ "National Institutes of Health (NIH) - Alla av oss" . allofus.nih.gov . Hämtad 2019-01-02 .
- ^ a b Penny, David; Collins, Lesley (2005-04-01). "Komplex spliceosomal organisation förfäder till existerande eukaryoter" . Molekylärbiologi och evolution . 22 (4): 1053–1066. doi : 10.1093/molbev/msi091 . ISSN 0737-4038 . PMID 15659557 .
- ^ Caetano-Anollés, Derek; Caetano-Anollés, Gustavo (2003-07-01). "Ett evolutionärt strukturerat universum av proteinarkitektur" . Genomforskning . 13 (7): 1563–1571. doi : 10.1101/gr.1161903 . ISSN 1549-5469 . PMC 403752 . PMID 12840035 .
- ^ Glansdorff, Nicolas; Xu, Ying; Labedan, Bernard (2008-07-09). "Den sista universella gemensamma förfädern: uppkomsten, konstitutionen och det genetiska arvet efter en svårfångad föregångare" . Biologi direkt . 3 (1): 29. doi : 10.1186/1745-6150-3-29 . ISSN 1745-6150 . PMC 2478661 . PMID 18613974 .
- ^ Kurland, CG; Canbäck, B.; Berg, OG (2007-12-01). "Ursprunget till moderna proteomer". Biochimie . 89 (12): 1454–1463. doi : 10.1016/j.biochi.2007.09.004 . ISSN 0300-9084 . PMID 17949885 .
- ^ Penny, D.; Collins, LJ; Kurland, CG (2006-05-19). "Genomik och den irreducerbara naturen hos eukaryota celler". Vetenskap . 312 (5776): 1011–1014. Bibcode : 2006Sci...312.1011K . doi : 10.1126/science.1121674 . ISSN 1095-9203 . PMID 16709776 . S2CID 30768101 .
- ^ Poole, AM; Jeffares, DC; Penny, D. (januari 1998). "Vägen från RNA-världen". Journal of Molecular Evolution . 46 (1): 1–17. Bibcode : 1998JMolE..46....1P . doi : 10.1007/PL00006275 . ISSN 0022-2844 . PMID 9419221 . S2CID 17968659 .
- ^ Forterre, Patrick; Philippe, Hervé (1999). "Var är roten till livets universella träd?". BioEssays . 21 (10): 871–879. doi : 10.1002/(SICI)1521-1878(199910)21:10<871::AID-BIES10>3.0.CO;2-Q . ISSN 1521-1878 . PMID 10497338 .
- ^ Cox, Simon J.; Daly, Toni K.; Collins, Lesley J.; Penny, David (2014-12-01). "Eukaryoternas och akaryoternas relativa åldrar". Journal of Molecular Evolution . 79 (5–6): 228–239. Bibcode : 2014JMolE..79..228P . doi : 10.1007/s00239-014-9643-y . ISSN 1432-1432 . PMID 25179144 . S2CID 17512331 .
- ^ Sagulenko, Evgeny; Fuerst, John Arlington (2012). "Nycklar till eukaryalitet: planctomycetes och förfäders utveckling av cellulär komplexitet" . Frontiers in Microbiology . 3 : 167. doi : 10.3389/fmicb.2012.00167 . ISSN 1664-302X . PMC 3343278 . PMID 22586422 .
- ^ a b Gilbert, Walter; Roy, Scott W. (2005-02-08). "Komplexa tidiga gener" . Proceedings of the National Academy of Sciences . 102 (6): 1986–1991. Bibcode : 2005PNAS..102.1986R . doi : 10.1073/pnas.0408355101 . ISSN 1091-6490 . PMC 548548 . PMID 15687506 .
- ^ "Introduktion till de basala eukaryoterna" . ucmp.berkeley.edu . Hämtad 2021-08-01 .
- ^ Gilbert, Walter; Roy, Scott William (mars 2006). "Utvecklingen av spliceosomala introner: mönster, pussel och framsteg". Naturrecensioner Genetik . 7 (3): 211–221. doi : 10.1038/nrg1807 . ISSN 1471-0064 . PMID 16485020 . S2CID 33672491 .
- ^ Rogozin, Igor B.; Sverdlov, Alexander V.; Babenko, Vladimir N.; Koonin, Eugene V. (juni 2005). "Analys av evolutionen av exon-intronstrukturen av eukaryota gener" . Genomgångar i bioinformatik . 6 (2): 118–134. doi : 10.1093/bib/6.2.118 . ISSN 1467-5463 . PMID 15975222 .
- ^ Sullivan, James C.; Reitzel, Adam M.; Finnerty, John R. (2006). "En hög andel introner i mänskliga gener var närvarande tidigt i djurets evolution: bevis från den basala metazoanen Nematostella vectensis". Genominformatik. Internationell konferens om genominformatik . 17 (1): 219–229. ISSN 0919-9454 . PMID 17503371 .
- ^ Koonin, Eugene V.; Rogozin, Igor B.; Csuros, Miklos (2011-09-15). "En detaljerad historia av intronrika eukaryota förfäder härledd från en global undersökning av 100 kompletta genom" . PLOS Computational Biology . 7 (9): e1002150. Bibcode : 2011PLSCB...7E2150C . doi : 10.1371/journal.pcbi.1002150 . ISSN 1553-7358 . PMC 3174169 . PMID 21935348 .