Sekvensmontering
Inom bioinformatik hänvisar sekvensmontering till att ställa in och sammanfoga fragment från en längre DNA- sekvens för att rekonstruera den ursprungliga sekvensen . Detta behövs eftersom DNA-sekvenseringsteknik kanske inte kan "läsa" hela genom på en gång, utan snarare läser små bitar på mellan 20 och 30 000 baser, beroende på vilken teknik som används. Vanligtvis resulterar de korta fragmenten (avläsningarna) från hagelgevärssekvensering av genomiskt DNA, eller gentranskript ( EST ).
Problemet med sekvensmontering kan jämföras med att ta många kopior av en bok, föra var och en av dem genom en dokumentförstörare med en annan skärare, och plocka ihop bokens text bara genom att titta på de strimlade bitarna. Förutom den uppenbara svårigheten med denna uppgift, finns det några extra praktiska problem: originalet kan ha många upprepade stycken, och vissa strimlor kan modifieras under fragmenteringen för att ha stavfel. Utdrag ur en annan bok kan också läggas till, och vissa strimlor kan vara helt oigenkännliga.
Genomsamlare
De första sekvensmontörerna började dyka upp i slutet av 1980-talet och början av 1990-talet som varianter av enklare sekvensanpassningsprogram för att sätta ihop stora mängder fragment genererade av automatiserade sekvenseringsinstrument som kallas DNA-sekvenserare . När de sekvenserade organismerna växte i storlek och komplexitet (från små virus över plasmider till bakterier och slutligen eukaryoter ), behövde monteringsprogrammen som användes i dessa genomprojekt allt mer sofistikerade strategier för att hantera:
- terabyte av sekvenseringsdata som behöver bearbetas på datorkluster ;
- identiska och nästan identiska sekvenser (så kallade upprepningar ) som i värsta fall kan öka tids- och rymdkomplexiteten hos algoritmer kvadratiskt;
- DNA-läser fel i fragmenten från sekvenseringsinstrumenten, vilket kan förvirra sammansättningen.
Inför utmaningen att sammanställa de första större eukaryota genomen – fruktflugan Drosophila melanogaster år 2000 och det mänskliga genomet bara ett år senare – utvecklade forskare assemblers som Celera Assembler och Arachne som kan hantera genom på 130 miljoner (t.ex. fruktflugan). D. melanogaster ) till 3 miljarder (t.ex. det mänskliga genomet) baspar. Efter dessa ansträngningar byggde flera andra grupper, mestadels vid de stora genomsekvenseringscentren, storskaliga assemblers, och en öppen källkodssatsning känd som AMOS lanserades för att samla alla innovationer inom genomsammansättningsteknologi under ramverket med öppen källkod .

EST montörer
Uttryckt sekvenstagg eller EST-sammansättning var en tidig strategi, från mitten av 1990-talet till mitten av 2000-talet, för att sätta ihop individuella gener snarare än hela genom. Problemet skiljer sig från genomsammansättning på flera sätt. Ingångssekvenserna för EST-sammansättning är fragment av det transkriberade mRNA från en cell och representerar endast en undergrupp av hela genomet. Ett antal algoritmiska problem skiljer sig mellan genom och EST-sammansättning. Till exempel har genom ofta stora mängder repetitiva sekvenser, koncentrerade i de intergena regionerna. Transkriberade gener innehåller många färre upprepningar, vilket gör monteringen något lättare. Å andra sidan uttrycks (transkriberas) vissa gener i mycket högt antal (t.ex. hushållningsgener ), vilket innebär att till skillnad från helgenoms hagelgevärssekvensering, tas läsningarna inte enhetligt över genomet.
EST-sammansättning görs mycket mer komplicerad av funktioner som (cis-) alternativ splitsning , trans-splitsning , enkelnukleotidpolymorfism och post-transkriptionell modifiering . Från och med 2008 när RNA-Seq uppfanns, ersattes EST-sekvensering av denna mycket effektivare teknologi, beskriven under de novo transkriptommontering .
Typer av sekvensmontering
Det finns tre sätt att sammanställa sekvenseringsdata:
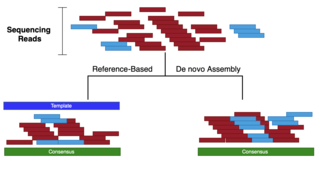
- De-novo: sammansättning av sekvensläsningar för att skapa sekvenser i full längd (ibland nya) utan att använda en mall (se de novo sekvensmontörer , de novo transkriptomsammansättning )
- Mappning/justering: sammansättning av läsningar genom att justera läsningar mot en mall (AKA-referens). Den samlade konsensusen kanske inte är identisk med mallen.
- Referensstyrd: gruppering av läsningar efter likhet med den mest lika regionen inom referensen (stegvis kartläggning). Läsningar inom varje grupp förkortas sedan för att efterlikna kvaliteten på korta läsningar. En typisk metod för att göra det är k-mer- metoden. Referensstyrd montering är mest användbar med långa läsningar .
Refererad-styrd montering är en kombination av de andra typerna. Denna typ tillämpas på långa läsningar för att efterlikna fördelar med korta läsningar (dvs. samtalskvalitet). Logiken bakom det är att gruppera läsningarna efter mindre fönster inom referensen. Läsningar i varje grupp kommer sedan att minskas i storlek med hjälp av k-mere-metoden för att välja högsta kvalitet och mest sannolika sammanhängande (contig). Contigs kommer sedan att fogas samman för att skapa en ställning. Den slutliga överenskommelsen görs genom att täppa till eventuella luckor i ställningen.
De-novo vs. kartläggning
När det gäller komplexitet och tidskrav är de-novo-sammansättningar storleksordningar långsammare och mer minneskrävande än kartläggningssammansättningar. Detta beror mest på det faktum att assemblyalgoritmen behöver jämföra varje läsning med varannan läsning (en operation som har en naiv tidskomplexitet på O( n 2 )). Nuvarande de-novo genomsamlare kan använda olika typer av grafbaserade algoritmer, såsom:
- Överlappning/Layout/Konsensus (OLC) tillvägagångssätt, som var typiskt för Sanger-datasamlare och bygger på en överlappningsgraf.
- de Bruijn Graph (DBG) tillvägagångssätt, som används mest för de korta läsningarna från Solexa- och SOLiD-plattformarna. Den förlitar sig på K-mer-grafer, som fungerar bra med stora mängder korta läsningar.
- Girig grafbaserad metod, som också kan använda en av OLC- eller DBG-metoderna. Med giriga grafbaserade algoritmer växer kontigerna [ ytterligare förklaring behövs ] genom girig förlängning, och tar alltid på sig den läsning som hittas genom att följa den överlappning med högst poäng.
Med hänvisning till jämförelsen som dragits med strimlade böcker i inledningen: medan man för kartläggning av sammanställningar skulle ha en mycket liknande bok som mall (kanske med namnen på huvudkaraktärerna och några platser ändrade), är de-novo sammanställningar en mer skrämmande utmaning i att man inte i förväg skulle veta om detta skulle bli en vetenskapsbok, en roman, en katalog eller ens flera böcker. Dessutom skulle varje strimla jämföras med varannan strimla.
Att hantera upprepningar i de-novo montering kräver konstruktion av en graf som representerar angränsande upprepningar. Sådan information kan härledas från att läsa ett långt fragment som täcker upprepningarna i sin helhet eller endast dess två ändar . Å andra sidan, i en kartläggning, lämnas vanligtvis delar med flera eller inga matchningar för en annan monteringsteknik att titta närmare på.
Sekvensmonteringspipeline (bioinformatik)
I allmänhet finns det tre steg för att montera sekvensläsningar till en ställning:
1) Förmontering: detta steg är väsentligt för att säkerställa integriteten för downline-analys, såsom variantanrop eller slutlig ställningssekvens. Detta steg består av två kronologiska arbetsflöden:
A) Kvalitetskontroll: Beroende på typen av sekvenseringsteknik kan olika fel uppstå som skulle leda till ett falskt basanrop . Till exempel kan sekvensering av "NAAAAAAAAAAAAN" och "NAAAAAAAAAAAAN" som inkluderar 12 adenin felaktigt kallas med 11 adenin istället. Sekvensering av ett mycket repetitivt segment av mål-DNA/RNA kan resultera i ett samtal som är en kort eller en bas till. Läskvalitet mäts vanligtvis av Phred som är en kodad poäng för varje nukleotidkvalitet inom en läsningssekvens. Vissa sekvenseringstekniker som PacBio har ingen poängmetod för deras sekvenserade läsningar. Ett vanligt verktyg som används i detta steg är FastQC.
B) Filtrering av läsningar: Läsningar som inte klarade kvalitetskontrollen bör tas bort från FastQ-filen för att få de bästa sammansättningarna.
2) Montering: under detta steg kommer läsjustering att användas med olika kriterier för att mappa varje läsning till den möjliga platsen. Den förutsagda positionen för en läsning baseras på antingen hur mycket av dess sekvens som är i linje med andra läsningar eller en referens. Olika inriktningsalgoritmer används för läsningar från olika sekvenseringsteknologier. Några av de vanligaste tillvägagångssätten i sammanställningen är de Bruijn -graf och överlappning. Läslängd, täckning , kvalitet och sekvenseringstekniken som används spelar en viktig roll för att välja den bästa inriktningsalgoritmen i fallet med Next Generation Sequencing . Å andra sidan kräver algoritmer som anpassar 3:e generationens sekvensläsningar förhandsstrategier för att ta hänsyn till den höga felfrekvensen som är förknippad med dem.
3) Efter montering: Detta steg fokuserar på att extrahera värdefull information från den sammansatta sekvensen. Jämförande genomik och populationsanalys är exempel på analys efter montering.
Påverkan av tekniska förändringar
Komplexiteten i sekvenssammansättningen drivs av två huvudfaktorer: antalet fragment och deras längder. Medan fler och längre fragment tillåter bättre identifiering av sekvensöverlappningar, utgör de också problem eftersom de underliggande algoritmerna visar kvadratiskt eller till och med exponentiellt komplexitetsbeteende för både antalet fragment och deras längd. Och även om kortare sekvenser är snabbare att justera, komplicerar de också layoutfasen för en sammanställning eftersom kortare läsningar är svårare att använda med upprepningar eller nästan identiska upprepningar.
Under de tidigaste dagarna av DNA-sekvensering kunde forskare bara få några få sekvenser av kort längd (några dussin baser) efter veckors arbete i laboratorier. Följaktligen kunde dessa sekvenser inriktas på några minuter för hand.
1975 uppfanns dideoxytermineringsmetoden ( AKA Sanger Sequencing ) och fram till strax efter 2000 förbättrades tekniken till en punkt där helautomatiska maskiner kunde churna ut sekvenser i ett mycket parallelliserat läge 24 timmar om dygnet. Stora genomcentra runt om i världen inrymde kompletta gårdar av dessa sekvenseringsmaskiner, vilket i sin tur ledde till att assemblerare måste optimeras för sekvenser från helgenoms hagelgevärssekvenseringsprojekt där läsningarna
- är cirka 800–900 baser långa
- innehåller sekvenseringsartefakter som sekvenserings- och kloningsvektorer
- har felfrekvenser mellan 0,5 och 10 %
Med Sanger-tekniken kunde bakterieprojekt med 20 000 till 200 000 avläsningar enkelt sättas ihop på en dator. Större projekt, som det mänskliga genomet med cirka 35 miljoner läsningar, behövde stora datorgårdar och distribuerad datoranvändning.
År 2004/2005 hade 454 Life Sciences pyrosequencing blivit kommersiellt lönsam . Denna nya genererade sekvenseringsmetod läser mycket kortare än de för Sanger-sekvensering: initialt cirka 100 baser, nu 400-500 baser. Dess mycket högre genomströmning och lägre kostnad (jämfört med Sanger-sekvensering) gjorde att genomcentren tog in denna teknologi, vilket i sin tur drev utvecklingen av sekvenssammanställare som effektivt kunde hantera lässeten. Den stora mängden data i kombination med teknikspecifika felmönster i läsningarna försenade utvecklingen av assemblers; i början av 2004 var endast Newbler assembler från 454 tillgänglig. Släppt i mitten av 2007, hybridversionen av MIRA assembler av Chevreux et al. var den första fritt tillgängliga montören som kunde montera 454 läsningar samt blandningar av 454 läsningar och Sanger-läsningar. Att sätta ihop sekvenser från olika sekvenseringsteknologier myntades därefter hybridmontering .
Från 2006 har Illumina -tekniken (tidigare Solexa) varit tillgänglig och kan generera cirka 100 miljoner avläsningar per körning på en enda sekvenseringsmaskin. Jämför detta med de 35 miljoner läsningarna av det mänskliga genomprojektet som behövde flera år för att produceras på hundratals sekvenseringsmaskiner. Illumina var initialt begränsad till en längd av endast 36 baser, vilket gjorde den mindre lämplig för de novo-sammansättning (som de novo-transkriptom-sammansättning ), men nyare iterationer av teknologin uppnår läslängder över 100 baser från båda ändarna av en 3-400bp klon . Tillkännagav i slutet av 2007, SHARCGS assembler av Dohm et al. var den första publicerade assembler som användes för en sammanställning med Solexa-läsningar. Den följdes snabbt av ett antal andra.
släpptes nya teknologier som SOLiD från Applied Biosystems , Ion Torrent och SMRT och nya teknologier (t.ex. Nanopore-sekvensering) fortsätter att dyka upp. Trots de högre felfrekvenserna för dessa tekniker är de viktiga för montering eftersom deras längre läslängd hjälper till att lösa upprepningsproblemet. Det är omöjligt att montera genom en perfekt upprepning som är längre än den maximala läslängden; Men när läsningarna blir längre blir chansen för en perfekt upprepning som stor liten. Detta ger längre sekvensläsningar en fördel vid sammansättning av repetitioner även om de har låg noggrannhet (~85%).
Monteringsalgoritmer
Olika organismer har en distinkt region med högre komplexitet inom deras genom. Därför behövs olika beräkningsmetoder. Några av de vanligaste algoritmerna är:
- Graph Assembly: bygger på grafteori inom datavetenskap. De Bruijn-grafen är ett exempel på detta tillvägagångssätt och använder k-mers för att sammanställa en sammanhängande av läsningar.
- Greedy Graph Assembly: denna metodpoäng som var och en läggs till i sammansättningen och väljer högsta möjliga poäng från den överlappande regionen.
Givet en uppsättning sekvensfragment är syftet att hitta en längre sekvens som innehåller alla fragmenten (se figuren under Typer av sekvenssammansättning) :
- Beräkna parvisa anpassningar av alla fragment.
- Välj två fragment med den största överlappningen.
- Slå samman valda fragment.
- Upprepa steg 2 och 3 tills endast ett fragment är kvar.
Resultatet kanske inte är en optimal lösning på problemet.
Program
För en lista över de-novo- montörer, se De novo-sekvensmontörer . För en lista över mappningslinjer, se Lista över sekvensanpassningsprogram § Kortläst sekvensjustering .
Några av de vanliga verktygen som används i olika monteringssteg listas i följande tabell:
| programvara | Läs typ | Verktyg webbsida | Anteckningar |
|---|---|---|---|
| FastQC | Flera olika | https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ | Detta är ett vanligt verktyg som används för att kontrollera läskvaliteten från olika sekvenseringsteknologier som Illumina , 454 och PacBio . |
| BWA | Kort & lång läsning | https://sourceforge.net/projects/bio-bwa/files/ | Detta är ett kommandoradsverktyg . Mest känd för lätt körning och exakt sekvensuppriktning. |
| Minikarta 2 | Långa läsningar | https://github.com/lh3/minimap2 | Detta kommandoradsverktyg är designat för att hantera PacBio & Oxford Nanopore och läser med 15 % felfrekvens. |
| LoReTTA | Långa läsningar | https://github.com/salvocamiolo/LoReTTA/releases/tag/v0.1 | Detta verktyg är utformat för att sätta ihop (referensstyrda) virala genom med större noggrannhet med hjälp av PacBio CCS-läsningar. |
| Spader | Kort & lång läsning | http://cab.spbu.ru/software/spades/ | Detta är ett monteringsverktyg som körs på kommandoraden. |
| Samtools | Uppriktningsanalys | https://samtools.github.io | Detta är användbart efter montering. Den kan generera olika statistik och utföra flera filtreringssteg till justeringsfilen. |
Se även
- De novo sekvensmontörer
- Sekvensjustering
- De novo transkriptomsammansättning
- Ställskyddsproblem
- Lista över sekvenserade djurgenom