Proteashämmare (biologi)
Inom biologi och biokemi är proteashämmare , eller antiproteaser , molekyler som hämmar funktionen av proteaser ( enzymer som hjälper till med nedbrytningen av proteiner ) . Många naturligt förekommande proteashämmare är proteiner .
Inom medicin används proteashämmare ofta omväxlande med alfa 1-antitrypsin (A1AT, som av denna anledning förkortas PI). A1AT är verkligen den proteashämmare som oftast är involverad i sjukdomar, nämligen vid alfa-1-antitrypsinbrist .
Klassificering
Proteashämmare kan klassificeras antingen efter vilken typ av proteas de hämmar eller efter deras verkningsmekanism. 2004 introducerade Rawlings och kollegor en klassificering av proteashämmare baserad på likheter som kan detekteras på nivån av aminosyrasekvens. Denna klassificering identifierade initialt 48 familjer av inhibitorer som kunde grupperas i 26 relaterade superfamiljer (eller klaner) efter deras struktur. Enligt MEROPS-databasen finns det nu 81 familjer av inhibitorer. Dessa familjer är namngivna med ett I följt av ett nummer, till exempel innehåller I14 hirudinliknande hämmare.
Genom proteas
Klasser av proteaser är:
- Asparaginsyraproteashämmare
- Cysteinproteashämmare
- Metalloproteashämmare
- Serinproteashämmare
- Treoninproteashämmare
- Trypsinhämmare
Genom mekanism
Klasser av hämmande verkningsmekanismer är:
- Självmordshämmare
- Övergångstillståndshämmare
- Proteinproteashämmare (se serpiner )
- Kelateringsmedel
Familjer
Inhibitor I4
Detta är en familj av proteas- självmordshämmare som kallas serpiner . Den innehåller hämmare av flera cystein- och serinproteasfamiljer. Deras verkningsmekanism bygger på att de genomgår en stor konformationsförändring som inaktiverar deras måls katalytiska triad .
Inhibitor I9
| Peptidashämmare I9 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
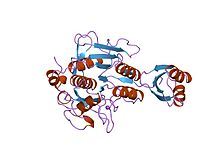 subtilisin bpn' prosegment (77 rester) komplexbundet med en mutant subtilisin bpn' (266 rester). kristall ph 4,6. kristallisationstemperatur 20 c diffraktionstemperatur-160 c
| |||||||||
| Identifierare | |||||||||
| Symbol | Inhibitor_I9 | ||||||||
| Pfam | PF05922 | ||||||||
| InterPro | IPR010259 | ||||||||
| MEROPS | I9 | ||||||||
| SCOP2 | 1gns / SCOPe / SUPFAM | ||||||||
| |||||||||
Proteinaspropeptidinhibitorer (ibland kallade aktiveringspeptider) är ansvariga för moduleringen av veckningen och aktiviteten av peptidasproenzymet eller zymogenet . Pro-segmentet dockar in i enzymet och skyddar substratets bindningsställe och främjar därigenom hämning av enzymet. Flera sådana propeptider delar en liknande topologi, trots ofta låga sekvensidentiteter . Propeptidregionen har en antiparallell-alfa/antiparallell-beta-veckning med öppen sandwich, med två alfa-helixar och fyra beta-strängar med en (beta/alfa/beta)x2-topologi. Peptidasinhibitor I9-familjen innehåller propeptiddomänen vid N-terminalen av peptidaser som tillhör MEROPS-familjen S8A, subtilisiner . Propeptiden avlägsnas genom proteolytisk klyvning; avlägsnande aktiverar enzymet.
Inhibitor I10
| Serinendopeptidashämmare | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 lösningsstruktur av marinostatin, en proteashämmare, innehållande två esterbindningar.
| |||||||||
| Identifierare | |||||||||
| Symbol | Inhibitor_I10 | ||||||||
| Pfam | PF12559 | ||||||||
| InterPro | IPR022217 | ||||||||
| |||||||||
Denna familj inkluderar både mikroviridiner och marinostatiner. Det verkar troligt att det i båda fallen är C-terminalen som blir den aktiva inhibitorn efter posttranslationella modifieringar av prepeptidens fullängd. Det är esterbindningarna inom nyckelregionen med 12 rester som cirkulerar molekylen och ger den dess hämmande konformation .
Inhibitor I24
| PinA-peptidasinhibitor- | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| identifierare | |||||||||
| Symbol | Inhibitor_I24 | ||||||||
| Pfam | PF10465 | ||||||||
| InterPro | IPR019506 | ||||||||
| MEROPS | I24 | ||||||||
| |||||||||
Denna familj inkluderar PinA, som hämmar endopeptidaset La. Det binder till La- homotetrameren men interfererar inte med ATP- bindningsstället eller det aktiva stället för La.
Inhibitor I29
| Katepsinpropeptidinhibitordomän (I29) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
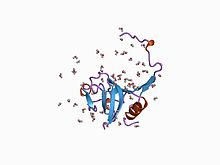 kristallstruktur av en cysteinproteasproform
| |||||||||
| Identifierare | |||||||||
| Symbol | Inhibitor_I29 | ||||||||
| Pfam | PF08246 | ||||||||
| InterPro | IPR013201 | ||||||||
| |||||||||
Inhibitor I29 -domänen , som tillhör MEROPS-peptidasinhibitorfamiljen I29, finns vid N-terminalen av en mängd olika peptidasprekursorer som tillhör MEROPS-peptidasunderfamiljen C1A; dessa inkluderar cathepsin L, papain och procaricain. Den bildar en alfa-spiralformad domän som löper genom det substratbindande stället, vilket förhindrar åtkomst. Avlägsnande av denna region genom proteolytisk klyvning resulterar i aktivering av enzymet . Denna domän finns också, i en eller flera kopior, i en mängd olika cysteinpeptidashämmare såsom salarin.
Inhibitor I34
| Sackaropepsinhämmare I34 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 strukturen av proteinas a komplexbunden med en ia3 mutant hämmare
| |||||||||
| Identifierare | |||||||||
| Symbol | Inhibitor_I34 | ||||||||
| Pfam | PF10466 | ||||||||
| InterPro | IPR019507 | ||||||||
| MEROPS | I34 | ||||||||
| |||||||||
Sackaropepsinhämmaren I34 är mycket specifik för asparaginpeptidaset sackaropepsin . I frånvaro av sackaropepsin är den i stort sett ostrukturerad, men i dess närvaro genomgår inhibitorn en konformationsförändring som bildar en nästan perfekt alfa-helix från Asn 2 till Met 32 i den aktiva ställespalten i peptidaset.
Inhibitor I36
| Peptidashämmare familj I36 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
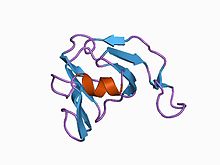 3D-strukturen av streptomyces metalloproteinashämmare, smpi, isolerad från streptomyces nigrescens tk-23, nmr, minimerad medelstruktur
| |||||||||
| Identifierare | |||||||||
| Symbol | Inhibitor_I36 | ||||||||
| Pfam | PF03995 | ||||||||
| Pfam klan | CL0333 | ||||||||
| InterPro | IPR007141 | ||||||||
| MEROPS | I36 | ||||||||
| SCOP2 | 1bhu / SCOPe / SUPFAM | ||||||||
| |||||||||
Peptidashämmarfamiljen I36-domän finns endast i ett litet antal proteiner som är begränsade till Streptomyces -arter . Alla har fyra konserverade cysteiner som troligen bildar två disulfidbindningar . Ett av dessa proteiner från Streptomyces nigrescens är den välkarakteriserade metalloproteinashämmaren SMPI .
Strukturen för SMPI har bestämts . Den har 102 aminosyrarester med två disulfidbryggor och hämmar specifikt metalloproteinaser som termolysin , som tillhör MEROPS peptidasfamiljen M4. SMPI består av två beta-ark , var och en bestående av fyra antiparallella beta-strängar. Strukturen kan betraktas som två grekiska nyckelmotiv med 2-faldig inre symmetri, en grekisk nyckel beta-pipa . En unik strukturell egenskap som finns i SMPI är i dess förlängning mellan den första och andra strängen av det andra grekiska nyckelmotivet som är känt för att vara involverat i den hämmande aktiviteten av SMPI. I frånvaro av sekvenslikhet visar SMPI- strukturen tydlig likhet med båda domänerna av ögonlinskristallinerna , båda domänerna av kalciumsensorproteinet-S, såväl som jästdödartoxinet med en domän . Jästdödartoxinstrukturen ansågs vara en föregångare till beta-gamma-kristallina proteiner med två domäner, på grund av dess strukturella likhet med varje domän av beta-gamma-kristallinerna. SMPI tillhandahåller således ett annat exempel på en endomänproteinstruktur som motsvarar den förfädersvecka från vilken tvådomänsproteinerna i beta-gamma-kristallin superfamiljen tros ha utvecklats .
Inhibitor I42
| Chagasinfamiljens peptidashämmare I42 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 lösningsstruktur av trypanosoma cruzi cysteinproteashämmare chagasin
| |||||||||
| Identifierare | |||||||||
| Symbol | Inhibitor_I42 | ||||||||
| Pfam | PF09394 | ||||||||
| InterPro | IPR018990 | ||||||||
| MEROPS | I42 | ||||||||
| |||||||||
Inhibitorfamiljen I42 inkluderar chagasin, en reversibel hämmare av papainliknande cysteinproteaser . Chagasin har en beta-fatstruktur , vilket är en unik variant av immunglobulinvecket med homologi med human CD8alfa .
Inhibitor I48
| Peptidashämmare klitocypin | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identifierare | |||||||||
| Symbol | Inhibitor_I48 | ||||||||
| Pfam | PF10467 | ||||||||
| InterPro | IPR019508 | ||||||||
| MEROPS | I48 | ||||||||
| |||||||||
Inhibitorfamiljen I48 inkluderar klitocypin, som binder och hämmar cysteinproteinaser. Det har ingen likhet med några andra kända cysteinproteinashämmare men har viss likhet med en lektinliknande familj av proteiner från svamp .
Inhibitor I53
| Trombinhämmare Madanin | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identifiers | |||||||||
| Symbol | Inhibitor_I53 | ||||||||
| Pfam | PF11714 | ||||||||
| InterPro | IPR021716 | ||||||||
| MEROPS | I53 | ||||||||
| |||||||||
Medlemmar av denna familj är peptidashämmaren madaninproteiner . Dessa proteiner isolerades från fästingsaliv .
Inhibitor I67
| Bromelainhämmare VI | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 nmr-struktur av bromelainhämmare vi från ananasstam
| |||||||||
| Identifierare | |||||||||
| Symbol | Inhibitor_I67 | ||||||||
| Pfam | PF11405 | ||||||||
| InterPro | IPR022713 | ||||||||
| MEROPS | I67 | ||||||||
| |||||||||
Bromelainhämmare VI, i familjen Inhibitor I67, är en dubbelkedjig hämmare som består av en kedja med 11 rester och en kedja med 41 rester .
Inhibitor I68
| Karboxypeptidashämmare I68 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 kristallstruktur av fästingkarboxipeptidasinhibitorn i komplex med humant karboxipeptidas b
| |||||||||
| Identifierare | |||||||||
| Symbol | Inhibitor_I68 | ||||||||
| Pfam | PF10468 | ||||||||
| InterPro | IPR019509 | ||||||||
| MEROPS | I68 | ||||||||
| |||||||||
Karboxypeptidasinhibitor I68-familjen representerar en familj av fästingkarboxipetidashämmare.
Inhibitor I78
| Identifierare | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| av familjen Peptidashämmare I78 | |||||||||
| Symbol | Inhibitor_I78 | ||||||||
| Pfam | PF11720 | ||||||||
| Pfam klan | CL0367 | ||||||||
| InterPro | IPR021719 | ||||||||
| MEROPS | I78 | ||||||||
| |||||||||
Familjen peptidashämmare I78 inkluderar Aspergillus elastashämmare .
Föreningar
- Aprotinin
- Bestatin
- Calpain-hämmare I och II
- Kymostatin
- E-64
- Leupeptin ( N -acetyl- L -leucyl - L -leucyl- L -argininal)
- alfa-2-makroglobulin
- Pefabloc SC
- Pepstatin
- PMSF (fenylmetansulfonylfluorid)
- TLCK
- Trypsinhämmare