mRNA-övervakning
mRNA-övervakningsmekanismer är vägar som används av organismer för att säkerställa trohet och kvalitet hos budbärar-RNA ( mRNA)-molekyler. Det finns ett antal övervakningsmekanismer i cellerna. Dessa mekanismer fungerar i olika steg av mRNA- biogenesvägen för att detektera och bryta ned transkript som inte har bearbetats korrekt.
Översikt
Översättningen av budbärar-RNA -transkript till proteiner är en viktig del av molekylärbiologins centrala dogm . mRNA-molekyler är dock utsatta för en mängd trohetsfel som kan orsaka fel vid översättning av mRNA till kvalitetsproteiner . RNA-övervakningsmekanismer är metoder som celler använder för att säkerställa kvaliteten och troheten hos mRNA-molekylerna. Detta uppnås i allmänhet genom att markera avvikande mRNA-molekyler för nedbrytning av olika endogena nukleaser .
mRNA-övervakning har dokumenterats i bakterier och jäst . Hos eukaryoter är dessa mekanismer kända för att fungera i både kärnan och cytoplasman . Trolighetskontroller av mRNA-molekyler i kärnan resulterar i nedbrytning av felaktigt bearbetade transkript innan export till cytoplasman. Transkript är föremål för ytterligare övervakning en gång i cytoplasman. Cytoplasmatiska övervakningsmekanismer bedömer mRNA- transkript med avseende på frånvaro av eller närvaro av prematura stoppkodon.
Tre övervakningsmekanismer är för närvarande kända för att fungera inom celler : den nonsens-medierade mRNA-sönderfallsvägen (NMD); de nonstop-medierade mRNA-sönderfallsvägarna (NSD); och den no-go-medierade mRNA-sönderfallsvägen (NGD).
Nonsens-medierat mRNA-sönderfall
Översikt
Nonsens-medierat sönderfall är involverat i detektion och sönderfall av mRNA-transkript som innehåller prematura termineringskodon (PTC). PTC kan uppstå i celler genom olika mekanismer: könslinjemutationer i DNA; somatiska mutationer i DNA; fel i transkription ; eller fel i posttranskriptionell mRNA-bearbetning. Underlåtenhet att känna igen och sönderfalla dessa mRNA-transkript kan resultera i produktion av trunkerade proteiner som kan vara skadliga för organismen. Genom att orsaka sönderfall av C-terminalt trunkerade polypeptider, kan NMD-mekanismen skydda celler mot skadliga dominant -negativa och vinst av funktionseffekter . PTC har varit inblandade i cirka 30 % av alla ärftliga sjukdomar; som sådan spelar NMD-vägen en viktig roll för att säkerställa en organisms totala överlevnad och kondition.
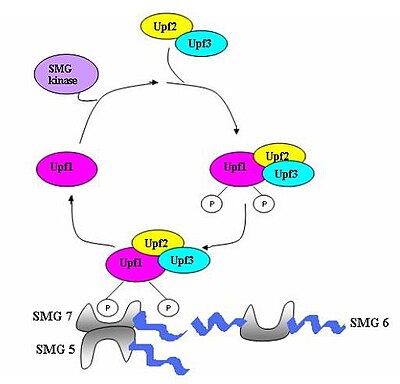
Ett övervakningskomplex som består av olika proteiner (eRF1, eRF3, Upf1, Upf2 och Upf3) sätts ihop och skannar mRNA:t för förtida stoppkodon. Sammansättningen av detta komplex utlöses av för tidig översättningsavslutning. Om ett för tidigt stoppkodon detekteras signaleras mRNA-transkriptet för nedbrytning - kopplingen mellan detektion och nedbrytning sker.
Sju smg-gener (smg1-7) och tre UPF-gener (Upf1-3) har identifierats i Saccharomyces cerevisiae och Caenorhabditis elegans som väsentliga transagerande faktorer som bidrar till NMD-aktivitet. Alla dessa gener är bevarade i Drosophila melanogaster och andra däggdjur där de också spelar avgörande roller i NMD. I eukaryoter finns det tre komponenter som bevaras i NMD-processen. Dessa är komplexen Upf1/SMG-2, Upf2/SMG-3 och Upf3/SMG-4. Upf1/SMG-2 är ett fosfoprotein i flercelliga organismer och tros bidra till NMD via sin fosforyleringsaktivitet. Men de exakta interaktionerna mellan proteinerna och deras roller i NMD är för närvarande omtvistade.
Mekanism hos däggdjur
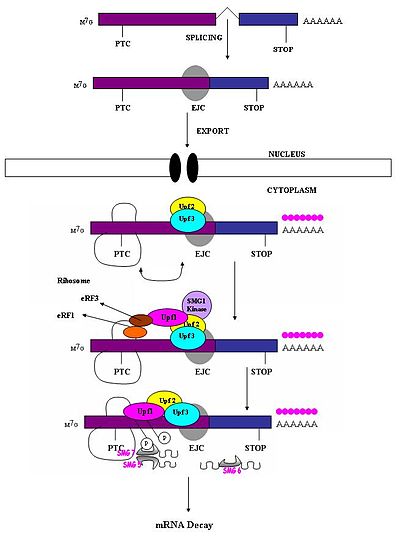
Ett för tidigt stoppkodon måste identifieras som annorlunda än ett normalt stoppkodon så att endast det förra utlöser ett NMD-svar. Det har observerats att förmågan hos ett nonsenskodon att orsaka mRNA-nedbrytning beror på dess relativa läge till nedströmssekvenselementet och associerade proteiner. Studier har visat att nukleotider mer än 50–54 nukleotider uppströms om den sista exon-exon-övergången kan rikta in sig på mRNA för sönderfall. De nedströms från denna region kan inte göra det. Sålunda ligger nonsenskodoner mer än 50-54 nukleotider uppströms från den sista exongränsen medan naturliga stoppkodon är belägna inom terminala exoner. Exon junction complexes (EJCs) markerar exon-exon gränserna. EJC är multiproteinkomplex som sätts samman under splitsning i en position cirka 20–24 nukleotider uppströms från splitsningsövergången. Det är denna EJC som tillhandahåller positionsinformation som behövs för att skilja prematura stoppkodon från naturliga stoppkodon. Igenkänning av PTC:er verkar vara beroende av definitionerna av exon-exon-övergångarna. Detta tyder på involvering av spliceosomen i NMD hos däggdjur. Forskning har undersökt möjligheten av spliceosom involvering i däggdjurs NMD och har fastställt att detta är en trolig möjlighet. Dessutom har det observerats att NMD-mekanismer inte aktiveras av nonsens-transkript som genereras från gener som naturligt inte innehåller introner (t.ex. Histon H4, Hsp70, melanokortin-4-receptor).
När ribosomen når en PTC interagerar translationsfaktorerna eRF1 och eRF3 med kvarhållna EJC-komplex genom en multiproteinbrygga. Interaktionerna mellan UPF1 och det terminerande komplexet och med UPF2 /UPF3 hos de bibehållna EJC:erna är kritiska. Det är dessa interaktioner som riktar sig mot mRNA:t för snabb sönderfall av endogena nukleaser

Mekanism hos ryggradslösa djur
Studier som involverar organismer såsom S. cerevisiae , D. melanogaster och C. elegans har visat att PTC-igenkänning som involverar ryggradslösa organismer inte involverar exon-exon-gränser. Dessa studier tyder på att ryggradslösa NMD förekommer oberoende av splitsning. Som ett resultat av detta krävs inte EJC som är ansvariga för att markera exon-exongränser i ryggradslösa NMD. Flera modeller har föreslagits för att förklara hur PTC särskiljs från normala stoppkodon hos ryggradslösa djur. En av dessa antyder att det kan finnas ett nedströms sekvenselement som fungerar liknande exonövergångarna hos däggdjur. En andra modell föreslår att ett brett närvarande särdrag i mRNA, såsom en 3'-poly-A-svans, kan ge den positionsinformation som krävs för igenkänning. En annan modell, kallad "faux 3'UTR-modellen", föreslår att för tidig översättningsuppsägning kan skiljas från normal uppsägning på grund av inneboende egenskaper som kan göra det möjligt för den att känna igen sin närvaro i en olämplig miljö. Dessa mekanismer har dock ännu inte slutgiltigt demonstrerats.
Mekanism i växter
Det finns två mekanismer för PTC-igenkänning i växter: beroende på dess avstånd från EJC (som hos ryggradsdjur) eller från poly-A-svansen. NMD-mekanismen i växter inducerar sönderfallet av mRNA som innehåller en 3'UTR längre än 300 nukleotider, det är därför andelen mRNA med längre 3'UTR är mycket lägre i växter än hos ryggradsdjur.
NMD undvikande
mRNA med nonsensmutationer anses generellt vara målinriktade för sönderfall via NMD-vägarna. Närvaron av detta prematura stoppkodon omkring 50-54 nukleotider 5' till exonövergången verkar vara utlösaren för snabbt sönderfall; dock har det observerats att vissa mRNA-molekyler med ett för tidigt stoppkodon kan undvika upptäckt och sönderfall. I allmänhet har dessa mRNA-molekyler stoppkodonet mycket tidigt i läsramen (dvs PTC är AUG-proximal). Detta verkar vara en motsägelse till den nuvarande accepterade modellen av NMD eftersom denna position är signifikant 5' om exon-exon-övergången.
Detta har visats i β-globulin. β-globulin-mRNA som innehåller en nonsensmutation tidigt i genens första exon är mer stabila än NMD-känsliga mRNA-molekyler. Den exakta mekanismen för att undvika upptäckt är för närvarande inte känd. Det har föreslagits att det poly-A-bindande proteinet (PABP) verkar spela en roll i denna stabilitet. Det har visats i andra studier att närvaron av detta protein nära AUG-proximala PTC:er verkar främja stabiliteten hos dessa annars NMD-känsliga mRNA. Det har observerats att denna skyddande effekt inte är begränsad endast till p-globulinpromotorn. Detta tyder på att denna NMD-undvikande mekanism kan vara utbredd i andra vävnadstyper för en mängd olika gener. Den nuvarande modellen för NMD kan behöva ses över vid ytterligare studier.
Nonstop-medierat mRNA-sönderfall
Översikt
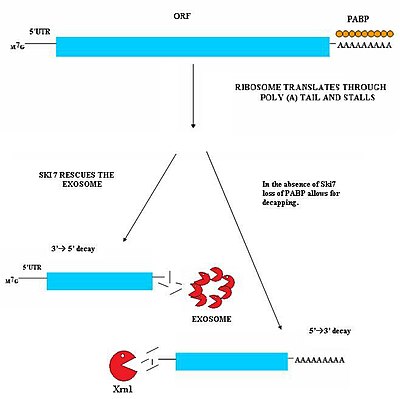
Nonstop mediated decay (NSD) är involverad i detekteringen och sönderfallet av mRNA-transkript som saknar ett stoppkodon. Dessa mRNA-transkript kan uppstå från många olika mekanismer såsom för tidig 3'-adenylering eller kryptiska polyadenyleringssignaler inom den kodande regionen av en gen. Denna brist på ett stoppkodon resulterar i ett betydande problem för celler. Ribosomer som översätter mRNA:t översätts så småningom till 3'poly-A-svansregionen av transkript och stall. Som ett resultat kan den inte skjuta ut mRNA:t. Ribosomer kan således bli sekvestrerade associerade med nonstop-mRNA:t och skulle inte vara tillgängliga för att översätta andra mRNA-molekyler till proteiner. Nonstop-medierat sönderfall löser detta problem genom att både frigöra de stannade ribosomerna och markera nonstop-mRNA:t för nedbrytning i cellen av nukleaser. Nonstop-medierat sönderfall består av två distinkta vägar som sannolikt verkar tillsammans för att sönderfalla nonstop mRNA.
Ski7 väg
Denna väg är aktiv när Ski7-protein är tillgängligt i cellen. Ski7-proteinet tros binda till det tomma A-stället i ribosomen. Denna bindning gör det möjligt för ribosomen att skjuta ut den fasta nonstop mRNA-molekylen – detta frigör till och med ribosomen och gör att den kan översätta andra transkript. Ski7 är nu associerad med nonstop mRNA och det är denna association som riktar sig mot nonstop mRNA för igenkänning av den cytosoliska exosomen . Ski7-exosomkomplexet deadenylerar snabbt mRNA-molekylen vilket gör att exosomen kan sönderfalla transkriptet på ett 3' till 5'-sätt.
Icke-Ski7 väg
En andra typ av NSD har observerats i jäst. I denna mekanism resulterar frånvaron av Ski7 i förlusten av poly-A svansbindande PABP-proteiner genom verkan av translationsribosomen. Avlägsnandet av dessa PABP-proteiner resulterar sedan i förlusten av det skyddande 5'm7G-locket . Förlusten av locket resulterar i snabb nedbrytning av transkriptet av ett endogent 5'-3' exonukleas såsom XrnI.
No-Go förfall
No-Go decay (NGD) är den senast upptäckta övervakningsmekanismen. Som sådan är det för närvarande inte väl förstått. Även om autentiska mål för NGD är dåligt förstådda, verkar de till stor del bestå av mRNA-transkript på vilka ribosomer har stannat under translation. Detta stall kan orsakas av en mängd olika faktorer, inklusive starka sekundära strukturer , som fysiskt kan blockera translationsmaskineriet från att flyttas ner i transkriptet. Dom34/Hbs1 binder sannolikt nära A-stället för avstannade ribosomer och kan underlätta återvinning av komplex. I vissa fall klyvs transkriptet också på ett endonukleolytiskt sätt nära stallplatsen; emellertid förblir identiteten för det ansvariga endonukleaset omtvistad. De fragmenterade mRNA-molekylerna bryts sedan helt ned av exosomen på ett 3' till 5' sätt och av Xrn1 på ett 5' till 3' sätt. Det är för närvarande inte känt hur denna process frigör mRNA från ribosomerna, men Hbs1 är nära besläktat med Ski7-proteinet som spelar en tydlig roll i ribosomfrisättningen i Ski7-medierad NSD. Det postuleras att Hbs1 kan spela en liknande roll i NGD.
Evolution
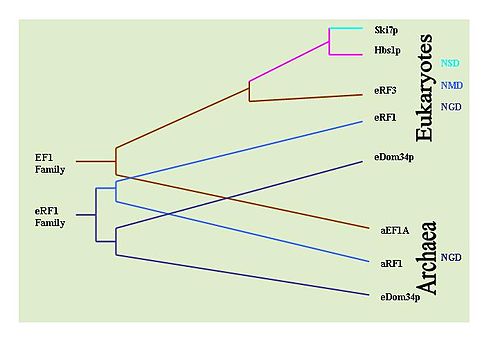
Det är möjligt att bestämma den evolutionära historien för dessa mekanismer genom att observera bevarandet av nyckelproteiner som är inblandade i varje mekanism. Till exempel: Dom34/Hbs1 är associerade med NGD; Ski7 förknippas med NSD; och eRF-proteinerna är associerade med NMD. För detta ändamål har omfattande BLAST -sökningar utförts för att fastställa förekomsten av proteinerna i olika typer av organismer. Det har fastställts att NGD Hbs1 och NMD eRF3 endast finns i eukaryoter. NGD Dom34 är dock universell i eukaryoter och arkéer . Detta tyder på att NGD verkar ha varit den första utvecklade mRNA-övervakningsmekanismen. NSD Ski7-proteinet verkar vara strikt begränsat till jästarter, vilket tyder på att NSD är den senast utvecklade övervakningsmekanismen. Detta lämnar som standard NMD som den andra utvecklade övervakningsmekanismen.