Hepatit delta virus ribozym
| Hepatit delta-virus ribozym | |
|---|---|
 Förutspådd sekundär struktur och sekvenskonservering av HDV-
| |
| ribozymidentifierare | |
| Symbol | HDV_ribozym |
| Rfam | RF00094 |
| Övriga uppgifter | |
| RNA -typ | Gen ; ribozym |
| Domän(er) | Virus |
| SÅ | SO:0000374 |
| PDB- strukturer | PDBe |
för hepatit delta-virus (HDV) är ett icke-kodande RNA som finns i hepatit delta-viruset som är nödvändigt för viral replikation och är det enda kända humana viruset som använder ribozymaktivitet för att infektera sin värd. Ribozymet verkar för att bearbeta RNA-transkripten till enhetslängder i en självklyvningsreaktion under replikering av hepatit delta-viruset, som tros föröka sig genom en dubbel rullande cirkelmekanism . Ribozymet är aktivt in vivo i frånvaro av några proteinfaktorer och var det snabbaste kända naturligt förekommande självklyvande RNA:t vid tidpunkten för dess upptäckt.
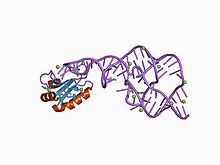
Kristallstrukturen för detta ribozym har lösts med hjälp av röntgenkristallografi och visar fem spiralformade segment förbundna med en dubbel pseudoknot .
Förutom sense (genomisk version) har alla HDV-virus också en antigenomisk version av HDV-ribozym. Denna version är inte den exakta komplementära sekvensen utan antar samma struktur som den sens (genomiska) strängen. De enda "väsentliga" skillnaderna mellan de två är en liten utbuktning i P4-stammen och en kortare J4/2-övergång. Både de genomiska och antigenomiska ribozymen är nödvändiga för replikering.
HDV-liknande ribozymer
HDV-ribozymet är strukturellt och biokemiskt relaterat till många andra självklyvande ribozymer. Dessa andra ribozymer hänvisas ofta till som exempel på HDV-ribozymer på grund av dessa likheter, även om de inte finns i hepatit delta-virus. De kan också kallas "HDV-liknande" för att indikera detta faktum.
HDV-liknande ribozymer inkluderar däggdjurs-CPEB3-ribozym , retrotransposonmedlemmar (t.ex. i R2-RNA-elementet i insekter och i L1Tc och förmodligen andra retrotransposoner i trypanosomatider) och sekvenser från bakterier. Grupperingen är förmodligen ett resultat av konvergent evolution : Deltavirus som hittats utanför människor har också ett DV-ribozym, och inga föreslagna horisontella genöverföringsscenarier kan ännu förklara detta.
Katalysmekanism
HDV-ribozymet katalyserar klyvning av fosfodiesterbindningen mellan substratnukleotiden eller oligonukleotiden och ribozymets 5'-hydroxyl. I hepatit delta-viruset börjar denna substratnukleotidsekvens med uridin och är känd som U(-1), men identiteten för -1-nukleotiden förändrar inte katalyshastigheten signifikant. Det finns bara ett krav på dess kemiska natur, eftersom som visas av Perrotta och Been, substitution av U(-1)-ribosen med deoxiribos avskaffar reaktionen, vilket är förenligt med förutsägelsen att 2'-hydroxylen är nukleofilen i kemisk reaktion. Till skillnad från många andra ribozymer, såsom hammarhuvud-ribozym, har HDV-ribozym inga uppströmskrav för katalys och kräver endast en enda -1-ribonukleotid som ett substrat för att effektivt reagera.
Till en början trodde man att den 75:e nukleotiden i ribozymet, ett cytosin känt som C75, kunde fungera som en allmän bas med N3 från C75 som abstraherar en proton från 2'-hydroxylen i U(-1) nukleotiden till underlätta nukleofil attack på fosfodiesterbindningen. Men även om det är väl etablerat att N3 för C75 har ett pKa stört från sitt normala värde på 4,45 och är närmare cirka 6,15 eller 6,40, är det inte tillräckligt neutralt för att fungera som en allmän baskatalysator. Istället tros N3 i C75 fungera som en Lewis-syra för att stabilisera den lämnande 5'-hydroxylen i ribozymet; detta stöds av dess närhet till 5'-hydroxylen i kristallstrukturen. Substitution av C75-nukleotiden med någon annan nukleotid upphäver eller försämrar väsentligt ribozymaktivitet, även om denna aktivitet delvis kan återställas med imidazol, vilket ytterligare implicerar C75 i katalytisk aktivitet.
C75 i HDV-ribozym har varit föremål för flera studier på grund av dess säregna pKa. De typiska pKa-värdena för de fria nukleosiderna är omkring 3,5 till 4,2; dessa lägre pKa-värden är sura och det är osannolikt att de skulle bli basiska. Det är emellertid troligt att den strukturella miljön inom ribozymet, som inkluderar en desolvatiserad aktiv platsklyfta, ger negativ elektrostatisk potential som skulle kunna störa pKa för cytosin tillräckligt för att fungera som en Lewis-syra.
Förutom Lewis-syrastabilisering av den lämnande 5'-hydroxylgruppen är det nu också accepterat att HDV-ribozym kan använda en metalljon för att hjälpa till med aktivering av 2'-hydroxylen för attack på U(-1)-nukleotiden. En magnesiumjon i ribozymets aktiva ställe är koordinerad till 2'-hydroxyl-nukleofilen och ett syre i det sprickbara fosfatet, och kan fungera som en Lewis-syra för att aktivera 2'-hydroxylen. Dessutom är det möjligt att fosfatet av U23 kan fungera som en Lewis-syra för att acceptera en proton från 2'-hydroxylen med magnesiumet som en koordinerande jon. Eftersom HDV-ribozym inte kräver att metalljoner har aktivitet, är det inte ett obligatoriskt metalloenzym, men närvaron av magnesium i det aktiva stället förbättrar avsevärt klyvningsreaktionen. HDV-ribozymet verkar ha ett ospecifikt krav på att låga mängder tvåvärda katjoner ska vikas, eftersom det är aktivt i Mg2+, Ca2 + , Mn2 + och Sr2 + . I frånvaro av metalljoner verkar det troligt att vatten kan ersätta magnesiums roll som en Lewis-syra.
Reglering genom uppströms RNA
Som begränsad av den snabba självklyvande naturen hos HDV-ribozym, utfördes de tidigare ribonukleasexperimenten på 3'-produkten av självklyvning snarare än prekursorn. Flankerande sekvens är emellertid känd för att delta i regleringen av självklyvningsaktiviteten hos HDV-ribozym. Därför har uppströmssekvensen 5' till självklyvningsstället inkorporerats för att studera den resulterande självklyvningsaktiviteten för HDV-ribozym. Två alternativa strukturer har identifierats.
Den första hämmande strukturen viks av ett förlängt transkript (dvs -30/99 transkript, koordinater refereras mot självklyvningsstället) som sträcker sig från 30 nt uppströms om klyvningsstället till 15 nt nedströms om 3'-änden. Den flankerande sekvensen binder ribozymet i en kinetisk fälla under transkription och resulterar i den extremt minskade självklyvningshastigheten. Denna självklyvningsförhindrande struktur inkluderar 3 alternativa stammar: Alt1, Alt2 och Alt3, som stör den aktiva konformationen. Alt1 är en 10-bp långdistansinteraktion bildad av en hämmande uppströmssträcka (-25/-15 nt) och nedströmssträckan (76/86 nt). Alt1 stör stammen P2 i den aktiva konformationen där P2 föreslås ha en aktiverande roll för både genomiskt och antigenomiskt ribozym. Alt2 är en interaktion mellan uppströms flankerande sekvens och ribozymet, och Alt3 är en icke-nativ ribozym-ribozym-interaktion.
Den sekundära strukturen av denna hämmande konformation stöds av olika experimentella tillvägagångssätt. Först utfördes direkt sondering via ribonukleaser och den efterföljande modelleringen via mfold 3.0 med användning av begränsningar från sonderingsresultaten överensstämmer med den föreslagna strukturen. För det andra användes en serie DNA-oligomerer som var komplementära till olika regioner av AS1/2 för att rädda ribozymaktiviteten; resultaten bekräftar AS1/2:s hämmande roll. För det tredje introducerar mutationsanalys enkla/dubbla mutationer utanför ribozymet för att säkerställa att den observerade ribozymaktiviteten är direkt associerad med stabiliteten hos Alt1. Stabiliteten hos AS1 har visat sig vara omvänt relaterad till självklyvningsaktiviteten.
Den andra tillåtande strukturen gör det möjligt för HDV-ribozym att självklyva co-transkriptionellt och denna struktur inkluderar vidare -54/-18 nt-delen av RNA-transkriptet. Den uppströms inhiberande -24/-15-sträckan från den tidigare nämnda inhiberande konformationen är nu sekvestrerad i en hårnål P(-1) belägen uppströms om klyvningsstället. P(-1)-motivet återfinns emellertid endast i den genomiska sekvensen, vilket kan vara korrelerat med fenomenet att genomiska HDV-RNA-kopior är mer rikligt förekommande i de infekterade levercellerna. Experimentella bevis stöder också denna alternativa struktur. För det första används strukturell kartläggning via ribonukleas för att sondera -54/-1-fragmentet istället för hela prekursortranskriptet på grund av den snabbklyvande naturen hos denna struktur, vilket överensstämmer med den lokala hårnålen P(-1) (mellan -54/ -40 och -18/-30 nt). För det andra finns evolutionär konservering i P(-1) och länkningsregionen mellan P(-1) och P1 bland 21 genomiska HDV-RNA-isolat.
Används vid beredning av RNA-transkript
De speciella egenskaperna hos HDV-ribozymets klyvningsreaktion gör det till ett användbart verktyg för att framställa RNA-transkript med homogena 3′-ändar, ett alternativ till transkription av RNA med T7-RNA-polymeras som ofta kan producera heterogena ändar eller oönskade tillägg. cDNA-versionen av ribozymet kan framställas intill cDNA för mål-RNA-sekvensen och RNA framställt från transkription med T7-RNA-polymeras. Ribozymsekvensen kommer effektivt att klyva sig själv utan krav nedströms, eftersom -1-nukleotiden är invariant, vilket lämnar ett 2′–3′ cykliskt fosfat som lätt kan avlägsnas genom behandling med ett fosfatas eller T4-polynukleotidkinas. Mål-RNA:t kan sedan renas med gelrening.