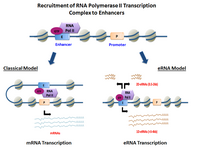
Enhancer RNA
Enhancer-RNA (eRNA) representerar en klass av relativt långa icke-kodande RNA- molekyler (50-2000 nukleotider) transkriberade från DNA-sekvensen av förstärkarregioner . De upptäcktes först 2010 genom användning av genomomfattande tekniker som RNA-seq och ChIP-seq . eRNA kan delas in i två huvudklasser: 1D eRNA och 2D eRNA, som skiljer sig främst när det gäller storlek, polyadenyleringstillstånd och transkriptionell riktning. Uttrycket av ett givet eRNA korrelerar med aktiviteten hos dess motsvarande förstärkare i målgener. Allt fler bevis tyder på att eRNA aktivt spelar en roll i transkriptionell reglering i cis och i trans , och medan deras verkningsmekanismer förblir oklara, har några modeller föreslagits.
Upptäckt
Förstärkare som platser för extragen transkription upptäcktes initialt i genomomfattande studier som identifierade förstärkare som vanliga regioner av RNA-polymeras II (RNA pol II)-bindande och icke-kodande RNA- transkription . Nivån av RNA pol II-förstärkarinteraktion och RNA-transkriptbildning visade sig vara mycket varierande bland dessa initiala studier. Med användning av explicita kromatinsignaturtoppar befanns en betydande andel (~70%) av extragena RNA Pol II- transkriptionsstartställen överlappa förstärkarställen i murina makrofager . Av 12 000 neuronala förstärkare i musgenomet befanns nästan 25% av platserna binda RNA Pol II och generera transkript . I parallella studier identifierades 4 588 extragena RNA Pol II- bindningsställen med hög konfidens i murina makrofager stimulerade med den inflammatoriska mediatern lipopolysackarid för att inducera transkription. Dessa eRNA, till skillnad från budbärar-RNA (mRNA), saknade modifiering genom polyadenylering , var i allmänhet korta och icke-kodande och transkriberades dubbelriktat. Senare studier avslöjade transkriptionen av en annan typ av eRNA, genererad genom enkelriktad transkription, som var längre och innehöll en poly A-svans . Dessutom var eRNA-nivåer korrelerade med mRNA- nivåer av närliggande gener , vilket tyder på den potentiella reglerande och funktionella rollen för dessa icke-kodande förstärkar - RNA -molekyler .
Biogenes
Sammanfattning
eRNA:n transkriberas från DNA-sekvenser uppströms och nedströms om extragena förstärkarregioner . Tidigare har flera modellförstärkare visat förmågan att direkt rekrytera RNA Pol II och allmänna transkriptionsfaktorer och bilda pre-initiation complex (PIC) före transkriptionsstartstället vid promotorn av gener . I vissa celltyper har aktiverade förstärkare visat förmågan att både rekrytera RNA Pol II och även tillhandahålla en mall för aktiv transkription av deras lokala sekvenser .
transkriptionens riktning , genererar förstärkarregioner två olika typer av icke-kodande transkript , 1D-eRNA och 2D-eRNA. Naturen hos pre-initieringskomplexet och specifika transkriptionsfaktorer som rekryteras till förstärkaren kan styra typen av genererade eRNA. Efter transkription finns majoriteten av eRNA kvar i kärnan . I allmänhet är eRNA mycket instabila och bryts ned aktivt av den nukleära exosomen . Inte alla förstärkare transkriberas, med icke-transkriberade förstärkare som är betydligt fler än de transkriberade i storleksordningen dussintals tusentals i varje given celltyp .
1D eRNA
I de flesta fall genererar enkelriktad transkription av förstärkarregioner långa (>4kb) och polyadenylerade eRNA. Förstärkare som genererar polyA+ eRNA har ett lägre H3K4me1 /me3-förhållande i sin kromatinsignatur än 2D-eRNA. PolyA+ eRNA skiljer sig från långa multiexoniska polytranskript (meRNA) som genereras genom transkriptionsinitiering vid intragena förstärkare . Dessa långa icke-kodande RNA, som exakt återspeglar värdgenens struktur förutom den alternativa första exonen , visar dålig kodningspotential. Som ett resultat kan polyA+ 1D-eRNA representera en blandad grupp av sanna förstärkarmallade RNA:n och multiexoniska RNA:n.
2D eRNA
Dubbelriktad transkription vid förstärkarställen genererar jämförelsevis kortare (0,5-2 kb) och icke-polyadenylerade eRNA. Förstärkare som genererar polyA-eRNA har en kromatinsignatur med ett högre H3K4me1/me3-förhållande än 1D-eRNA. I allmänhet förstärkartranskription och produktion av dubbelriktade eRNA en stark korrelation av förstärkaraktivitet på gentranskription .
Frekvens och tidpunkt för eRNA-uttryck
Arner et al. identifierade 65 423 transkriberade förstärkare (som producerar eRNA) bland 33 olika celltyper under olika förhållanden och olika tidpunkter för stimulering. Transkriptionen av förstärkare föregick i allmänhet transkription av transkriptionsfaktorer som i sin tur i allmänhet föregick budbärar-RNA (mRNA) transkription av gener.
Carullo et al. undersökte en speciell celltyp, neuroner (från primära neuronkulturer). De uppvisade 28 492 förmodade förstärkare som genererade eRNA. Dessa eRNA transkriberades ofta från båda strängarna av förstärkar-DNA i motsatta riktningar. Carullo et al. använde dessa odlade neuroner för att undersöka tidpunkten för specifika förstärkar-eRNA jämfört med mRNA för deras målgener. De odlade neuronerna aktiverades och RNA isolerades från dessa neuroner 0, 3,75, 5, 7,5, 15, 30 och 60 minuter efter aktivering. Under dessa experimentella förhållanden fann de att 2 av de 5 förstärkarna av den omedelbara tidiga genen (IEG) FOS , det vill säga FOS-förstärkare 1 och FOS-förstärkare 3, blev aktiverade och initierade transkription av deras eRNA (eRNA1 och eRNA3). FOS eRNA1 och eRNA3 uppreglerades signifikant inom 7,5 minuter, medan FOS mRNA endast uppreglerades 15 minuter efter stimulering. Liknande mönster inträffade vid IEGs FOSb och NR4A1 , vilket indikerar att för många IEG:er föregår eRNA-induktion mRNA-induktion som svar på neuronal aktivering.
Medan vissa förstärkare kan aktivera sina målpromotorer vid sina målgener utan att transkribera eRNA, transkriberar de flesta aktiva förstärkare eRNA under aktivering av sina målpromotorer.
Funktioner av eRNA som hittades under perioden 2013 till 2021
Funktionerna för eRNA som beskrivs nedan har rapporterats i olika biologiska system, ofta demonstrerade med ett litet antal specifika förstärkar-målgenpar. Det är inte klart i vilken utsträckning funktionerna hos eRNA som beskrivs här kan generaliseras till de flesta eRNA.
eRNA i loopbildning

Kromosomslingorna som visas i figuren, som för en förstärkare till promotorn av dess målgen, kan styras och bildas av eRNA:t som transkriberas från förstärkaren efter att förstärkaren har aktiverats.
Ett transkriberat förstärkar-RNA (eRNA) som interagerar med komplexet av mediatorproteiner (se figur), särskilt Mediator-subenhet 12 ( MED12 ), verkar vara väsentligt för att bilda kromosomslingan som för förstärkaren i nära association med målgenens promotor av förstärkaren i fallet med fem gener studerade av Lai et al. Hou och Kraus, beskriver två andra studier som rapporterar liknande resultat. Arnold et al. granska ytterligare 5 fall där eRNA är aktivt för att bilda förstärkar-promotorslingan.
eRNA interagerar med proteiner för att påverka transkriptionen
Ett väl studerat eRNA är eRNA från förstärkaren som interagerar med promotorn för genen för prostataspecifik antigen (PSA). PSA eRNA är starkt uppreglerat av androgenreceptorn . Hög PSA eRNA har då en dominoeffekt. PSA eRNA binder till och aktiverar den positiva transkriptionsförlängningsfaktorn P-TEFb- proteinkomplexet som sedan kan fosforylera RNA-polymeras II (RNAP II), vilket initierar dess aktivitet för att producera mRNA . P-TEFb kan också fosforylera den negativa förlängningsfaktorn NELF (som pausar RNAP II inom 60 nukleotider efter att mRNA-initiering börjar). Fosforylerad NELF frisätts från RNAP II, vilket sedan tillåter RNAP II att ha produktiv mRNA- progression (se figur). Uppreglerat PSA eRNA ökar därigenom uttrycket av 586 androgenreceptorresponsiva gener. Nedbrytning av PSA eRNA eller radering av en uppsättning nukleotider från PSA eRNA orsakar minskad närvaro av fosforylerad (aktiv) RNAP II vid dessa gener, vilket orsakar deras minskade transkription.
Den negativa förlängningsfaktorn NELF-proteinet kan också frigöras från dess interaktion med RNAP II genom direkt interaktion med vissa eRNA. Schaukowitch et al. visade att eRNA från två omedelbara tidiga gener (IEG) direkt interagerade med NELF-proteinet för att frigöra NELF från RNAP II pausade vid promotorerna för dessa två gener, vilket gjorde att dessa två gener sedan kunde uttryckas.
Dessutom verkar eRNA interagera med så många som 30 andra proteiner.
Föreslagna funktionsmekanismer fram till 2013
Föreställningarna om att inte alla förstärkare transkriberas samtidigt och att eRNA- transkription korrelerar med förstärkarspecifik aktivitet stödjer idén att individuella eRNA bär distinkta och relevanta biologiska funktioner. Det finns dock fortfarande ingen konsensus om den funktionella betydelsen av eRNA. Dessutom kan eRNA lätt brytas ned genom exosomer och nonsens-medierat förfall , vilket begränsar deras potential som viktiga transkriptionsregulatorer. Hittills har fyra huvudmodeller av eRNA-funktion föreslagits, var och en med stöd av olika experimentella bevis .
Transkriptionsbrus
Eftersom flera studier har visat att RNA Pol II kan hittas i ett mycket stort antal extragena regioner, är det möjligt att eRNA helt enkelt representerar produkten av slumpmässig "läckande" transkription och inte har någon funktionell betydelse. Den icke-specifika aktiviteten av RNA Pol II skulle därför tillåta extragent transkriptionsbrus på platser där kromatin redan är i ett öppet och transkriptionellt kompetent tillstånd. Detta skulle förklara även vävnadsspecifikt eRNA-uttryck eftersom öppna platser också är vävnadsspecifika.
Transkriptionsberoende effekter
RNA Pol II -medierad gentranskription inducerar en lokal öppning av kromatintillstånd genom rekrytering av histonacetyltransferaser och andra histonmodifierare som främjar eukromatinbildning . Det föreslogs att närvaron av dessa enzymer också skulle kunna inducera en öppning av kromatin vid förstärkarregioner , som vanligtvis finns på avlägsna platser men kan rekryteras till målgener genom looping av DNA . I denna modell uttrycks därför eRNA som svar på RNA Pol II- transkription och har därför ingen biologisk funktion.
Funktionell aktivitet i cis
Medan de två tidigare modellerna antydde att eRNA inte var funktionellt relevanta, säger denna mekanism att eRNA är funktionella molekyler som uppvisar cis -aktivitet. I denna modell kan eRNA lokalt rekrytera regulatoriska proteiner på sin egen syntesplats. Som stöd för denna hypotes tros transkript som härrör från förstärkare uppströms om Cyclin D1 -genen fungera som adaptrar för rekrytering av histonacetyltransferaser . Det visade sig att utarmning av dessa eRNA ledde till Cyclin D1 transkriptionell tystnad.
Funktionell aktivitet i trans
Den sista modellen involverar transkriptionsreglering av eRNA på avlägsna kromosomala platser. Genom differentiell rekrytering av proteinkomplex kan eRNA påverka transkriptionskompetensen hos specifika loci . Evf-2 representerar ett bra exempel på sådant trans- regulatoriskt eRNA eftersom det kan inducera uttrycket av Dlx2, vilket i sin tur kan öka aktiviteten hos Dlx5- och Dlx6- förstärkarna . Transagerande eRNA kan också fungera i cis och vice versa.
Experimentell upptäckt
Detekteringen av eRNA är ganska ny (2010) och har möjliggjorts genom användning av genomomfattande undersökningstekniker som RNA-sekvensering ( RNA-seq ) och kromatinimmunutfällningssekvensering ( ChIP-seq ). RNA-seq tillåter direkt identifiering av eRNA genom att matcha det detekterade transkriptet med motsvarande förstärkarsekvens genom bioinformatiska analyser. ChIP-seq representerar ett mindre direkt sätt att bedöma förstärkartranskription men kan också ge viktig information eftersom specifika kromatinmärken är associerade med aktiva förstärkare . Även om vissa data förblir kontroversiella, är konsensus i litteraturen att den bästa kombinationen av posttranslationella modifieringar av histon vid aktiva förstärkare är gjord av H2AZ , H3K27ac och ett högt förhållande mellan H3K4me1 över H3K4me3 . ChIP- experiment kan också utföras med antikroppar som känner igen RNA Pol II , som kan hittas på platser för aktiv transkription . Den experimentella detekteringen av eRNA kompliceras av deras låga endogena stabilitet tillförd av exosomnedbrytning och nonsens-medierat förfall . En jämförande studie visade att analyser som berikar för capped och begynnande RNA (med strategier som nuclei run-on och storleksval) kunde fånga fler eRNA jämfört med kanoniska RNA-seq . Dessa analyser inkluderar Global/Precision Run-on med cap-selektion (GRO/PRO-cap), capped-small RNA-seq (csRNA-seq), Native Elongating Transcript-Cap Analysis of Gene Expression (NET-CAGE) och Precision Run-On-sekvensering (PRO-seq). Icke desto mindre kan det faktum att eRNA tenderar att uttryckas från aktiva förstärkare göra deras upptäckt till ett användbart verktyg för att skilja mellan aktiva och inaktiva förstärkare .
Implikationer i utveckling och sjukdom
Bevis på att eRNA orsakar nedströmseffekter på effektiviteten av förstärkaraktivering och gentranskription tyder på dess funktionella kapacitet och potentiella betydelse. Transkriptionsfaktorn p53 har visats binda förstärkarregioner och generera eRNA på ett p53 - beroende sätt. I cancer spelar p53 en central roll i tumörsuppression eftersom mutationer av genen visas i 50 % av tumörerna . Dessa p53 -bundna förstärkarregioner (p53BER ) har visat sig interagera med flera lokala och distala genmål involverade i cellproliferation och överlevnad. Dessutom har eRNA som genererats av aktiveringen av p53BERs visat sig krävas för effektiv transkription av p53- målgenerna , vilket indikerar den troliga viktiga reglerande rollen för eRNA i tumörsuppression och cancer .
Variationer i förstärkare har varit inblandade i mänskliga sjukdomar , men ett terapeutiskt tillvägagångssätt för att manipulera förstärkaraktivitet är för närvarande inte möjligt. Med uppkomsten av eRNA som viktiga komponenter i förstärkaraktivitet , kan kraftfulla terapeutiska verktyg som RNAi tillhandahålla lovande vägar för att rikta störningar av genuttryck.