RNA-immunutfällningschip
RIP-chip (RNA immunoprecipitation chip) är en molekylärbiologisk teknik som kombinerar RNA immunoprecipitation med en mikroarray . Syftet med denna teknik är att identifiera vilka RNA- sekvenser som interagerar med ett speciellt RNA-bindande protein av intresse in vivo . Det kan också användas för att bestämma relativa nivåer av genuttryck , för att identifiera delmängder av RNA som kan samregleras eller för att identifiera RNA som kan ha relaterade funktioner. Denna teknik ger insikt i den post-transkriptionella genregleringen som sker mellan RNA och RNA-bindande proteiner.
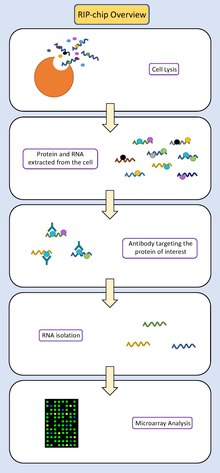
Proceduröversikt
- Samla och lysera de intressanta cellerna .
- Isolera alla RNA-fragment och proteinerna bundna till dem från lösningen.
- Immunoprecipitera proteinet av intresse. Lösningen som innehåller de proteinbundna RNA:n tvättas över pärlor som har konjugerats till antikroppar . Dessa antikroppar är utformade för att binda till proteinet av intresse. De drar proteinet (och eventuella RNA-fragment som är specifikt bundna till det) ut ur lösningen som innehåller resten av cellinnehållet.
- Dissociera det proteinbundna RNA:t från antikropp-pärlkomplexet . Använd sedan en centrifug för att separera det proteinbundna RNA:t från de tyngre antikroppspärlkomplexen, behåll det proteinbundna RNA:t och kassera pärlorna.
- Ta bort RNA från proteinet av intresse .
- Isolera RNA-fragmenten från proteinet med hjälp av en centrifug.
- Använd PCR för omvänd transkription för att omvandla RNA-fragmenten till cDNA (DNA som är komplementärt till RNA-fragmenten).
- Märk dessa cDNA-fragment fluorescerande.
- Förbered genchippet . Detta är ett litet chip som har DNA-sekvenser bundna till sig på kända platser. Dessa DNA-sekvenser motsvarar alla kända gener i arvsmassan hos organismen som forskaren arbetar med (eller en undergrupp av gener som forskaren är intresserad av). De cDNA-sekvenser som har samlats in kommer att vara komplementära till några av dessa DNA-sekvenser, eftersom cDNA representerar en undergrupp av RNA:n som transkriberats från genomet.
- Låt cDNA-fragmenten kompetitivt hybridisera till de DNA-sekvenser som är bundna till chipet .
- Detektion av den fluorescerande signalen från cDNA:t bundet till chippet berättar för forskarna vilka gen(er) på chipet som hybridiserades till cDNA:t.
De gener som fluorescerande identifieras av chipanalysen är de gener vars RNA interagerar med det ursprungliga proteinet av intresse. Styrkan hos den fluorescerande signalen för en viss gen kan indikera hur mycket av det specifika RNA:t som fanns i det ursprungliga provet, vilket indikerar uttrycksnivån för den genen.
Utveckling och liknande tekniker
Tidigare tekniker som syftar till att förstå protein-RNA-interaktioner inkluderade RNA- elektroforetiska mobilitetsförskjutningsanalyser och UV-tvärbindning följt av RT-PCR, men sådan selektiv analys kan inte användas när de bundna RNA:erna ännu inte är kända. För att lösa detta kombinerar RIP-chip RNA-immunfällning för att isolera RNA-molekyler som interagerar med specifika proteiner med en mikroarray som kan belysa identiteten för de RNA som deltar i denna interaktion. Alternativ till RIP-chip inkluderar:
- RIP-seq : Innebär sekvensering av RNA:n som drogs ner med hjälp av högkapacitetssekvensering snarare än att analysera dem med en mikroarray. Författarna Zhao et al., 2010. kombinerade RNA-immunutfällningsproceduren med RNA-sekvensering . Med hjälp av specifika antikroppar (α-Ezh2) immunoprecipiterade de nukleärt RNA isolerat från mus- ES- celler och sekvenserade därefter det neddragna RNA:t med hjälp av nästa generations sekvenseringsplattform, Illumina.
- KLIP : Det RNA-bindande proteinet är tvärbundet till RNA:t via användning av UV-ljus före lysering, vilket följs av RNA-fragmentering, immunoutfällning, tvätt med hög salthalt, SDS-PAGE, membranöverföring, proteinasdigerering, cDNA-biblioteksberedning och sekvensering för att identifiera de direkta RNA-bindningsställena. CLIP har först kombinerats med hög genomströmningssekvensering i HITS-CLIP för att bestämma Nova-RNA-bindningsställen i mushjärnan, och i iCLIP som möjliggjorde amplifiering av trunkerade cDNA och introducerade användningen av UMI.
- ChIP-on-chip : En liknande teknik som detekterar bindningen av proteiner till genomiskt DNA snarare än RNA.