Högupplöst smält
High Resolution Melt ( HRM ) -analys är en kraftfull teknik inom molekylärbiologi för detektion av mutationer , polymorfismer och epigenetiska skillnader i dubbelsträngade DNA- prover. Det upptäcktes och utvecklades av Idaho Technology och University of Utah. Det har fördelar jämfört med andra genotypningsteknologier , nämligen:
- Det är kostnadseffektivt jämfört med andra genotypningsteknologier som sekvensering och TaqMan SNP-typning. Detta gör den idealisk för storskaliga genotypningsprojekt.
- Den är snabb och kraftfull och kan därför snabbt genotypa många prover.
- Det är enkelt. Med en HRM-analys av god kvalitet kan kraftfull genotypning utföras av icke-genetiker i vilket laboratorium som helst med tillgång till en HRM-kapabel realtids-PCR-maskin.
Metod
HRM-analys utförs på dubbelsträngade DNA-prover. Vanligtvis kommer användaren att använda polymeraskedjereaktion (PCR) före HRM-analys för att amplifiera DNA-regionen där deras mutation av intresse ligger. I provröret finns nu många kopior av DNA-området av intresse. Denna region som förstärks är känd som amplikon. Efter PCR-processen börjar HRM-analysen. Processen är helt enkelt en exakt uppvärmning av amplikon-DNA från cirka 50 ˚C upp till cirka 95 ˚C. Vid någon tidpunkt under denna process uppnås amplikonets smälttemperatur och de två DNA-strängarna separeras eller "smälter" isär.
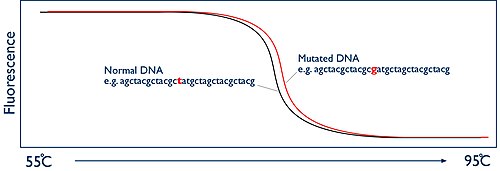
Nyckeln till HRM är att övervaka denna separation av strängar i realtid. Detta uppnås genom att använda ett fluorescerande färgämne. Färgämnena som används för HRM är kända som interkalerande färgämnen och har en unik egenskap. De binder specifikt till dubbelsträngat DNA och när de binds fluorescerar de ljust. I frånvaro av dubbelsträngat DNA har de inget att binda till och de fluorescerar endast på en låg nivå. I början av HRM-analysen är det en hög nivå av fluorescens i provet på grund av miljarder kopior av amplikonet. Men när provet värms upp och de två strängarna av DNA smälter isär, minskar närvaron av dubbelsträngat DNA och därmed minskar fluorescensen. HRM-maskinen har en kamera som övervakar denna process genom att mäta fluorescensen. Maskinen plottar sedan helt enkelt dessa data som en graf känd som en smältkurva, som visar nivån av fluorescens kontra temperaturen:
Jämförelse av smältkurvor
Smälttemperaturen för amplikonen vid vilken de två DNA-strängarna går isär är helt förutsägbar. Det är beroende av DNA-basernas sekvens. Om du jämför två prover från två olika personer bör de ge exakt samma formade smältkurva. Men om en person har en mutation i den DNA-region du har amplifierat, kommer detta att ändra temperaturen vid vilken DNA-strängarna smälter isär. Så nu ser de två smältkurvorna olika ut. Skillnaden kanske bara är liten, kanske en bråkdel av en grad, men eftersom HRM-maskinen har förmågan att övervaka denna process i "hög upplösning" är det möjligt att noggrant dokumentera dessa förändringar och därför identifiera om en mutation är närvarande eller inte .
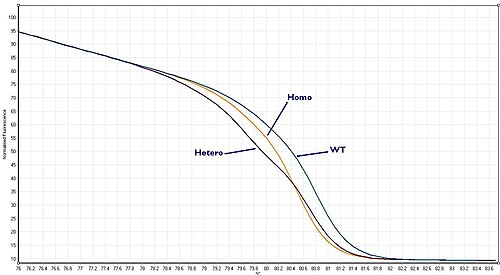
Vildtyp, heterozygot eller homozygot?
Saker och ting blir lite mer komplicerade än så här eftersom organismer innehåller två ( eller fler ) kopior av varje gen, kända som de två allelerna . Så om ett prov tas från en patient och amplifieras med PCR amplifieras båda kopiorna av regionen av DNA (alleler) av intresse. Så om vi letar efter mutation finns det nu tre möjligheter:
- Ingen av allelerna innehåller en mutation
- En eller annan allel innehåller en mutation
- Båda allelerna innehåller en mutation.
Dessa tre scenarier är kända som "Wild-type", "Heterozygote" respektive "Homozygote". Var och en ger en smältkurva som är något annorlunda. Med en HRM-analys av hög kvalitet är det möjligt att skilja mellan alla dessa tre scenarier.
Homozygota allelvarianter kan kännetecknas av en temperaturförskjutning på den resulterande smältkurvan producerad av HRM-analys. I jämförelse kännetecknas heterozygoter av förändringar i formen av smältkurvan. Detta beror på basparsfelmatchning genererad som ett resultat av destabiliserad heteroduplex-annealing mellan vildtyps- och variantsträngar. Dessa skillnader kan lätt ses på den resulterande smältkurvan och smältprofilskillnaderna mellan de olika genotyperna kan förstärkas visuellt genom att generera en skillnadskurva
Ansökningar
SNP-typning/punktmutationsdetektion
Konventionella SNP -typningsmetoder är vanligtvis tidskrävande och dyra, och kräver att flera sondbaserade analyser multiplexeras tillsammans eller användning av DNA-mikroarrayer. HRM är mer kostnadseffektivt och minskar behovet av att designa flera par av primers och behovet av att köpa dyra prober. HRM-metoden har framgångsrikt använts för att detektera en enda G till A-substitution i genen Vssc (Voltage Sensitive Sodium Channel) som ger resistens mot akariciden permetrin i skabbkvalster. Denna mutation resulterar i en kodande förändring i proteinet (G1535D). Analysen av skabbkvalster som samlats in från misstänkta permetrinkänsliga och toleranta populationer av HRM visade distinkta smältprofiler. Amplikonerna från de känsliga kvalstren observerades ha en högre smälttemperatur i förhållande till de toleranta kvalstren, som förväntat från den högre värmestabiliteten hos GC - basparet
Inom ett område som är mer relevant för klinisk diagnostik har HRM visat sig vara i princip lämplig för detektion av mutationer i bröstcancermottaglighetsgenerna BRCA1 och BRCA2. Mer än 400 mutationer har identifierats i dessa gener. Sekvensering av gener är guldstandarden för att identifiera mutationer. Sekvensering är tidskrävande och arbetskrävande och föregås ofta av tekniker som används för att identifiera heteroduplex-DNA, som sedan ytterligare förstärker dessa problem. HRM erbjuder en snabbare och bekvämare metod med slutna rör för att bedöma förekomsten av mutationer och ger ett resultat som kan undersökas ytterligare om det är av intresse. I en studie utförd av Scott et al. 2006 användes 3 cellinjer med olika BRCA-mutationer för att bedöma HRM-metoden. Det visade sig att smältprofilerna för de resulterande PCR-produkterna kunde användas för att särskilja närvaron eller frånvaron av en mutation i amplikonet. På liknande sätt under 2007 Krypuy et al. visade att den noggranna designen av HRM-analyser (med avseende på primerplacering) framgångsrikt kunde användas för att detektera mutationer i TP53-genen, som kodar för tumörsuppressorproteinet p53 i kliniska prover av bröst- och äggstockscancer. Båda dessa studier lyfte fram det faktum att förändringar i smältprofilen kan vara i form av en förskjutning i smälttemperaturen eller en uppenbar skillnad i formen på smältkurvan. Båda dessa parametrar är en funktion av amplikonsekvensen. Konsensus är att HRM är en kostnadseffektiv metod som kan användas som en initial screening för prover som misstänks ha polymorfismer eller mutationer. Detta skulle minska antalet prover som behöver undersökas ytterligare med mer konventionella metoder.
Zygositetstestning
För närvarande finns det många metoder som används för att bestämma zygositetsstatusen för en gen på ett visst ställe. Dessa metoder inkluderar användningen av PCR med specifikt utformade prober för att detektera varianterna av generna (SNP-typning är det enklaste fallet). I de fall där längre sträckor av variation är inblandade kan post PCR-analys av amplikonerna krävas. Förändringar i enzymrestriktion, elektroforetiska och kromatografiska profiler kan mätas. Dessa metoder är vanligtvis mer tidskrävande och ökar risken för amplikonkontamination i laboratoriet, på grund av behovet av att arbeta med höga koncentrationer av amplikoner i labbet efter PCR. Användningen av HRM minskar den tid som krävs för analys och risken för kontaminering. HRM är en mer kostnadseffektiv lösning och det högupplösta elementet tillåter inte bara bestämning av homo och heterozygositet , det löser också information om typen av homo och heterozygositet, med olika genvarianter som ger upphov till olika former av smältkurvor. En studie av Gundry et al. 2003, visade att fluorescerande märkning av en primer (i paret) har visat sig vara gynnsam framför att använda ett interkalerande färgämne såsom SYBR green I . Emellertid har framsteg gjorts i utvecklingen och användningen av förbättrade interkalerande färgämnen som minskar problemet med PCR-hämning och oro över icke-mättande interkalering av färgämnet.
Epigenetik
HRM-metoden har också utnyttjats för att ge en tillförlitlig analys av metyleringsstatusen för DNA. Detta är av betydelse eftersom förändringar i metyleringsstatusen för tumörsuppressorgener, gener som reglerar apoptos och DNA-reparation, är kännetecken för cancer och även har konsekvenser för svar på kemoterapi. Till exempel kan cancerpatienter vara mer känsliga för behandling med DNA-alkyleringsmedel om promotorn för DNA-reparationsgenen MGMT hos patienten är metylerad. I en studie som testade metyleringsstatusen för MGMT- promotorn på 19 kolorektala prover visade sig 8 prover vara metylerade. En annan studie jämförde den prediktiva kraften hos MGMT- promotormetylering i 83 höggradiga gliompatienter erhållna genom antingen MSP , pyrosekvensering och HRM. HRM-metoden visade sig vara åtminstone likvärdig med pyrosekvensering vid kvantifiering av metyleringsnivån.
Metylerat DNA kan behandlas med bi-sulfitmodifiering, som omvandlar icke-metylerade cytosiner till uracil . Därför kommer PCR-produkter som härrör från en mall som ursprungligen var ometylerad att ha en lägre smältpunkt än de som härrör från en metylerad mall. HRM erbjuder också möjligheten att bestämma andelen metylering i ett givet prov, genom att jämföra det med en standardkurva som genereras genom att blanda olika förhållanden av metylerat och icke-metylerat DNA. Detta kan ge information angående graden av metylering som en tumör kan ha och därmed ge en indikation på tumörens karaktär och hur långt den avviker från vad som är "normalt".
HRM är också praktiskt fördelaktigt för användning i diagnostik, på grund av dess förmåga att anpassas till screeningtestning med hög genomströmning, och återigen minimerar det möjligheten för amplikonspridning och kontaminering inom ett laboratorium, på grund av dess stängda rörformat.
Interkalerande färgämnen
För att följa övergången av dsDNA (dubbelsträngat) till ssDNA (enkelsträngat) används interkalerande färgämnen. Dessa färgämnen visar differentiell fluorescensemission beroende på deras association med dubbelsträngat eller enkelsträngat DNA. SYBR Green I är ett första generationens färgämne för HRM. Det fluorescerar när det interkaleras i dsDNA och inte ssDNA. Eftersom det kan hämma PCR vid höga koncentrationer, används det i undermättande koncentrationer. Nyligen har vissa forskare avrådt från användningen av SYBR Green I för HRM och hävdar att betydande protokolländringar krävs. Detta beror på att det antyds att bristen på noggrannhet kan bero på "dye jumping", där färg från en smält duplex kan återinföras i regioner av dsDNA som ännu inte hade smält. Nya mättande färgämnen som LC Green och LC Green Plus, ResoLight, EvaGreen, Chromofy och SYTO 9 finns tillgängliga på marknaden och har använts framgångsrikt för HRM. Vissa grupper har dock framgångsrikt använt SYBR Green I för HRM med Corbett Rotorgene-instrumenten och förespråkar användningen av SYBR Green I för HRM-tillämpningar.
Design av högupplösta smältexperiment
Högupplösta smältanalyser involverar vanligtvis qPCR-amplifiering följt av en smältkurva som samlas in med ett fluorescerande färgämne. På grund av känsligheten hos högupplöst smältanalys är det nödvändigt att noggrant överväga PCR-cykelförhållanden, mall-DNA-kvalitet och smältkurvparametrar. För exakta och repeterbara resultat måste PCR-termiska cyklingsförhållanden optimeras för att säkerställa att den önskade DNA-regionen amplifieras med hög specificitet och minimal bias mellan sekvensvarianter. Smältkurvan utförs vanligtvis över ett brett temperaturområde i små (~0,3 °C) steg som är tillräckligt långa (~10 sekunder) för att DNA ska nå jämvikt vid varje temperatursteg.
Förutom typiska primerdesignöverväganden , innebär designen av primers för högupplösta smältanalyser att maximera de termodynamiska skillnaderna mellan PCR-produkter som tillhör olika genotyper. Mindre amplikoner ger i allmänhet större smälttemperaturvariationer än längre amplikoner, men variabiliteten kan inte förutsägas med ögat. Av denna anledning är det viktigt att noggrant förutsäga smältkurvan för PCR-produkter när man designar primers som kommer att särskilja sekvensvarianter. Specialprogramvara, såsom uMelt och DesignSignatures , är tillgängliga för att hjälpa till att designa primers som kommer att maximera smältkurvans variation specifikt för högupplösta smältanalyser.