GFP-cDNA
GFP -cDNA- projektet dokumenterar lokaliseringen av proteiner till subcellulära avdelningar i den eukaryota cellen med fluorescensmikroskopi . Experimentella data kompletteras med bioinformatiska analyser och publiceras online i en databas . En sökfunktion gör det möjligt att hitta proteiner som innehåller egenskaper eller motiv av särskilt intresse. Rainer Pepperkoks forskargrupper vid European Molecular Biology Laboratory (EMBL) och Stefan Wiemann vid German Cancer Research Center (DKFZ).
Vilka typer av experiment görs?
cDNA från nya identifierade öppna läsramar (ORF) är märkta med Green Fluorescent Protein ( GFP) och uttrycks i eukaryota celler. Därefter registreras den subcellulära lokaliseringen av fusionsproteinerna med fluorescensmikroskopi.
Steg:
1. Storskalig kloning
All storskalig manipulation av ORF kräver kloningsteknologier som är fria från restriktionsenzymer . I detta avseende har de som använder rekombinationskloning (Gateway of Invitrogen eller Creator of BD Biosciences) visat sig vara de mest lämpliga. Denna kloningsteknologi är baserad på rekombinationsmekanismer som används av fager för att integrera deras DNA i värdgenomet . Det gör att ORF:erna snabbt och bekvämt kan skjutas mellan funktionellt användbara vektorer utan behov av konventionell restriktionskloning. I cDNA-GFP-projektet överförs ORF:erna till CFP/YFP- expressionsvektorer . För lokaliseringsanalysen genereras både N- och C-terminala fusioner. Detta maximerar möjligheten att korrekt fastställa lokaliseringen, eftersom närvaron av GFP kan maskera inriktningssignaler som kan finnas i ena änden av det naturliga proteinet.
N-terminal fluorescerande fusioner
Infoga din gen av intresse i MCS uppströms om den fluorescerande proteingenen och uttryck din gen som en fusion till N-terminalen av det fluorescerande proteinet.
C-Terminal Fluorescent Fusions
Infoga din gen av intresse i MCS nedströms den fluorescerande proteingenen och uttryck din gen som en fusion till C-terminalen av det fluorescerande proteinet.
2. Transfektion av eukaryota celler, uttryck
Fusionsvektorerna transfekteras i Vero-celler (apnjurefibroblaster ) . Särskilt intressanta ORF screenas också för lokalisering i PC12-celler och hippocampala neuroner .
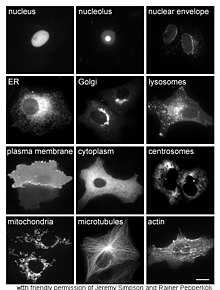
3. Proteinlokalisering
Vid olika tidpunkter registreras den subcellulära lokaliseringen av fusionsproteinerna via fluorescensmikroskopi. I slutet av den levande cellavbildningen kan cellerna fortfarande fixeras och kolokaliseringsexperiment göras.
4. Bioinformatisk analys
Eftersom sekvensen för cDNA:erna är känd kan bioinformatik göra förutsägelser angående lokaliseringen och funktionen av det kodade proteinet. Bioinformatikanalysen underlättas av den bioinformatiska sökmotorn Harvester .
5. Tilldelning av subcellulär lokaliseringskategori
Resultat från de N- och C-terminala fusionerna utvärderas och i sin tur jämförs dessa data med de bioinformatiska förutsägelserna. En slutlig subcellulär lokalisering (från cirka 20 kategorier) tilldelas sedan för varje ORF. Liknande lokaliseringar med både N- och C-terminala konstruktioner ger en högre grad av tillförlitlighet för resultatet. För de ORF:er där de två fusionerna inte ger ett liknande lokaliseringsmönster, övervägs en rad andra kriterier, inklusive bioinformatiska förutsägelser. Ibland kan en tydlig lokalisering inte tilldelas.
Vilken data publiceras?
Varje datablad innehåller fluorescensbilder av både N- och C-terminala fusioner, den tilldelade lokaliseringen, andra lokaliseringar, kommentarer och Swissprot ID. För varje proteininlägg tillhandahålls en länk till motsvarande bioinformatiksida för Harvester .
Hur använder jag GFP-cDNA-databasen?
Bilder av alla lokaliserade proteiner och deras bioinformatiska analys kan ses via knapparna 'Resultattabell' eller 'Resultatbilder'. Använd dessutom sökfönstret på inträdessidan för att hitta proteiner som innehåller egenskaper eller motiv av särskilt intresse för dig som har lokaliserats i detta projekt.