Pseudoaminosyrasammansättning
Pseudoaminosyrasammansättning , eller PseAAC , inom molekylärbiologi , introducerades ursprungligen av Kuo-Chen Chou 2001 för att representera proteinprover för att förbättra förutsägelse av subcellulär lokalisering av protein och förutsägelse av membranproteintyp . Liksom metoden med vaniljaminosyrasammansättning (AAC) karakteriserar den proteinet huvudsakligen med hjälp av en matris av aminosyrafrekvenser, vilket hjälper till att hantera proteiner utan signifikant sekventiell homologi med andra proteiner. Jämfört med AAC ingår även ytterligare information i matrisen för att representera vissa lokala egenskaper, såsom korrelation mellan rester på ett visst avstånd. När man behandlar fallen av PseAAC har Chous invarianssats ofta använts.
Bakgrund
För att förutsäga den subcellulära lokaliseringen av proteiner och andra attribut baserat på deras sekvens, används vanligtvis två typer av modeller för att representera proteinprover: (1) den sekventiella modellen och (2) den icke-sekventiella modellen eller diskreta modellen.
Den mest typiska sekventiella representationen för ett proteinprov är hela dess aminosyrasekvens (AA), som kan innehålla dess mest fullständiga information. Detta är en uppenbar fördel med den sekventiella modellen. För att få de önskade resultaten används vanligtvis de sekvens-likhet-sökning-baserade verktygen för att utföra förutsägelsen.
Givet en proteinsekvens P med aminosyrarester, dvs.
där R1 representerar den första resten av proteinet P , R2 den andra resten och så vidare . Detta är representationen av proteinet under den sekventiella modellen.
Emellertid misslyckas denna typ av tillvägagångssätt när ett frågeprotein inte har signifikant homologi med det eller de kända proteinerna. Således föreslogs olika diskreta modeller som inte förlitar sig på sekvensordning. Den enklaste diskreta modellen är att använda aminosyrasammansättningen (AAC) för att representera proteinprover. Under AAC-modellen kan proteinet P i Eq.1 också uttryckas av
där är de normaliserade förekomstfrekvenserna av de 20 naturliga aminosyrorna i P och T den transponerande operatorn. AAC för ett protein är trivialt härlett med proteinets primära struktur känd som ges i ekv.1 ; det är också möjligt genom hydrolys utan att veta den exakta sekvensen, och ett sådant steg är faktiskt ofta en förutsättning för proteinsekvensering .
På grund av sin enkelhet användes aminosyrasammansättningsmodellen (AAC) flitigt i många tidigare statistiska metoder för att förutsäga proteinattribut. Men all sekvens-ordningsinformation går förlorad. Detta är dess främsta brist.
Begrepp
För att undvika att fullständigt förlora informationen om sekvensordning föreslogs konceptet PseAAC ( pse udo a mino a cid - sammansättning ). Till skillnad från den konventionella aminosyrasammansättningen (AAC) som innehåller 20 komponenter där var och en reflekterar förekomstfrekvensen för en av de 20 naturliga aminosyrorna i ett protein, innehåller PseAAC en uppsättning av mer än 20 diskreta faktorer, där de första 20 representerar komponenterna i dess konventionella aminosyrasammansättning medan de ytterligare faktorerna inkorporerar viss sekvens-ordningsinformation via olika pseudokomponenter.
De ytterligare faktorerna är en serie av rangordnade korrelationsfaktorer längs en proteinkedja, men de kan också vara alla kombinationer av andra faktorer så länge som de kan återspegla någon slags sekvensordningseffekter på ett eller annat sätt. Därför är kärnan i PseAAC att den å ena sidan täcker AA-sammansättningen, men å andra sidan innehåller den informationen bortom AA-sammansättningen och kan därför bättre återspegla funktionen hos en proteinsekvens genom en diskret modell.
Samtidigt har olika sätt att formulera PseAAC-vektorn också utvecklats, vilket sammanfattas i en översiktsartikel från 2009.
Algoritm
Enligt PseAAC-modellen kan proteinet P i Eq.1 formuleras som
där komponenterna ( ) ges av
där är viktfaktorn, och k -th tier-korrelationsfaktorn som återspeglar sekvensordningens korrelation mellan alla -de mest sammanhängande resterna som formulerats av
med
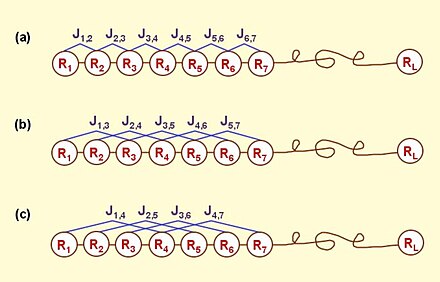
där är den -te funktionen av aminosyran och det totala antalet funktioner som beaktas. Till exempel, i originalpapperet av Chou, Φ och är respektive hydrofobicitetsvärde, hydrofilicitetsvärde och sidokedjemassa för aminosyran ; medan Φ och motsvarande värden för aminosyran . Därför är det totala antalet funktioner som betraktas där . Det kan ses från ekv.3 att de första 20 komponenterna, dvs är associerade med den konventionella AA-sammansättningen av protein, medan de återstående komponenterna är korrelationsfaktorerna som återspeglar 1:a nivån, 2:a nivån, … och -th tier sekvensordningens korrelationsmönster ( Figur 1 ). Det är genom dessa ytterligare faktorer som några viktiga sekvensordningseffekter införlivas.
i ekv.3 är en parameter för heltal och att välja ett annat heltal för kommer att leda till en dimensions-andra PseAA-komposition.
Att använda Eq.6 är bara ett av många sätt att härleda korrelationsfaktorerna i PseAAC eller dess komponenter. De andra, såsom det fysikalisk-kemiska avståndsläget och det amfifila mönsterläget, kan också användas för att härleda olika typer av PseAAC, vilket sammanfattas i en översiktsartikel från 2009. utökades formuleringen av PseAAC ( Eq.3 ) till en form av den allmänna PseAAC som ges av:
där nedsänkningen är ett heltal, och dess värde och komponenterna kommer att bero på hur man extraherar den önskade informationen från aminosyrasekvensen för P i ekv.1 .
Den allmänna PseAAC kan användas för att återspegla alla önskade egenskaper enligt målen för forskning, inklusive de kärnfunktioner som funktionell domän , sekventiell evolution och genontologi för att förbättra förutsägelsekvaliteten för den subcellulära lokaliseringen av proteiner. såväl som deras många andra viktiga egenskaper.







![p_{u}={\begin{cases}{\dfrac {f_{u}}{\sum _{{i=1}}^{{20}}f_{i}\,+\,w\sum _{{k=1}}^{{\lambda }}\tau _{k}}},&(1\leq u\leq 20)\\[10pt]{\dfrac {w\tau _{{u-20}}}{\sum _{{i=1}}^{{20}}f_{i}\,+\,w\sum _{{k=1}}^{{\lambda }}\tau _{k}}},&(20+1\leq u\leq 20+\lambda )\end{cases}}\qquad {\text{(4)}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/9e51062076d0296d9b652c39f3ba5e010aabeb2b)




![{\mathrm {J}}_{{i,i+k}}={\frac {1}{\Gamma }}\sum _{{q=1}}^{{\Gamma }}\left[\Phi _{{q}}\left({\mathrm {R}}_{{i+k}}\right)-\Phi _{{q}}\left({\mathrm {R}}_{{i}}\right)\right]^{2}\qquad {\text{(6)}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/95f4c0827bc12464e0e8ca739608a3a42081c0d2)

















