Perfekt fylogeni
Perfekt fylogeni är en term som används i beräkningsfylogenetik för att beteckna ett fylogenetiskt träd där alla interna noder kan märkas så att alla tecken utvecklas ner i trädet utan homoplasi . Det vill säga egenskaper håller inte för evolutionär konvergens och har inte analoga strukturer. Statistiskt kan detta representeras som en förfader som har tillståndet "0" i alla egenskaper där 0 representerar en avsaknad av den egenskapen. Var och en av dessa egenskaper ändras från 0 till 1 exakt en gång och återgår aldrig till tillstånd 0. Det är sällsynt att faktiska data följer begreppet perfekt fylogeni.
Byggnad
I allmänhet finns det två olika datatyper som används vid konstruktionen av ett fylogenetiskt träd. I avståndsbaserade beräkningar skapas ett fylogenetiskt träd genom att analysera sambanden mellan avståndet mellan arter och kantlängderna på ett motsvarande träd. Genom att använda ett karaktärsbaserat tillvägagångssätt används karaktärstillstånd över arter som en input i ett försök att hitta det mest "perfekta" fylogenetiska trädet.
De statistiska komponenterna i ett perfekt fylogenetiskt träd kan bäst beskrivas enligt följande:
En perfekt fylogeni för en n x m teckentillståndsmatris M är ett rotat träd T med n blad som uppfyller:
i. Varje rad av M märker exakt ett blad av T ii. Varje kolumn av M betecknar exakt en kant av T iii. Varje inre kant av T är märkt med minst en kolumn av M
iv. Tecknen som är associerade med kanterna längs den unika vägen från rot till ett blad v anger exakt teckenvektorn för v , dvs teckenvektorn har en 1-post i alla kolumner som motsvarar tecken associerade till bankanter och en 0-post i övrigt.
Det är värt att notera att det är mycket sällsynt att hitta faktiska fylogenetiska data som följer de begrepp och begränsningar som beskrivs här. Därför är det ofta så att forskare tvingas kompromissa genom att utveckla träd som helt enkelt försöker minimera homoplasi, hitta en maximal kardinalitetsuppsättning av kompatibla karaktärer eller konstruera fylogenier som matchar så nära som möjligt de partitioner som karaktärerna antyder.
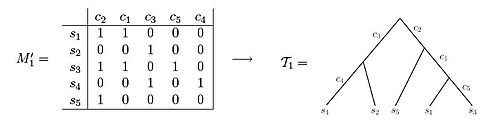
Exempel
Båda dessa datamängder illustrerar exempel på teckentillståndsmatriser . Genom att använda matris M'1 kan man observera att det resulterande fylogenetiska trädet kan skapas så att vart och ett av tecknen märker exakt en kant av trädet . Däremot, när man observerar matrisen M'2, kan man se att det inte finns något sätt att ställa in det fylogenetiska trädet så att varje tecken betecknar endast en kantlängd. Om proverna kommer från data för variant allelfrekvens (VAF) från en population av celler som studeras, är posterna i teckenmatrisen mutationsfrekvenser och tar ett värde mellan 0 och 1. Nämligen, om c i {\ representerar en position i genomet, då kommer posten som motsvarar och prov att hålla frekvenserna av genom i prov med en mutation i position .
- Karaktärstillståndsmatriser
Användande
Perfekt fylogeni är ett teoretiskt ramverk som även kan användas i mer praktiska metoder. Ett sådant exempel är Incomplete Directed Perfect Phylogeny. Detta koncept innebär att man använder perfekta fylogenier med verkliga, och därför ofullständiga och ofullkomliga, datamängder. En sådan metod använder SINEs för att bestämma evolutionär likhet. Dessa korta interspersed element finns över många genom och kan identifieras av deras flankerande sekvenser. SINEs ger information om nedärvning av vissa egenskaper hos olika arter. Tyvärr, om en SINE saknas är det svårt att veta om dessa SINE fanns före raderingen. Genom att använda algoritmer härledda från perfekt fylogenidata kan vi försöka rekonstruera ett fylogenetiskt träd trots dessa begränsningar.
Perfekt fylogeni används också vid konstruktionen av haplotypkartor . Genom att använda de koncept och algoritmer som beskrivs i perfekt fylogeni kan man fastställa information om saknade och otillgängliga haplotypdata. Genom att anta att uppsättningen av haplotyper som är resultatet av genotypkartläggning motsvarar och ansluter sig till begreppet perfekt fylogeni (liksom andra antaganden som perfekt mendelskt arv och det faktum att det bara finns en mutation per SNP), kan man sluta sig till saknade haplotypdata.
Att sluta sig till en fylogeni från bullriga VAF-data under PPM är ett svårt problem. De flesta inferensverktyg inkluderar några heuristiska steg för att göra slutledningar beräkningsmässigt följbara. Exempel på verktyg som härleder fylogenier från bullriga VAF-data inkluderar AncesTree, Canopy, CITUP, EXACT och PhyloWGS. I synnerhet utför EXACT exakta slutledningar genom att använda GPU:er för att beräkna en posterior sannolikhet på alla möjliga träd för problem med liten storlek. Utökningar av PPM har gjorts med tillhörande verktyg. Till exempel tillåter verktyg som MEDICC, TuMult och FISHtrees antalet kopior av ett givet genetiskt element, eller ploidi, att både öka eller minska, vilket effektivt tillåter borttagning av mutationer.
externa länkar
- Lista över fylogenetik programvara
- Ett av flera program tillgängliga för analys och skapande av fylogenetiska träd
- Ett annat sådant program för fylogenetisk trädanalys
- Ytterligare program för trädanalys
- En artikel som beskriver ett exempel på hur perfekt fylogeni kan användas utanför genetikområdet, som i språkföreningar
- Github för "Algorithm for clonal tree rekonstruktion från multi-sample cancer sekvenseringsdata" (AncesTree)
- Github för "Åtkomst av intratumörheterogenitet och spårning av longitudinell och spatial klonal evolutionär historia genom nästa generations sekvensering" (Canopy)
- Github för "Klonalitetsinferens i tumörer som använder fylogeni" (CITUP)
- Github för "Exakt slutledning under den perfekta fylogenimodellen" (EXAKT)
- Github för "Rekonstruera subklonal sammansättning och evolution från helgenomsekvensering av tumörer" (PhyloWGS)