Omvänd vaccinologi
Omvänd vaccinologi är en förbättring av vaccinologi som använder bioinformatik och omvänd farmakologi , pionjär av Rino Rappuoli och först användes mot Serogrupp B meningokocker . Sedan dess har det använts på flera andra bakterievacciner. [ fullständig hänvisning behövs ]
Beräkningsmetod
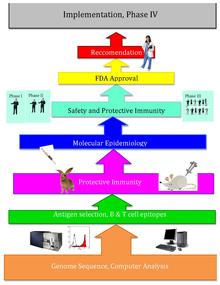
Grundtanken bakom omvänd vaccinologi är att ett helt patogent genom kan screenas med hjälp av bioinformatiska metoder för att hitta gener. Vissa egenskaper som generna övervakas för kan indikera antigenicitet och inkluderar gener som kodar för proteiner med extracellulär lokalisering , signalpeptider och B- cellsepitoper . Dessa gener filtreras för önskvärda egenskaper som skulle göra bra vaccinmål såsom yttre membranproteiner . När kandidaterna väl har identifierats produceras de syntetiskt och screenas i djurmodeller av infektionen.
Historia
Efter att Craig Venter publicerade genomet för den första frilevande organismen 1995, blev arvsmassan från andra mikroorganismer mer lättillgänglig under slutet av 1900-talet. Omvänd vaccinologi, design av vacciner med hjälp av patogenens sekvenserade genom, kom från denna nya mängd genomisk information, såväl som tekniska framsteg. Omvänd vaccinologi är mycket effektivare än traditionell vaccinologi, som kräver odling av stora mängder specifika mikroorganismer såväl som omfattande våta laboratorietester. [ citat behövs ]
År 2000 utvecklade Rino Rappuoli och J. Craig Venter Institute det första vaccinet med användning av omvänd vaccination mot serogrupp B meningokocker. J. Craig Venter Institute och andra fortsatte sedan arbetet med vacciner för A Streptococcus, B Streptococcus, Staphylococcus aureus och Streptococcus pneumoniae.
Omvänd vaccinologi med Meningococcus B
Försök med omvänd vaccinologi började först med Meningococcus B (MenB). Meningokocker B orsakade över 50 % av meningokockmeningit, och forskare hade inte kunnat skapa ett framgångsrikt vaccin för patogenen på grund av bakteriens unika struktur. Denna bakteries polysackaridskal är identiskt med det hos ett mänskligt självantigen, men dess ytproteiner varierar mycket; och bristen på information om ytproteinerna gjorde att det var extremt svårt att utveckla ett vaccin. Som ett resultat vände sig Rino Rappuoli och andra forskare mot bioinformatik för att designa ett funktionellt vaccin.
Rappuoli och andra vid J. Craig Venter Institute sekvenserade först MenB-genomet. Sedan skannade de det sekvenserade genomet efter potentiella antigener. De hittade över 600 möjliga antigener, som testades genom uttryck i Escherichia coli . De mest universellt tillämpliga antigenerna användes i prototypvaccinerna. Flera visade sig fungera framgångsrikt hos möss, men dessa proteiner ensamma interagerade inte effektivt med det mänskliga immunsystemet på grund av att de inte inducerade ett bra immunsvar för att skyddet skulle uppnås. Senare, genom tillsats av yttre membranvesiklar som innehåller lipopolysackarider från rening av blebs på gramnegativa kulturer. Tillsatsen av detta adjuvans (som tidigare identifierats genom att använda konventionella vaccinologiska metoder) förstärkte immunsvaret till den nivå som krävdes. Senare visade sig vaccinet vara säkert och effektivt hos vuxna människor.
Efterföljande omvänd vaccinologisk forskning
Under utvecklingen av MenB-vaccinet antog forskare samma metoder för omvänd vaccination för andra bakteriella patogener. A Streptococcus och B Streptococcus vacciner var två av de första omvända vaccinerna som skapades. Eftersom dessa bakteriestammar inducerar antikroppar som reagerar med mänskliga antigener, behövde vaccinerna för dessa bakterier inte innehålla homologier med proteiner som kodas i det mänskliga genomet för att inte orsaka biverkningar, vilket etablerade behovet av genombaserad omvänd vaccination.
Senare användes Reverse Vaccinology för att utveckla vacciner för antibiotikaresistenta Staphylococcus aureus och Streptococcus pneumoniae
För-och nackdelar
Den stora fördelen med omvänd vaccinologi är att hitta vaccinmål snabbt och effektivt. Traditionella metoder kan ta årtionden att reda ut patogener och antigener, sjukdomar och immunitet. In silico kan dock vara mycket snabb, vilket gör det möjligt att identifiera nya vacciner för testning på bara några år. Nackdelen är att endast proteiner kan målinriktas med denna process. Medan konventionella vaccinologiska metoder kan hitta andra biomolekylära mål såsom polysackarider . [ citat behövs ]
Tillgänglig programvara
Även om användningen av bioinformatisk teknologi för att utveckla vacciner har blivit typiskt under de senaste tio åren, har generella laboratorier ofta inte den avancerade programvaran som kan göra detta. Det finns dock ett växande antal program som gör information om omvänd vaccinologi mer tillgänglig. NERVE är ett relativt nytt databehandlingsprogram. Även om det måste laddas ner och inte inkluderar alla epitopförutsägelser, hjälper det att spara lite tid genom att kombinera beräkningsstegen för omvänd vaccinologi i ett program. Vaxign, ett ännu mer omfattande program, skapades 2008. Vaxign är webbaserat och helt tillgängligt för allmänheten.
Även om Vaxign har visat sig vara extremt exakt och effektiv, använder vissa forskare fortfarande onlineprogramvaran RANKPEP för peptidbindningsförutsägelser. Både Vaxign och RANKPEP använder PSSM (Position Specific Scoring Matrices) vid analys av proteinsekvenser eller sekvensanpassningar.
Datorstödda bioinformatikprojekt blir extremt populära, eftersom de hjälper till att styra laboratorieexperimenten.
Annan utveckling på grund av omvänd vaccinologi och bioinformatik
- Omvänd vaccinologi har orsakat ett ökat fokus på patogen biologi.
- Omvänd vaccinologi ledde till upptäckten av pili i grampositiva patogener som A streptokocker, B streptokocker och pneumokocker. Tidigare trodde man att alla grampositiva bakterier inte hade någon pili.
- Omvänd vaccinologi ledde också till upptäckten av faktor G-bindande protein i meningokocker, som binder till komplementfaktor H hos människor. Bindning till komplementfaktorn H tillåter meningokocker att växa i humant blod samtidigt som alternativa vägar blockeras. Denna modell passar inte många djurarter, som inte har samma komplementfaktor H som människor, vilket indikerar differentiering av meningokocker mellan olika arter.
- ^ Pizza, M.; Rappuoli, R.; Scarlato, V.; Masignani, V.; Giuliani, M.; Aricò, B. (10 mars 2000). "Identifiering av vaccinkandidater mot serogrupp B meningokocker genom helgenomsekvensering" . Vetenskap . 287 (5459): 1816–1820. Bibcode : 2000Sci...287.1816. . doi : 10.1126/science.287.5459.1816 . PMID 10710308 .
- ^ Rappuoli, Rino. Reverse Vaccinology Current Opinion in Microbiology 2000, 3:445–450
- ^ CH Woelk, et al. "Förbättra omvänd vaccinologi med en maskininlärningsmetod." Vaccin 29, nr. 45 (nd): 8156-8164. Science Citation Index, EBSCOhost (tillgänglig 30 september 2012).
- ^ Michalik, Marcin; Djahanshiri, Bardya; Leo, Jack C.; Linke, Dirk (2016), Thomas, Sunil (red.), "Reverse Vaccinology: The Pathway from Genomes and Epitope Predictions to Tailored Recombinant Vaccines", Vaccine Design: Methods and Protocols: Volume 1: Vaccines for Human Diseases , New York, NY: Springer Publishing ; Humana Press , vol. 1403, s. 87–106, doi : 10.1007/978-1-4939-3387-7_4 , ISBN 978-1-4939-3387-7 , PMID 27076126
- ^ a b c d e f g h Alessandro S, Rino R. Recension: Omvänd Vaccinologi: Utveckla vacciner i eran av genomik. Immunitet [serie online]. nd;33:530-541. Tillgänglig från: ScienceDirect , Ipswich, MA. Åtkomst 30 september 2012.
- ^ Rappuoli, R. & A. Aderem. 2011. En vision 2020 för vacciner mot HIV, tuberkulos och malaria. Nature 473: 463.
- ^ He Y, Xiang Z, Mobley H. Vaxign: det första webbaserade vaccindesignprogrammet för omvänd vaccinologi och tillämpningar för vaccinutveckling. Journal of Biomedicine & Biotechnology [serie online]. 2010;Tillgänglig från: CINAHL Plus med fulltext, Ipswich, MA. Åtkomst 30 september 2012.
- ^ Reche PA, Glutting JP och Reinherz EL. Förutsägelse av MHC klass I bindande peptider med hjälp av profilmotiv. Human Immunology 63, 701-709 (2002).
- ^ Sandro V, Jennifer L. G, Francesco F, et al. Recension: Datorstödd bioteknik: från immuninformatik till omvänd vaccinologi. Trends In Biotechnology [serie online]. nd;26:190-200. Tillgänglig från: ScienceDirect, Ipswich, MA. Åtkomst 30 september 2012.