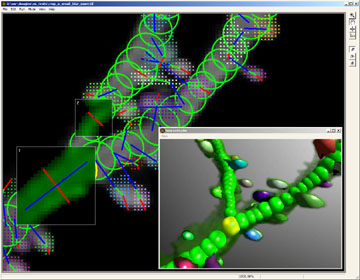
NeuronStudio
NeuronStudio var ett icke-kommersiellt program skapat vid Icahn School of Medicine vid Mount Sinai av Computational Neurobiology and Imaging Center. Detta program utförde automatisk spårning och rekonstruktion av neuronstrukturer från konfokala bildstackar. De resulterande modellerna exporterades sedan till fil med standardformat för vidare bearbetning, modellering eller för statistiska analyser. NeuronStudio hanterade morfologiska detaljer på skalor som spänner över lokal dendritisk ryggradsgeometri genom komplex trädtopologi till det grova rumsliga arrangemanget av multi-neuronnätverk. Dess förmåga till automatiserad digitalisering undvek de subjektiva felen i manuell spårning. Programmet upphörde att stödjas 2012 och projektsidorna togs så småningom bort från ISMMS-webbplatsen. Dess dokumentation och Windows-källkoden är dock fortfarande tillgängliga via Internet Archive .
Dekonvolution
Deconvolution av bilddata är avgörande för korrekta 3D-rekonstruktioner. Deconvolution är ett tillvägagångssätt för bildåterställning där "a priori" kunskap om det optiska systemet i form av en punktspridningsfunktion (PSF) används för att få en bättre uppskattning av objektet. En punktspridningsfunktion kan antingen beräknas från de faktiska mikroskopparametrarna, mätas med pärlor, eller uppskattas och iterativt förfinas ( Blind deconvolution) . PSF kan justeras lokalt för att ta hänsyn till variationer i vävnadens brytningsegenskaper med djup och provegenskaper. För automatiserad användning med stora, kaklade vävnadsblock är detta snabbare och mer exakt än att använda en experimentellt bestämd PSF .
Skelettbildning och diameteruppskattning
Kvantiseringsfel uppstår i standardskelettiseringsalgoritmer från heltalsnaturen hos digitala bilder. Kravet på korrekt representation av fin dendritisk geometri har krävt utvecklingen av nya anpassningar av standardskelettiserings- och diameteruppskattningsalgoritmer för att korrigera för dessa kvantiseringsfel. Iterativa förtunningsskelettiseringsmetoder kan ge ett avstånd i voxlar från varje trädnod till objektets yta. Detta avstånd är D6-metriken, som erhålls genom att räkna antalet voxlar när de tas bort i den minsta 6-anslutna banan från ytan till den mediala axeln. I befintliga skelettiserings- eller vektoriseringsalgoritmer för dendritisk morfometri, är grentvärsnittet vid valfri nod approximativt som cirkulärt, med D6-metriken som ger uppskattningen av en enda diameter. Precisionen för denna diameteruppskattning är begränsad till den fysiska storleken på voxlarna. För små strukturer som tunna dendriter och ryggar, som endast omfattar ett fåtal voxlar även vid maximal bildupplösning, kan felet vara betydande om detta mått används direkt (se figur). För att minimera kvantiseringsfel och mer exakt utvärdera nodernas geometri, finns en ny uppskattningsteknik, Rayburst Sampling Algorithm som använder den ursprungliga gråskaledatan snarare än de segmenterade bilderna för exakt, kontinuerlig radieuppskattning och multiriktad radiesampling för att mer exakt representera icke-cirkulära grentvärsnitt och icke-sfäriska ryggradshuvuden.
Rayburst algoritm
Rayburst-samplingsalgoritmen använder den ursprungliga gråskaledatan snarare än de segmenterade bilderna för exakt, kontinuerlig radieuppskattning och multiriktad radie-sampling för att mer exakt representera icke-cirkulära grentvärsnitt och icke-sfäriska ryggradshuvuden. Algoritmen förberäknar en uppsättning enhetsvektorer som samplar data i flera riktningar (samplingskärnan) från vilken en uppskattning av nodens geometri beräknas. Noggrann representation av varje riktning av provtagningskärnan kräver att de N vektorerna bör vara jämnt fördelade över enhetssfären. Algoritmen använder en partikelfysiksimulering där en uppsättning N av slumpmässigt orienterade enhetsvektorer genereras, vilket resulterar i en slumpmässig, olikformig fördelning av punkter på sfären. Varje partikel får sedan en repulsiv kraft från varannan partikel, proportionell mot den omvända kvadraten på avståndet mellan dem. Genom att iterativt förskjuta partikeln i riktningen för de resulterande krafterna, arrangerar partiklarna sig själva. Detta system kommer att tendera till en stabil, minimal energikonfiguration inom cirka 40 iterationer, där varje partikel är maximalt separerad från sina närmaste grannar. Informationen kan användas för att rekonstruera 3D-grenar av godtyckligt oregelbundna former. Diametern för ett ekvivalent cirkulärt tvärsnitt beräknas i planet vinkelrätt mot den mediala axeln med hjälp av Median Lower Band Diameter (MLBD). För att beräkna MLBD, ta uppsättningen av sampel och lägg till motsvarande par av motsatta vektorer. Sortera vektorerna efter storlek, definiera det nedre bandet som de nedre 50 % och använd avståndet vid position N/4, som representerar medianen för det nedre bandet, för att uppskatta diametern.
Användning av NeuronStudio
NeuronStudio används främst för kvantitativ morfologisk analys av dendritiska ryggraden . Den största fördelen med att använda NeuronStudio-baserad spårning av dendriter och spines är att metoderna baserade på automatisk spårning inte är känsliga för dendriternas drift eller rörelse, eftersom varje dendrit kan spåras separat i varje stack. Detta gör det möjligt att jämföra ryggar från mycket långa inspelningar eller mer åtskilda tidpunkter. Eftersom NeuronStudio-analysen resulterar i exakta 3D-mätningar av ryggradshuvudens bredd och ryggradslängd, är denna metod användbar för att upptäcka förändringar i ryggradens morfologi. Vidare möjliggör användningen av datorbaserad mätning analys av hundratals ryggar på relativt kort tid.
Se även
externa länkar
- Rodriguez, Alfredo (2010-02-18). "Verktyg: NeuronStudio (Beta) Version 0.9.92, senast uppdaterad den 19 november 2009" . CNIC, Mount Sinai School of Medicine. Arkiverad från originalet 2018-09-16.
-
"Källkod för NeuronStudio 0.9.92" . CNIC, Mount Sinai School of Medicine. 2010-09-30. Arkiverad från originalet 2018-07-03.
Härledda verk (programvara baserad på delar av denna källkod) är tillåtna förutsatt att kredit ges [till författaren ] i publiceringen och dokumentationen av sådana verk.
{{ citera webben }}: Extern länk i|quote=
- ^ Rodriguez, A.; Ehlenberger, D.; Kelliher, K.; Einstein, M.; Henderson, SC; Morrison, JH; Hof, PR; Wearne, SL (2003). "Automatisk rekonstruktion av tredimensionell neuronal morfologi från laserskanningsmikroskopbilder". Metoder . 30 (1): 94–105. doi : 10.1016/s1046-2023(03)00011-2 . PMID 12695107 .
- ^ Wearne, SL; Rodriguez, A.; Ehlenberger, DB; Rocher, AB; Henderson, SC; Hof, PR (2005). "Nya tekniker för avbildning, digitalisering och analys av tredimensionell neuronal morfologi på flera skalor". Neurovetenskap . 136 (3): 661–680. CiteSeerX 10.1.1.329.9265 . doi : 10.1016/j.neuroscience.2005.05.053 . PMID 16344143 . S2CID 16510030 .
- ^ Rodriguez, A.; Ehlenberger, D.; Hof, PR; Wearne, SL (2006). "Rayburst sampling, en algoritm för automatiserad tredimensionell formanalys från laserskannande mikroskopibilder". Naturprotokoll . 1 (4): 2156–2161. doi : 10.1038/nprot.2006.313 . PMID 17487207 . S2CID 16141708 .
- ^ Bertling, Enni; Ludwig, Anastasia; Koskinen, Mikko; Hotulainen, Pirta (2012). "Metoder för tredimensionell analys av dendritisk ryggradsdynamik". I Conn, P. Michael (red.). Imaging and Spectroscopic Analysis of Living Cells: Imaging Live Cells in Health and Disease (PDF) . Metoder inom enzymologi. Vol. 506. Elsevier. s. 391–406. doi : 10.1016/B978-0-12-391856-7.00043-3 . ISBN 978-0-12-391856-7 . ISSN 0076-6879 . PMID 22341234 . Hämtad 2021-07-14 .