Hepatit A-virus internt ribosominträdesställe (IRES)
| Hepatit A-virus internt ribosominträdesställe (IRES) | |
|---|---|
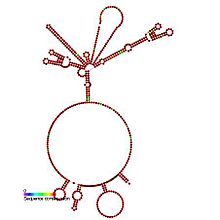 Förutspådd sekundär struktur och sekvenskonservering av IRES_HepA-
| |
| identifierare | |
| Symbol | IRES_HepA |
| Alt. Symboler | HepA_IRES |
| Rfam | RF00228 |
| Övriga uppgifter | |
| RNA -typ | Cis-reg ; IRES |
| Domän(er) | Virus |
| GÅ | GO: 0043022 |
| SÅ | SO:0000243 |
| PDB- strukturer | PDBe |
Denna familj representerar det interna ribosominträdesstället (IRES) av hepatit A-viruset . HAV IRES är en 450 nukleotider lång sekvens belägen i den 735 nt långa 5' UTR (otranslaterad region) av hepatit A viralt RNA-genom . IRES-element tillåter cap- och ändoberoende translation av mRNA i värdcellen. IRES uppnår detta genom att förmedla den interna initieringen av translation genom att rekrytera ett ribosomalt 40S pre-initieringskomplex direkt till initieringskodonet och eliminerar kravet på eukaryotisk initieringsfaktor, eIF4F.
Historia och bakgrund
IRES upptäcktes först i RNA-genomet av picornaviridae av Norman Sonenberg 1988. Inledningsvis kallades den identifierade 5'-UTR av poliovirus (PV) som styrde inre initiering för proteinsyntes ribosomlandningsplatta (RLP). Denna term ersattes med inre ribosominträdesställe strax efter, vilket också är vanligare idag. användes endast encefalomykoarditvirus ( EMCV ) och PV för att visa IRES-förmåga. Forskning som identifierade IRES i HAV gjordes 1993 av Michael J. Glass, Xi-Yu Jia och Donald F. Summers. Deras forskning visade att IRES för HAV var belägen nedströms om nukleotid 45 och inkluderade upp till nukleotid 734. Cap-oberoende intern initiering av proteinsyntes skiljer sig från normal cellulär cap-beroende translationsinitiering. eIF4 (eukaryot initieringsfaktor 4) som ett helt komplex som består av eIF4A, eIF4B, eIF4E och eIF4G. eIF4F används för att referera till komplexet som består av eIF4E, 4G och 4A. I cap-beroende translationsinitiering binder eIF4F till m7G cap på 5' UTR-änden, rekryterar den 40S lilla ribosomala subenheten och skannar nedströms efter AUG-startkodonet. Cap-oberoende intern initiering har visat sig störa antingen delar av eIF4 eller hela komplexet.
Fungera
Obelagt i cytoplasman initierar HAV-genomet translation oberoende av en 5'-kapsel för att syntetisera dess virala proteiner. Den sekundära strukturen av HAV IRES är både nödvändig och tillräcklig för att genomet ska rekrytera en ribosom och initiera translation. En värdcellsribosom känner igen IRES och kommer direkt in i sekvensen snarare än att skanna från 5'-änden. HAV-genomet kodar inte för proteiner som har värdproteinavstängningsförmåga. Därför måste HAV IRES konkurrera med värdcellens m7G-kapslade mRNA. Tyvärr är HAV IRES-initiering av translation inte lika effektiv som en typisk värdcells m7G-cap. Även om HAV IRES-strukturen har affinitet för eIF4F, är dess affinitet inte alls lika hög som värdcellens täckta mRNA. Detta resulterar i en längre period som krävs tills maximal utsöndring av viruset uppnås. Detta resulterar också i att celldöd endast inträffar från värdens immunsvar snarare än lysis.
Undertryckande av IRES
Eftersom eIF4F spelar en viktig roll för HAV IRES-initiering är det ett mål för undertryckande av IRES. Klyvning av eIF4G , en proteinställning av eIF4F, genom sekvensspecifika proteaser 2A-proteas eller L-proteas, kommer att resultera i starkt hämmad HAV IRES-aktivitet. Dessa proteaser kodas av andra medlemmar av familjen picornaviridae . Mul- och klövsjukevirus (FMDV), till exempel, kodar för dessa proteaser för att hämma cellulär mRNA-translation samtidigt som det tillåter att viralt RNA translateras. Kravet på en intakt eIF4G för IRES-initiering är specifikt för HAV IRES bland andra picornavirus. eIF4E-bindande protein I (4E-BP1) kommer också att störa eIF4G-proteinet. 4E-BP1 fungerar genom att sekvestrera eIF4E som därigenom inhiberar dess association med eIF4G och resulterar i HAV IRES-inaktivering (1). En annan metod för att inaktivera eIF4F-aktivitet är genom effekterna av m7GpppG cap-analogen, som riktar sig mot eIF4E och sedan kan förhindra dess association med capped 5'-ändar av mRNA. Den exakta mekanismen i vilken denna cap-analog interfererar med IRES är inte klar, men det föreslås att bindningen av denna analog till eIF4E resulterar i en konformationsförändring av eIF4G vilket är det som stör eIF4G:s normala funktion.
glyceraldehyd-3-fosfat-dehydrogenas (GAPDH)
Glyceraldehyd-3-fosfat-dehydrogenas ( GAPDH ) är ett cellulärt enzym som vanligtvis är involverat i glykolys . GAPDH är känt för att binda till de överlappande platserna inom stamslingan IIIa inom HAV IRES. Stam-loopen IIIa innehåller en UU- nukleotiddeletion inuti en 5-nukleotidsekvens som förstärker IRES-aktiviteten. GAPDH som effektivt binder till denna region kommer att destabilisera den sekundära strukturen som IRES bildar, vilket undertrycker IRES:s förmåga att utföra den cap-oberoende translationen.
La proteinundertryckning
Ett värdcellsprotein som finns allmänt och uteslutande i eukaryota celler, La-protein binder direkt till specifika regioner på HAV IRES under mRNA-translation såväl som RNA-replikation . I en studie från 2008 observerades cytoplasmatisk La reducera HAV IRES-initiering. Men 2014 visade en nyare studie framgångsrik hämning (in vivo) av La-protein som en föreslagen metod för att hämma HAV IRES-translationen och replikeringen, vilket betyder att den mer än sannolikt spelar en integrerad roll i HAV-translationen och replikeringen.
Amantadin
Amantadine , en tricyklisk symmetrisk amin , är en beprövad suppressor som specifikt hämmar den HAV IRES-beroende translationen av HAV-RNA. Ett experiment från 2005 visade att amantadin undertryckte HAV IRES-translation och utlöste inte ett interferonsvar, vilket indikerar lovande antiviral användning av amantadin. För influensa A-virus är dess primära verkningsmetod som ett antiviralt medel att förhindra att det virala genomet tas bort, vilket hämmar den HAV IRES-medierade translationen och replikationen. Amantadines effektivitet härrör från IRES-läget i 5'NTR-regionen som har en hög affinitet för antivirala medel, vilket gör det till ett effektivt mål. Det avslöjades också att M2-proteinet från influensa A-virus kan vara ett annat livskraftigt mål för det potentiella antivirala medlet.
Alla picornavirus har visat sig innehålla IRES. Det finns fyra klasser av IRES inom picornaviridae , från 270–450 nt. Bland picornavirus innehåller många 5' UTR också ytterligare strukturella element uppströms, vilket kan hjälpa det virala genomet i replikationen. Många picornavirus IRES tillåter också att många virus blockerar cap-beroende initiering, vilket resulterar i att värdcellproteinsyntesen stängs av. De fyra klasserna är Entero-/rhinovirus IRES, Cardio-/aftovirus IRES, HAV IRES, HCV-liknande picornavirus IRES. Dessa IRES kategoriseras efter sina nukleotidsekvenser men delar strukturell likhet eftersom det är RNA-strukturen som har förmågan att internt rekrytera translationsmaskineri. Entero-/rhinovirus IRES-element delar vissa strukturella motiv med HAV IRES. HAV IRES, entero-/rhinovirus och cardio-/aptovirus IRES är alla cirka 450 nt men skiljer sig mycket åt i sina strukturer. Ett kardiovirus, EMCV och ett aftovirus, mul- och klövsjukevirus (FMDV) delar ungefär 50 % identiska IRES-element. HCV-liknande picornavirus IRES innehåller den största skillnaden i IRES-element från de andra tre klasserna. Det finns ett brett utbud av picornaviridae -virus som har mycket konserverade HCV-liknande IRES-element, av vilka några förutspås fortfarande vara identifierade. Det är viktigt att notera att HAV IRES-aktivitet skiljer sig från de andra tre klasserna i dess specifika krav på en intakt eIF4G. Andra picornavirus kodar för proteiner som kommer att klyva eIF4G för ökad IRES-aktivitet.