HIKESHI
| HIKESHI | |||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| |||||||||||||||||||||||||||||||||||||||||||||||
| Identifierare | |||||||||||||||||||||||||||||||||||||||||||||||
| , HSPC179, Hikeshi, L7RN6, OPI10, HSPC138, C11orf73, HLD13, kromosom 11 öppen läsram 73, Hikeshi, värmechockprotein nukleär importfaktor, värmechockprotein nukleär importfaktor hikeshi Externa ID: | |||||||||||||||||||||||||||||||||||||||||||||||
| n | |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| Wikidata | |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
HIKESHI är ett protein som är viktigt för lungutveckling och multicellulär organismutveckling som hos människor kodas av HIKESHI -genen . HIKESHI finns på kromosom 11 hos människor och kromosom 7 hos möss. Liknande sekvenser ( ortologer ) finns i de flesta djur- och svamparter. Mushomolog, dödlig gen på kromosom 7 Rinchik 6-protein kodas av l7Rn6 -genen.
Gen
HIKESHI är en proteinkodande gen i Homo sapiens. Alternativa namn för genen är FLJ43020, HSPC138, HSPC179 och L7RN6. Belägen på långa armen av kromosom 11 vid område q14.2, är hela genen inklusive introner och exoner 42 698 baspar på plussträngen. mRNA , med baspar 239 till 832 som representerar de kodande regionerna.
Alternativ skarvning
Variant 1 är den längsta och vanligaste proteinkodande varianten. De tre andra huvudvarianterna använder en alternativ exonsekvens som kastar bort läsramen, vilket orsakar tidig avslutning av mRNA-sekvensen och genomgår proteinsönderfall. Tabellen nedan visar de olika varianterna och exonanvändningen.
| Variant | Exon 1 | Exon 2 | Exon 3 | Exon 4 | Exon 5 | Exon 6 | Exon 7 | Proteinkodning |
|---|---|---|---|---|---|---|---|---|
| 1 | x | x | x | x | x | Ja | ||
| 2 | x | x | x | x | x | x | Nej | |
| 3 | x | x | x | x | x | Nej | ||
| 4 | x | x | x | x | Nej |
De fyra varianterna som visas i tabellen ovan är de vanligaste isoformerna som finns i mänskliga celler. Det finns totalt 13 alternativt splitsade sekvenser och tre osplitsade former som använder två alternativa promotorer. mRNA-varianterna skiljer sig åt på kombinationen av 8 olika exoner, alternativa, överlappande exoner och retentionen av introner . Förutom alternativ splitsning skiljer sig mRNA:n genom trunkering på 3'-änden. Variant 1 är en av tio mRNA som har visat sig koda för ett protein, medan resten verkar bundna till nonsensmedierad mRNA-sönderfall. AceView representation av C11orf73 isoformer 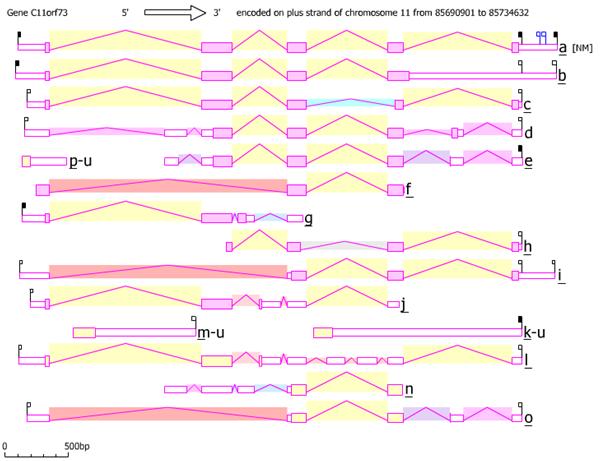
Promotor
Promoter-regionen, GXP 47146, hittades med hjälp av verktyget ElDorado från Genomatix. Sekvensen på 840 bp är belägen före HIKESHI-genen vid DNA-punkterna 86012753 till 86013592. Promotorn är konserverad i 12 av 12 ortologer och kodar för 6 relevanta transkript.
Konserverade transkriptionsfaktorbindningsställen från Genomatix ElDorado-verktyg:
| Detaljerad familjeinformation | Från | Till | Ankare | Orientering | Konserverad i Mus Musculus | Matrix Sim | Sekvens | Förekomst |
|---|---|---|---|---|---|---|---|---|
| Cellcykelregulatorer: Cellcykelhomologielement | 137 | 149 | 143 | + sträng | konserverad | 0,943 | ggacTTGAattca | 1 |
| GATA bindningsfaktorer | 172 | 184 | 178 | + sträng | konserverad | 0,946 | taaAGATttgagg | 1 |
| TATA-bindande proteinfaktor för ryggradsdjur | 193 | 209 | 201 | + sträng | konserverad | 0,983 | tcctaTAAAatttggat | 1 |
| Värmeschockfaktorer | 291 | 315 | 303 | + sträng | konserverad | 0,992 | cacagaaacgttAGAAgcatctctt | 4 |
| Mänskliga och murina ETS1-faktorer | 512 | 532 | 522 | + sträng | konserverad | 0,984 | taagccccGGAAgtacttgtt | 3 |
| Zinkfingertranskriptionsfaktor RU49, Zipro1 | 522 | 528 | 525 | + sträng | konserverad | 0,989 | aAGTAct | 2 |
| Krueppel som transkriptionsfaktorer | 618 | 634 | 626 | + sträng | konserverad | 0,925 | tggaGGGGcagacaccc | 1 |
| SOX/SRY-sex/testisbestämmande och HMG-boxfaktorer | 636 | 658 | 647 | + sträng | konserverad | 0,925 | cccgcaAATTctggaaggttctt | 1 |
Uppsägning
Avslutning av mRNA-produkten kodas för inom genens cDNA. Sluttermineringen av en mRNA-produkt har i allmänhet tre huvuddrag: poly A-signalen, poly A-svansen och ett sekvensområde som kan bilda en stamslingastruktur . Poly A-signalen är ett mycket konserverat ställe, sex nukleotider lång sekvens. I eukaryoter är sekvensen AATAAA och är belägen cirka 10-30 nukleotider från poly A-stället. AATAAA-sekvensen är en mycket konserverad, eukaryotisk polyA-signal som signalerar för polyadenylering av mRNA-produkten 10-30 baspar efter signalsekvensen. PolyA-platsen för C11orf73 är GTA.
Genexpression
HIKESHI bestämdes för att uttryckas allestädes närvarande på en hög nivå på 2,3 gånger över genomsnittet. C11orf73 uttrycks i ett stort antal mänskliga vävnader. Mellan uttrycksprofilerna och EST-profilen på UniGene visades endast 11 vävnader inte uttrycka C11orf73, troligen på grund av små provstorlekar i vävnaden.
Protein
Den mänskliga HIKESHI-genen kodar för ett protein som kallas okarakteriserat protein C11orf73 . Den homologa mus-L7rn6-genen kodar för ett protein som kallas dödlig gen på kromosom 7 Rinchik 6 .
1 mfgclvagrl vqtaaqqvae dkfvfdlpdy esinhvvvfm lgtipfpegm ggsvyfsypd 61 sngmpvwqll gfvtngkpsa ifkisglksg egsqhpfgam nivrtpsvaq igisvelldssvtqn qkmlds qkm favsq aqmtpspsem fipanvvlkw 181 yenfqrrlaq nplfwkt
Det kodade humana proteinet är 197 aminosyror långt och väger 21 628 Dalton. Genom analogi med musproteinet är den hypotetiska funktionen hos det mänskliga HIKESHI-proteinet organisationen och funktionen av den sekretoriska apparaten i lungceller.
| Protein med okänd funktion (DUF775) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identifierare | |||||||||
| Symbol | DUF775 | ||||||||
| Pfam | PF05603 | ||||||||
| InterPro | IPR008493 | ||||||||
| |||||||||
Proteindomänen känd som DUF775 (Domain of Unknown Function 775) är belägen inom både humana HIKESHI- och mus-L7rn6-proteiner . DUF775-domänen är 197 aminosyror lång, samma längd som proteinet. Andra proteiner som utgör superfamiljen DUF 775 per definition inkluderar alla ortologer av C11orf73.
Hydropatianalys visar att det inte finns några omfattande hydrofoba regioner i proteinet och därför dras slutsatsen att HIKESHI är ett cytoplasmatiskt protein. Den isoelektriska punkten för C11orf73 är 5,108 vilket tyder på att den fungerar optimalt i en surare miljö.
SNP
Den enda SNP, eller enkelnukleotidpolymorfismen , för C11orf73-sekvensen resulterar i en aminosyraförändring i proteinet. Bristen på andra SNP:er beror troligen på den höga nivån av bevarande av HIKESHI och den dödliga effekt som en mutation i proteinet ger organismen. Fenotypen för SNP är okänd.
| Fungera | dbSNP Allel | Proteinrester | Kodonposition | Aminosyraposition |
|---|---|---|---|---|
| Referens | C | Proline [P] | 1 | 47 |
| Missense | G | Alanin [A] | 1 | 47 |
Gene Neighborhood
De omgivande generna hos HIKESHI är CCDC81, ME3 och EED . Det genetiska grannskapet tittas på för att få en bättre förståelse för genens möjliga funktion genom att titta på de omgivande genernas funktion.
CCDC81-genen kodar för en okarakteriserad proteinprodukt och är orienterad på plussträngen. CCDC81 står för coiled-coil-domän som innehåller 81 isoform 1.
ME3-genen står för mitochondrial malic enzym 3 precursor. Äppelsyraenzym katalyserar den oxidativa dekarboxyleringen av malat till pyruvat med användning av antingen NAD+ eller NADP+ som en kofaktor. Däggdjursvävnader innehåller 3 distinkta isoformer av äppelenzym: en cytosolisk NADP(+)-beroende isoform, en mitokondriell NADP(+)-beroende isoform och en mitokondriell NAD(+)-beroende isoform. Denna gen kodar för en mitokondriell NADP(+)-beroende isoform. Flera alternativt splitsade transkriptvarianter har hittats för denna gen, men den biologiska giltigheten för vissa varianter har inte fastställts.
EED-genen står för embryonal ektodermutveckling isoform b och är en medlem av familjen Polycomb-grupp ( PcG). PcG-familjemedlemmar bildar multimera proteinkomplex, som är involverade i att upprätthålla det transkriptionella repressiva tillståndet för gener under successiva cellgenerationer. Detta protein interagerar med förstärkaren av zeste 2, den cytoplasmatiska svansen av integrin beta7, immunbristvirus typ 1 ( HIV -1) MA-protein och histon-deacetylasproteiner. Detta protein förmedlar repression av genaktivitet genom histon-deacetylering och kan fungera som en specifik regulator av integrinfunktion. Två transkriptvarianter som kodar för distinkta isoformer har identifierats för denna gen.
Interaktioner
Programmen STRING och Sigma-Aldrichs favoritgen föreslog möjliga proteininteraktioner med C11orf73. ARGUL1, CRHBP och EED härleddes från textmining och HNF4A kom från Sigma-Aldrich.
| Protein | Beskrivning | Metod | Göra |
|---|---|---|---|
| ARGUL1 | Okänd | Textmining | 0,712 |
| CRHBP | Kortikotropinfrisättande hormonbindande protein | Textmining | 0,653 |
| EED | Embryonal ektodermutveckling | Textmining | 0,420 |
| HNF4A | Transkriptionsregulator | Sigma-Aldrich | N/A |
ARGUL1 är ett okänt protein med en okänd funktion. CRHBP är ett kortikotrofinfrisättande hormonbindande protein som möjligen kan spela en roll i en signalkaskad som involverar eller aktiverar HIKESHI. EED, ett närliggande protein till C11orf73, är ett embryonalt ektodermutvecklingsprotein och är en medlem av familjen Polycomb-grupp (PcG). PcG-familjemedlemmar bildar multimera proteinkomplex, som är involverade i att upprätthålla det transkriptionella repressiva tillståndet för gener under successiva cellgenerationer. HNF4A är en transkriptionsregulator och det är okänt om HNF4A reglerar C11orf73s uttryck eller helt enkelt interagerar med det. [12
Evolutionshistoria
Organismernas evolutionära historia kan bestämmas med hjälp av ortologernas sekvenser som tidsreferenser för att skapa ett fylogenetiskt träd. CLUSTALW jämför flera sekvenser, programmet kan också användas för att skapa ett sådant fylogenetiskt träd baserat på ortologerna av C11orf73. Trädet till höger visar det genererade fylogenetiska trädet med en tidslinje baserad på tidpunkten för divergens. Trädet tillverkat av HIKESHI-ortologerna är identiskt med litteraturens fylogenetiska träd och grupperar till och med liknande organismer som fiskar, fåglar och svampar.
Ortologer
Homologa sekvenser är ortologa om de separerades av en artbildningshändelse: när en art divergerar till två separata arter, sägs de divergerande kopiorna av en enda gen i den resulterande arten vara ortologa. Ortologer, eller ortologa gener, är gener i olika arter som liknar varandra eftersom de härstammar från en gemensam förfader. Ortologa sekvenser ger användbar information i taxonomisk klassificering och fylogenetiska studier av organismer. Mönstret av genetisk divergens kan användas för att spåra organismers släktskap. Två organismer som är mycket nära besläktade kommer sannolikt att visa mycket lika DNA-sekvenser mellan två ortologer. Omvänt kommer sannolikt en organism som avlägsnas evolutionärt från en annan organism att uppvisa en större divergens i sekvensen av ortologerna som studeras.
Tabell över kromosom 11 öppen läsram 73 ortologer
| Arter | Vanligt namn | Proteinnamn | Tillträdesnummer | NT Längd | NT identitet | AA Längd | AA identitet | E-värde |
|---|---|---|---|---|---|---|---|---|
| Homo sapiens | Mänsklig | C11orf73 | NM_016401 | 1187 bp | 100 % | 197 aa | 100 % | 0 |
| Bon taurus | Ko | LOC504867 | NP_001029398 | 996 bp | 73,60 % | 197 aa | 98 % | 5.30E-84 |
| Mus musculus | Mus | l7Rn6 | NP_080580 | 1045 bp | 72,90 % | 197 aa | 97 % | 4.80E-83 |
| Gallus gallus | Kyckling | LOC427034 | N/A | 851 bp | 56,20 % | 197 aa | 88,3 % | 5.60E-76 |
| Taeniopygia guttata | Zebrafink | LOC100190155 | ACH44077 | 997 bp | 61,60 % | 997 aa | 87,80 % | 1.20E-75 |
| Xenopus laevis | Groda | MGC80709 | NP_001087012 | 2037 bp | 36,50 % | 197 aa | 86,80 % | 1.70E-75 |
| Oncorhynchus mykiss | Regnbågsforell | CK073 | NP_001158574 | 940 bp | 52,20 % | 197 aa | 75,10 % | 2.70E-66 |
| Tetradon nigroviridis | Tetradon | icke namngiven proteinprodukt | CAF89643 | N/A | N/A | 197 aa | 70,90 % | 1.40E-61 |
| Trichoplax adhaerens | Trichoplax adhaerens | TRIADDRAFT_19969 | XP_002108733 | 600 bp | 33,10 % | 199 aa | 52,30 % | 2.00E-47 |
| Culex quinquefasciatus | Mygga | konserverat hypotetiskt protein | XP_001843282 | 594 bp | 30,70 % | 197 aa | 49,30 % | 2.50E-41 |
| Drosophilia melanogaster | Flyga | CG13926 | NP_647633 | 594 bp | 31,50 % | 197 aa | 48,50 % | 4.50E-39 |
| Laccaria bicolor | Svamp | förutspått protein | XP_001878996 | 696 bp | 36,40 % | 202 aa | 35,20 % | 8.30E-24 |
| Candida albicans | Svampar | CaO19,13758 | XP_716157 | 666 bp | 36,10 % | 221 aa | 24 % | 5.70E-11 |
Tabellen visar de 13 sekvenserna (12 ortologer, 1 originalsekvens) tillsammans med proteinnamn, accessionsnummer, nukleotididentitet, proteinidentitet och E-värden. Accessionsnumren är identifikationsnumren från NCBI Protein-databasen. Nukleotidsekvensen kan nås från proteinets sekvenssida från DBSOURCE, som ger accessionsnumret och är en länk till nukleotidens sekvenssida. Längden på både nukleotid- och proteinsekvensen för varje ortolog och dess respektive organism anges också i tabellen. Bredvid sekvenslängderna finns identiteterna för ortologen till den ursprungliga HIKESHI-genen. Identiteterna och E-värdena förvärvades med hjälp av det globala anpassningsprogrammet, ALIGN, från SDSC Biology Workbench och BLAST från NCBI.
Grafen visar ortologens procentuella identitet mot organismens divergenstid för att producera en mestadels linjär kurva. De två huvudlederna inom kurvan antyder tider för genduplicering, för cirka 450 miljoner år respektive 1150 miljoner år sedan. Paralogerna från genduplikationerna är förmodligen så olika från de mycket konserverade ortologerna från HIKESHI att de inte hittades med hjälp av Blink- eller BLAST-verktygen.
Värdet m (totalt antal aminosyraförändringar som har inträffat i ett 100 aminosyrors segment), som är det korrigerade värdet av n (antal aminosyraskillnader från mallsekvensen), används också för att beräkna λ (genomsnittlig aminosyra). syraförändringar per år, vanligtvis representerade i värden på λE9).
m/100 = –ln(1-n/100) λ = (m/100)/(2*T)
externa länkar
- Human C11orf73- genomplacering och C11orf73- geninformationssida i UCSC Genome Browser .











