GLIMMA
| Utvecklare | Steven Salzberg och Arthur Delcher |
|---|---|
| Stabil frisättning | 3.02 / 9 maj 2006
|
| Tillgänglig i | C++ |
| Typ | Bioinformatikverktyg _ |
| Licens | OSI-certifierad programvara med öppen källkod under Artistic License |
| Hemsida |
Inom bioinformatik används GLIMMER (Gene Locator and Interpolated Markov ModelER) för att hitta gener i prokaryot DNA . "Det är effektivt för att hitta gener i bakterier , archea , virus , typiskt hitta 98-99% av alla relativt långa proteinkodande gener" . GLIMMER var det första systemet som använde den interpolerade Markov-modellen för att identifiera kodningsregioner. GLIMMER-programvaran är öppen källkod och underhålls av Steven Salzberg , Art Delcher och deras kollegor vid Center for Computational Biology vid Johns Hopkins University . De ursprungliga GLIMMER-algoritmerna och mjukvaran designades av Art Delcher, Simon Kasif och Steven Salzberg och tillämpades på bakteriegenomnotering i samarbete med Owen White .
versioner
GLIMMER 1.0
Den första versionen av GLIMMER "dvs, GLIMMER 1.0" släpptes 1998 och den publicerades i tidningen Microbial genidentifiering med hjälp av interpolerad Markov-modell . Markov-modeller användes för att identifiera mikrobiella gener i GLIMMER 1.0. GLIMMER tar hänsyn till de lokala kompositionssekvensberoendena, vilket gör GLIMMER mer flexibel och kraftfullare jämfört med Markov-modellen med fast ordning .
Det gjordes en jämförelse mellan interpolerad Markov-modell som används av GLIMMER och femte ordningens Markov-modell i papperet Microbial genidentifiering med hjälp av interpolerade Markov-modeller . "GLIMMER-algoritmen hittade 1680 gener av 1717 kommenterade gener i Haemophilus influenzae där femte ordningens Markov-modell hittade 1574 gener. GLIMMER hittade 209 ytterligare gener som inte ingick i 1717 kommenterade gener där femte ordningens Markov-modell hittade 104-genen."
GLIMMER 2.0
Andra versionen av GLIMMER, dvs. GLIMMER 2.0 släpptes 1999 och den publicerades i tidningen Improved microbial identification with GLIMMER . Detta dokument ger betydande tekniska förbättringar som att använda interpolerad kontextmodell istället för interpolerad Markov-modell och att lösa överlappande gener som förbättrar noggrannheten hos GLIMMER.
Interpolerade kontextmodeller används istället för interpolerade Markov-modeller som ger flexibiliteten att välja vilken bas som helst. I interpolerad Markov-modell bestäms sannolikhetsfördelningen av en bas från de omedelbart föregående baserna. Om den omedelbart föregående basen är irrelevant aminosyratranslation , överväger den interpolerade Markov-modellen fortfarande den föregående basen för att bestämma sannolikheten för en given bas där en interpolerad kontextmodell som användes i GLIMMER 2.0 kan ignorera irrelevanta baser. Falskt positiva förutsägelser ökades i GLIMMER 2.0 för att minska antalet falskt negativa förutsägelser. Överlappande gener löses också i GLIMMER 2.0.
Olika jämförelser mellan GLIMMER 1.0 och GLIMMER 2.0 gjordes i tidningen Förbättrad mikrobiell identifiering med GLIMMER som visar förbättringar i den senare versionen. "Känsligheten för GLIMMER 1.0 varierar från 98.4 till 99.7% med ett genomsnitt på 99.1%, medan GLIMMER 2.0 har ett känslighetsområde från 98.6 till 99.8% med ett genomsnitt på 99.3%. GLIMMER 2.0 är mycket effektiv för att hitta gener med hög densitet. parasiten Trypanosoma brucei , ansvarig för att orsaka afrikansk sömnsjuka, identifieras av GLIMMER 2.0"
GLIMMER 3.0
Tredje versionen av GLIMMER, "GLIMMER 3.0" släpptes 2007 och den publicerades i tidningen Identifying bacterial genes and endosymbiont DNA with Glimmer . Detta dokument beskriver flera stora förändringar som gjorts i GLIMMER-systemet inklusive förbättrade metoder för att identifiera kodande regioner och starta kodon . Poängsättning av ORF i GLIMMER 3.0 görs i omvänd ordning, dvs. börjar från stoppkodonet och går tillbaka mot startkodonet. Omvänd scanning hjälper till att identifiera den kodande delen av genen mer exakt som finns i IMMs sammanhangsfönster. GLIMMER 3.0 förbättrar också den genererade träningsdatan genom att jämföra lång-ORF med universell aminosyrafördelning av vitt skilda bakteriegenom." -ORF-utgång på 39 %."
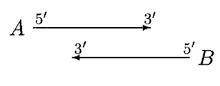
GLIMMER 3.0 minskar antalet falskt positiva förutsägelser som ökades i GLIMMER 2.0 för att minska antalet falskt negativa förutsägelser. "GLIMMER 3.0 har en prediktionsnoggrannhet på startplats på 99,5 % för 3'5'-matchningar, medan GLIMMER 2.0 har 99,1% för 3'5'-matchningar. GLIMMER 3.0 använder en ny algoritm för att skanna kodningsregioner, en ny startplatsdetekteringsmodul , och arkitektur som integrerar alla genförutsägelser över ett helt genom."
Teoretisk och biologisk grund
GLIMMER-projektet hjälpte till att introducera och popularisera användningen av modeller med variabel längd inom beräkningsbiologi och bioinformatik som sedan har tillämpats på många problem såsom proteinklassificering och andra. Variabel längdmodellering var ursprungligen banbrytande av informationsteoretiker och användes och populariserades sedan genialiskt inom datakomprimering (t.ex. Ziv-Lempel-komprimering). Förutsägelse och komprimering är intimt kopplade med minimibeskrivningslängdprinciper . Grundtanken är att skapa en ordbok över vanliga ord (motiv i biologiska sekvenser). Intuitionen är att de ofta förekommande motiven sannolikt är mest prediktiva och informativa. I GLIMMER är den interpolerade modellen en blandningsmodell av sannolikheterna för dessa relativt vanliga motiv. I likhet med utvecklingen av HMMs inom beräkningsbiologi, påverkades författarna till GLIMMER konceptuellt av den tidigare tillämpningen av en annan variant av interpolerade Markov-modeller på taligenkänning av forskare som Fred Jelinek (IBM) och Eric Ristad (Princeton). Inlärningsalgoritmen i GLIMMER skiljer sig från dessa tidigare tillvägagångssätt.
Tillgång
GLIMMER kan laddas ner från The Glimmers hemsida (kräver en C++- kompilator ). Alternativt är en onlineversion värd av NCBI [1] .
Hur det fungerar
- GLIMMER söker i första hand efter långa ORFS . En öppen läsram kan överlappa med vilken annan öppen läsram som helst som kommer att lösas med den teknik som beskrivs i underavsnittet. Genom att använda dessa långa ORFS och följa viss aminosyrafördelning genererar GLIMMER träningsuppsättningsdata .
- Med hjälp av dessa träningsdata tränar GLIMMER alla sex Markov-modeller av kodande DNA från noll till åtta ordning och tränar även modellen för icke-kodande DNA
- GLIMMER försöker beräkna sannolikheterna från data. Baserat på antalet observationer avgör GLIMMER om Markov-modellen med fast ordning eller interpolerad Markov-modell ska användas.
- Om antalet observationer är större än 400, använder GLIMMER fast ordning Markov-modellen för att erhålla sannolikheter.
- Om antalet observationer är mindre än 400 använder GLIMMER en interpolerad Markov-modell som kortfattat förklaras i nästa underavsnitt.
- GLIMMER erhåller poäng för varje lång-ORF som genereras med alla sex kodande DNA-modeller och även med icke-kodande DNA-modell.
- Om poängen som erhölls i föregående steg är större än en viss tröskel då förutspår GLIMMER att det är en gen.
De steg som förklaras ovan beskriver den grundläggande funktionaliteten hos GLIMMER. Det finns olika förbättringar av GLIMMER och några av dem beskrivs i följande underavsnitt.
GLIMMER-systemet
GLIMMER-systemet består av två program. Det första programmet kallas build-imm, som tar en ingångsuppsättning av sekvenser och matar ut den interpolerade Markov-modellen enligt följande.
Sannolikheten för varje bas dvs A,C,G,T för alla k-merer för 0 ≤ k ≤ 8 beräknas. Sedan, för varje k-mer , beräknar GLIMMER vikt. Sannolikhet för ny sekvens beräknas enligt följande.
där n är längden på sekvensen är oligomeren i position x. , den -ordningens interpolerade Markov-modellpoängen beräknas som
"där är vikten av k-mer vid position x-1 i sekvensen S och är uppskattningen som erhålls från träningsdata av sannolikheten för basen placerad vid position x i k -ordningsmodellen. "
Sannolikheten för basen givet i föregående baser beräknas enligt följande.
"Värdet på associerat med kan betraktas som ett mått på förtroende för detta värdes noggrannhet som en uppskattning av den sanna sannolikheten. GLIMMER använder två kriterier för att bestämma Det första av dessa är enkel frekvens förekomst där antalet förekomster av kontextsträngen i träningsdata överstiger ett specifikt tröskelvärde, sedan är satt till 1.0. Det nuvarande standardvärdet för tröskel är 400, vilket ger 95 % konfidens. När det inte finns tillräckligt många exempelförekomster av en kontextsträng använder build-imm ytterligare kriterier för att bestämma -värde. För en given kontextsträng av längden i, build-imm jämför de observerade frekvenserna för följande bas , , , med de tidigare beräknade interpolerade Markov-modellsannolikheterna med hjälp av nästa kortare sammanhang , , , I . Med hjälp av ett -test, avgör build-imm hur troligt det är att de fyra observerade frekvenserna överensstämmer med IMM-värdena från nästa kortare sammanhang."
Det andra programmet som kallas glimmer, använder sedan denna IMM för att identifiera förmodad gen i ett helt genom. GLIMMER identifierar alla öppna läsramar som ger högre värde än tröskeln och kontrollerar om det finns överlappande gener. Att lösa överlappande gener förklaras i nästa underavsnitt.
Ekvationer och förklaringar av termerna som används ovan är hämtade från artikeln 'Microbial genidentification using interpolated Markov-modeller
Lösa överlappande gener
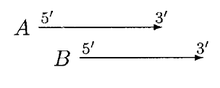
I GLIMMER 1.0, när två gener A och B överlappar varandra, poängsätts överlappningsregionen. Om A är längre än B, och om A får högre poäng på överlappningsområdet, och om flyttning av B:s startplats inte kommer att lösa överlappningen, så avvisas B.
GLIMMER 2.0 gav en bättre lösning för att lösa överlappningen. I GLIMMER 2.0, när två potentiella gener A och B överlappar varandra, poängsätts överlappningsregionen. Anta att gen A får högre poäng, fyra olika orienteringar beaktas.
I ovanstående fall tar inte flyttning av startplatser bort överlappningen. Om A är betydligt längre än B, så avvisas B eller så kallas både A och B gener, med en tveksam överlappning.
I ovanstående fall kan flyttning av B lösa överlappningen, A och B kan kallas icke överlappande gener men om B är betydligt kortare än A, avvisas B.
I ovanstående fall kan flyttning av A lösa överlappningen. A flyttas bara om överlappningen är en liten del av A eller annars förkastas B.
I ovanstående fall kan både A och B flyttas. Vi flyttar först början av B tills överlappningsområdet får högre poäng för B. Sedan flyttar vi början av A tills det får högre poäng. Sedan B igen, och så vidare, tills antingen överlappningen är eliminerad eller inga ytterligare drag kan göras.
Ovanstående exempel har hämtats från artikeln 'Identifiera bakteriella gener och endosymbiont DNA med Glimmer'
Ribosombindningsställen
Ribosombindningsställe (RBS)-signal kan användas för att hitta sann startplatsposition. GLIMMER-resultat skickas som en ingång för RBSfinder-programmet för att förutsäga ribosombindningsställen. GLIMMER 3.0 integrerar RBSfinder-programmet i själva genförutsägande funktionen.
ELPH-programvara (som fastställdes vara mycket effektiv för att identifiera RBS i tidningen) används för att identifiera RBS och är tillgänglig på denna webbplats . Gibbs samplingsalgoritm används för att identifiera delade motiv i vilken uppsättning sekvenser som helst. Dessa delade motivsekvenser och deras längd ges som input till ELPH. ELPH beräknar sedan positionsviktsmatrisen (PWM) som kommer att användas av GLIMMER 3 för att poängsätta eventuella RBS som hittats av RBSfinder. Ovanstående process görs när vi har en betydande mängd träningsgener. Om det finns ett otillräckligt antal träningsgener kan GLIMMER 3 starta upp sig själv för att generera en uppsättning genförutsägelser som kan användas som input till ELPH. ELPH beräknar nu PWM och denna PWM kan återigen användas på samma uppsättning gener för att få mer exakta resultat för startplatser. Denna process kan upprepas i många iterationer för att få mer konsekventa PWM- och genförutsägelseresultat.
Prestanda
Glimmer stödjer genomannoteringsinsatser på ett brett spektrum av bakterie-, arkeala och virala arter. I en storskalig reannoteringsinsats vid DNA Data Bank of Japan (DDBJ, som speglar Genbank ). Kosuge et al. (2006) undersökte metoderna för att hitta genen som används för 183 genom. De rapporterade att av dessa projekt var Glimmer genfinnaren för 49 %, följt av GeneMark med 12 %, med andra algoritmer som användes i 3 % eller färre av projekten. (De rapporterade också att 33% av genomen använde "andra" program, vilket i många fall innebar att de inte kunde identifiera metoden. Exklusive de fallen användes Glimmer för 73% av de genomen för vilka metoderna entydigt kunde identifieras. ) Glimmer användes av DDBJ för att återannotera alla bakteriegenom i International Nucleotide Sequence Databases. Den används också av denna grupp för att kommentera virus. Glimmer är en del av den bakteriella annoteringspipelinen vid National Center for Biotechnology Information (NCBI), som också har en webbserver för Glimmer, liksom webbplatser i Tyskland, Kanada.
Enligt Google Scholar har från och med början av 2011 den ursprungliga Glimmer-artikeln (Salzberg et al., 1998) citerats 581 gånger, och Glimmer 2.0-artikeln (Delcher et al., 1999) har citerats 950 gånger.
externa länkar
- Glimmers hemsida på CCB, Johns Hopkins University, från vilken programvaran kan laddas ner.




![{\displaystyle \operatorname {IMM_{k}(S_{x})=Y_{k}(S_{x-1})\cdot P_{k}(S_{x})+[1-{Y_{k}(S_{x-1})]\cdot IMM_{k-1}(S_{x})}} }](https://wikimedia.org/api/rest_v1/media/math/render/svg/2d303028692d57e04cae6e304ca63cd7db9b1181)



![{\displaystyle \operatorname {P_{i}(S_{x})=P(s_{x}|S_{x,j})={\frac {f(S_{x,j})}{\sum _{b\in {[acgt]}}\operatorname {f(S_{x,i},b)} }}} }](https://wikimedia.org/api/rest_v1/media/math/render/svg/0ed6988a29b7c1f3de8c00efa5f2bd50e895dc06)