ZC3H12B
| ZC3H12B | |||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Identifiers | |||||||||||||||||||||||||||||||||||||||||||||||
| , CXorf32, MCPIP2, zinkfinger CCCH-typ som innehåller 12B | |||||||||||||||||||||||||||||||||||||||||||||||
| externa ID :n | |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| Wikidata | |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
ZC3H12B , även känd som CXorf32 eller MCPIP2 , är ett protein som kodas av genen ZC3H12B lokaliserat på kromosom Xq12 hos människor.
Gen
ZC3H12B-genen är sammansatt av 19 709 baspar (bp) och innehåller 5 exoner . Den är belägen på X-kromosomen vid q12 på plussträngen.
ZC3H12B innehåller en ribonukleasdomän , såväl som en zinkfingerdomän av CCCH-typ. Ribonukleaser (RNaser) bryter ner RNA och är involverade i RNA-mognadsprocessen. De är också en försvarslinje mot viralt RNA (D'Alessio och Riordan 1997). Zinkfingrar av CCCH-typ är associerade med mRNA-destabilisering. Zinkfingrar av CCCH-typ har visat sig omvandla mRNA utan att PolyA-svansen tas bort (Lai och Blackshear 2001). ZC3H12B och dess paraloger ZC3H12A, ZC3H12C och ZC3H12D innehåller alla zinkfingerdomäner av CCCH-typ, som har associerats med cellcykel- och tillväxtfasövergångar i eukaryoter (InterPro).
Promotor
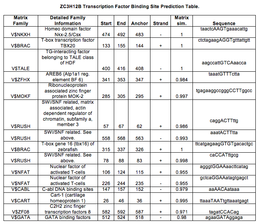
Genomatix ElDorado-programmet förutspådde en 601 bp promotor uppströms om ZC3H12B-genen med flera transkriptionsfaktorbindningsställen inklusive nukleär faktor av aktiverade T-celler och ribonukleoproteinassocierat zinkfingerprotein MOK-2 (även känt som ZNF239 ) .
mRNA
ZC3H12B innehåller 7273 bp mRNA. Det finns bara en förutspådd transkription av Aceview. Inga vikningsmönster har förutspåtts (Mfold). Det finns för introner utskurna från ZC3H12B.
Protein
ZC3H12B är ett troligt ribonukleas innehållande CCCH-typ zinkfingerdomän och ribonukleasdomäner. Proteinet med 836 aminosyror har en förutspådd molekylvikt på 94,2 kdal. Den innehåller inte en signalpeptid eller en transmembranregion. PSORTII förutspådde 65,2 % sannolikhet för nukleär lokalisering. Zinkfingrar och ribonukleaser av CCCH-typ är förmodligen lokaliserade i kärnan för RNA-klyvning och specifikt RNA-hårnålsklyvning (Boysen och Hearn 2008).
Strukturella egenskaper
Proteinets sekundära struktur är en blandning av alfaspiraler och betasträngar. De två domänerna som hittills identifierats är ribonukleas- och zinkfingerdomänerna av CCCH-typ.
Nedan visas en konserverad domän av ZC3H12B paralog, Mcpip1 (eller ZC3H12A). I en BLAST-strukturjämförelse fanns det en 82 % identitetsmatchning med 24 % frågetäckning, med ett förutspått e-värde på 2e-118. 82 % identitetsmatchning är tillräckligt för att göra jämförelser av ZC3H12B och Mcpip1 (ZC3H12A) zinkfingerkonserverade domäner, som båda förutspås bestå av betasträngar och alfahelixar.
Modifiering efter översättning
Phobius-programmet förutspådde icke-cytoplasmatisk proteinplacering. NetPhos 2.0. förutspådde 63 fosforyleringsställen i ZC3H12B, som är markerade på den konceptuella översättningen. YinOYang1.2. förutspådde tre 0-Beta-GlcNAc-bindningsställen, som konkurrerar med fosforyleringsställen. 0-Beta-GlcNAc är förmodligen den enda typen av glykosylering som förekommer i cellkärnan och/eller cytoplasman. Det finns en anmärkningsvärd koppling mellan antigenaktivering av lymfocyter och dynamisk 0-B-glykosylering i nukleära proteiner (Hart och Akimoto). NetNGlyc förutspådde glykosyleringsställen; emellertid uteslöts dessa ställen eftersom proteinet sannolikt är nukleärt och inte skulle genomgå denna form av glykosylering. Det fanns inga förutsagda acetyleringsställen vid N-terminalen av proteinet. Detta är ovanligt eftersom ungefär 85 % av mänskliga proteiner acetyleras vid N-terminalen för syntes, stabilisering och lokalisering av proteiner (Van Damm et al.). Inga positiva, negativa eller blandade laddningskluster närvarande. Inga hydrofoba segment upptäcktes (SAPS SDSC Biology Workbench). MitoProtII upptäckte inga mitokondrierexportsignaler. Dessa post-translationella tester tyder på att proteinet är beläget i kärnan och genomgår dynamisk fosforylering och 0-Beta-GlcNAc-modifieringar.
Evolution
Utvalda domäner av ZC3H12B är bevarade i de flesta ryggradsdjur, leddjur och annelider. Det finns inga konserverade domäner i domäner av bakterier eller arkéer. Det fanns inte signifikant konserverade domäner i jäst, växter eller protister.
Paraloger
Det finns tre paraloger av ZC3H12B som är i samma zinkfingerfamilj av CCCH-typ, alla som bibehåller mer än 50 % identitet med ZC3H12B baserat på BLAST-analys (NCBI).
| namn | Arter | NCBI anslutningsnummer | Längd (AA) | Protein identitet |
|---|---|---|---|---|
| ZC3H12B | Homo sapiens | NM_001010888.3 | 836aa | 100 % |
| ZC3H12A | Homo sapiens | NM_025079.2 | 599aa | 68 % |
| ZC3H12C | Homo sapiens | NM_033390.1 | 883aa | 53 % |
| ZC3H12D | Homo sapiens | NM_207360.2 | 527aa | 61 % |
Ortologer
ZC3H12B är bevarad i däggdjur, fåglar, insekter och nematoder (BLAST). Se tabellen nedan för en sammanfattning av ortologer av ZC3H12B hos människor.
| Arter | Artens vanliga namn | Divergens (MYA) | NCBI-accessionsnummer (protein) | Längd (aminosyror) | Protein identitet | Likhet | |
|---|---|---|---|---|---|---|---|
| Homo sapiens | Mänsklig | n/a | NP_001010888.3 | 836aa | 100 % | 100 % | |
| Paniskus | Schimpans | 6.3 | XP_003816967.1 | 836aa | 99 % | 99 % | |
| Pongo abelii | Orangutang | 15.7 | XP_002831786.1 | 836aa | 99 % | 99 % | |
| Macaca mulatta | Rhesusapa | 29 | XP_002806307.1 | 836aa | 99 % | 99 % | |
| Callithrix jacchus | Marmoset | 42,6 | XP_002762992.2 | 836aa | 98 % | 98 % | |
| Mus musculus | Mus | 92,3 | NP_001030079.2 | 835aa | 91 % | 94 % | |
| Sus scrofa | Gris | 94,2 | XP_003360389.1 | 836aa | 93 % | 96 % | |
| Gallus gallus | Kyckling | 296 | XP_003641177.1 | 837aa | 77 % | 85 % | |
| Chrysemys picta bellii | Målad sköldpadda | 296 | XP_005279572.1 | 838aa | 78 % | 86 % | |
| Oryzias latipes | Medaka | 400,1 | XP_004076599.1 | 845aa | 67 % | 77 % | |
| Gadus morhua | Atlantisk torsk | 400,1 | AFK76491.1 | 842aa | 29 % | 44 % | |
| Danio rerio | Zebrafisk | 400,1 | XP_001342172.3 | 982aa | 68 % | 77 % | |
| Petromyzon marinus | Nejonöga | 535,7 | ABO21295.1 | 222aa | 44 % | 58 % | |
| Branchiostoma floridae | Lancelet | 713,2 | XP_002598834.1 | 492aa | 66 % | 79 % | |
| Ciona intestinalis | Vas mantel | 722,5 | XP_002125834.1 | 863aa | 54 % | 66 % | |
| Strongylocentrotus purpuratus | Lila sjöborre | 742,9 | XP_787030.3 | 974aa | 58 % | 72 % | |
| Aplysiomorpha californica | Havshare | 782,7 | XP_005113312.1 | 1269aa | 51 % | 69 % | |
| Drosophila grimshawi | Hawaii fruktfluga | 782,7 | XP_001994140.1 | 548aa | 51 % | 69 % | |
| Anopheles gambiae | Mygga | 782,7 | XP_321880 | 637aa | 59 % | 75 % | |
| Apis mellifera | Honungsbi | 782,7 | XP_397264 | 652aa | 58 % | 73 % | |
| Caenorhabditis elegans | Rundmask (nematod) | 937,5 | NP_491985.4 | 634aa | 46 % | 64 % |
Uttryck och funktion
Mikroarrayer i normal vävnadsuttrycksprofilering visade ökat uttryck av genen i bukspottkörteln, prostata, hjärna, ryggmärg och tymus (GEO). Till skillnad från dess paraloger uttrycks det inte i makrofagaktiverade vävnader, vilket indikerar det paraloga förhållandet till det inflammatoriska svaret (Liang et al. 2008). ZC3H12B uttrycks övergående i hjärna, tymus och testikelvävnader (EST).
Samspel
Förutspådda interaktioner av Ingenuity Systems visade inga läkemedelsmålmolekyler i vägen och inga kända läkemedelsmål. De listade toppfunktionerna och sjukdomarna var cancer, organismskador och avvikelser, sjukdomar i reproduktionssystemet. Flera miRNA- interaktioner förutspåddes. De förutsagda miRNA-målen har ännu inte matchats till ZC3H12B-sekvensen och det är oklart om det finns en interaktion mellan de två. Tester som Forster Resonance Energy Transfer (FRET), co-immunoprecipitation, tvåhybridscreening, hydropatisk komplementaritet, kluster-mikroarray och ChiP skulle kunna användas i framtiden för att testa för nya protein/kromatininteraktioner med ZC3H12B.
Klinisk signifikans
Borttagningar av Xq12-lokuset har resulterat i flera störningar såsom androgenokänslighet , känslighet för prostatacancer , spinal och bulbar muskelatrofi hos Kennedy och mental retardation ; dock har inget samband hittats mellan dessa sjukdomar och ZC3H12B (NCBI).
Vidare läsning
- Liang J, Wang J, Azfer A, Song W, Tromp G, Kolattukudy PE, Fu M (mars 2008). "En ny CCCH-zinkfingerproteinfamilj reglerar proinflammatorisk aktivering av makrofager" . Journal of Biological Chemistry . 283 (10): 6337–46. doi : 10.1074/jbc.m707861200 . PMID 18178554 .
- Van Damme P, Hole K, Pimenta-Marques A, Helsens K, Vandekerckhove J, Martinho RG, Gevaert K, Arnesen T (juli 2011). "NatF bidrar till en evolutionär förändring av protein N-terminal acetylering och är viktig för normal kromosomsegregation. " PLOS Genetik . 7 (7): e1002169. doi : 10.1371/journal.pgen.1002169 . PMC 3131286 . PMID 21750686 .