TOPPAR
| Utvecklare | Bioinformatics Solutions Inc |
|---|---|
| Stabil frisättning | PEAKS 8,5 / 12 oktober 2017 |
| Operativ system | Windows |
| Typ | Mass Spec Protein Identification, De Novo Sequencing, Databas Search & Quantification |
| Licens | Proprietär kommersiell programvara |
| Hemsida | http://www.bioinfor.com/ |
PEAKS är ett proteomikprogram för tandemmasspektrometri designat för peptidsekvensering , proteinidentifiering och kvantifiering.
Beskrivning
PEAKS används ofta för peptididentifiering (Protein ID) genom de novo peptidsekvensering assisterad sökmotordatabassökning . PEAKS har också integrerat PTM- och mutationskarakterisering genom automatisk peptidsekvenstaggbaserad sökning (SPIDER) och PTM-identifiering.
PEAKS tillhandahåller en komplett sekvens för varje peptid, konfidenspoäng för individuella aminosyratilldelningar, enkel rapportering för analys med hög genomströmning, bland annat.
Programvaran har förmågan att jämföra resultat från flera sökmotorer. PEAKS inChorus kommer att korskontrollera testresultat automatiskt med andra protein-ID-sökmotorer, som Sequest, OMSSA, X!Tandem och Mascot. Detta tillvägagångssätt skyddar mot falska positiva peptidtilldelningar.
PEAKS Q är ett tilläggsverktyg för proteinkvantifiering, stödjande märkning ( ICAT , iTRAQ , SILAC , TMT , 018 , etc.) och etikettfria tekniker.
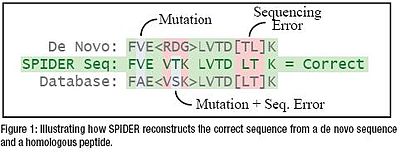
SPIDER är ett sekvenstaggbaserat sökverktyg inom PEAKS, som hanterar möjliga överlappningar mellan de novo sekvenseringsfelen och homologimutationerna. Den rekonstruerar den verkliga peptidsekvensen genom att kombinera både de novo-sekvenstaggen och homologen, automatiskt och effektivt.
En samling algoritmer som används inom programvaran PEAKS har anpassats och konfigurerats till ett specialiserat projekt, PEAKS AB, som har visat sig vara den första metoden för automatisk monoklonal antikroppssekvensering.