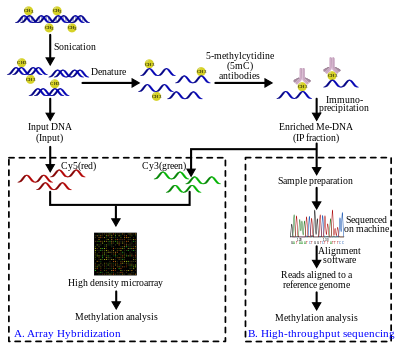
Metylerad DNA-immunfällning
Methylated DNA immunoprecipitation (MeDIP eller mDIP) är en storskalig ( kromosom- eller genomomfattande ) reningsteknik inom molekylärbiologi som används för att berika metylerade DNA-sekvenser . Den består av isolering av metylerade DNA-fragment via en antikropp framtagen mot 5-metylcytosin (5mC). Denna teknik beskrevs först av Weber M. et al. 2005 och har hjälpt till att bana väg för livskraftiga bedömningsinsatser på metylomnivå, eftersom den renade fraktionen av metylerat DNA kan matas in i DNA-detektionsmetoder med hög genomströmning såsom högupplösta DNA -mikroarrayer ( MeDIP-chip) eller nästa generations sekvensering (MeDIP-seq). Ändå är förståelsen av metylomen rudimentär; dess studie kompliceras av det faktum att, liksom andra epigenetiska egenskaper, mönster varierar från celltyp till celltyp.
Bakgrund
DNA-metylering , som hänvisar till den reversibla metyleringen av 5-positionen av cytosin av metyltransferaser , är en stor epigenetisk modifiering i flercelliga organismer. Hos däggdjur sker denna modifiering främst vid CpG-ställen , som i sin tur tenderar att samlas i regioner som kallas CpG-öar . Det finns en liten del av CpG-öarna som kan överlappa eller vara i närheten av promotorregioner för transkriptionsstartställen. Modifieringen kan också förekomma vid andra ställen, men metylering vid någon av dessa ställen kan undertrycka genuttryck genom att antingen interferera med bindningen av transkriptionsfaktorer eller modifiera kromatinstrukturen till ett repressivt tillstånd.
Sjukdomstillståndsstudier har till stor del drivit ansträngningen att förstå rollen av DNA-metylering. För närvarande ligger det stora forskningsintresset i att undersöka sjukdomstillstånd som cancer för att identifiera regioner av DNA:t som har genomgått omfattande metyleringsförändringar. Generna som finns i dessa regioner är av funktionellt intresse eftersom de kan erbjuda en mekanistisk förklaring till de underliggande genetiska orsakerna till en sjukdom. Till exempel visades det onormala metyleringsmönstret hos cancerceller initialt vara en mekanism genom vilken tumörsuppressorliknande gener tystas, även om det senare observerades att ett mycket bredare spektrum av gentyper påverkas.
Andra tekniker
Det finns två metoder för metyleringsanalys: typning och profileringsteknik. Typningsteknologier är inriktade på ett litet antal loci över många prover och involverar användning av tekniker som PCR , restriktionsenzymer och masspektrometri . Profileringsteknologier som MeDIP är inriktade på en genom- eller metylom- omfattande bedömning av metylering; detta inkluderar genomisk genomsökning (RLGS) och bisulfitomvandlingsbaserade metoder, som bygger på behandling av DNA med bisulfit för att omvandla ometylerade cytosinrester till uracil .
Andra teknikers begränsningar
Andra metoder för kartläggning och profilering av metylomen har varit effektiva men är inte utan sina begränsningar som kan påverka upplösning, genomströmningsnivå eller experimentella variationer. Till exempel begränsas RLGS av antalet restriktionsställen i genomet som kan vara mål för restriktionsenzymet; typiskt sett kan maximalt ~4100 landmärken bedömas. Bisulfitsekvenseringsbaserade metoder, trots möjlig ennukleotidupplösning, har en nackdel: omvandlingen av ometylerat cytosin till uracil kan vara instabilt. Dessutom, när bisulfitomvandling kopplas med DNA-mikroarrayer för att detektera bisulfitkonverterade ställen, är den reducerade sekvenskomplexiteten hos DNA ett problem. Mikroarrayer som kan profilera hela genomet blir svåra att designa eftersom färre unika sonder är tillgängliga.
Metoder
Följande avsnitt beskriver metoden för MeDIP i kombination med antingen högupplöst array-hybridisering eller high-throughput-sekvensering. Varje DNA-detektionsmetod kommer också kortfattat att beskriva bearbetning och analys efter laboratoriet. Olika efterbearbetning av rådata krävs beroende på vilken teknik som används för att identifiera de metylerade sekvenserna. Detta är analogt med data som genereras med ChIP-chip och ChIP-seq .
Metylerad DNA-immunfällning (MeDIP)
Genomiskt DNA extraheras ( DNA-extraktion ) från cellerna och renas. Det renade DNA:t utsätts sedan för sonikering för att klippa det till slumpmässiga fragment. Denna ultraljudsprocess är snabb, enkel och undviker fördomar av restriktionsenzym . De resulterande fragmenten sträcker sig från 300 till 1000 baspar (bp) i längd, även om de vanligtvis är mellan 400 och 600 bp. Den korta längden av dessa fragment är viktig för att erhålla adekvat upplösning, förbättra effektiviteten av nedströmssteget i immunoutfällning och reducera fragmentlängdseffekter eller fördomar. Storleken på fragmentet påverkar också bindningen av 5-metyl-cytidin (5mC) antikropp eftersom antikroppen behöver mer än bara en enda 5mC för effektiv bindning. För att ytterligare förbättra bindningsaffiniteten för antikropparna denatureras DNA-fragmenten för att producera enkelsträngat DNA. Efter denaturering inkuberas DNA:t med monoklonala 5mC-antikroppar. Den klassiska immunoutfällningstekniken tillämpas sedan: magnetiska pärlor konjugerade till anti-mus- IgG används för att binda anti-5mC-antikropparna, och obundet DNA avlägsnas i supernatanten. För att rena DNA proteinas K för att smälta antikropparna och frigöra DNA, som kan samlas in och förberedas för DNA-detektion.
För mer information om experimentstegen, se.
MeDIP och array-baserad hybridisering (MeDIP-chip)
En del av det ingående DNA:t som erhålls efter ultraljudssteget ovan är märkt med cyanin -5 (Cy5; rött) deoxicytosintrifosfat medan det metylerade DNA:t, anrikat efter immunoutfällningssteget, är märkt med cyanin -3 (Cy3; grönt) . De märkta DNA-proverna samhybridiseras på en 2-kanals genomisk mikroarray med hög densitet för att undersöka närvaro och relativa kvantiteter. Syftet med denna jämförelse är att identifiera sekvenser som visar signifikanta skillnader i hybridiseringsnivåer, och därigenom bekräfta att sekvensen av intresse är berikad. Arraybaserad identifiering av MeDIP-sekvenser är begränsad till arraydesignen. Som ett resultat är upplösningen begränsad till sonderna i arraydesignen. Det finns ytterligare standardsteg som krävs i signalbehandling för att korrigera för hybridiseringsproblem som brus, vilket är fallet med de flesta array-tekniker.
Se för mer information.
MeDIP och high-throughput sekvensering (MeDIP-seq)
MeDIP-seq-metoden, dvs kopplingen av MeDIP med nästa generations, kortlästa sekvenseringsteknologier som 454 pyrosequencing eller Illumina (Solexa), beskrevs först av Down et al. 2008. Sekvenseringen med hög genomströmning av de metylerade DNA-fragmenten ger ett stort antal korta avläsningar (36-50 bp eller 400 bp, beroende på teknologin). De korta läsningarna är anpassade till ett referensgenom med hjälp av anpassningsprogram som Mapping and Assembly with Quality ( Maq ), som använder en Bayesiansk metod, tillsammans med bas- och mappningskvaliteter för att modellera felsannolikheter för anpassningarna. Läsningarna kan sedan utökas för att representera ~400 till 700 bp fragmenten från ultraljudssteget. Täckningen av dessa utökade läsningar kan användas för att uppskatta metyleringsnivån i regionen. En genomwebbläsare som Ensembl kan också användas för att visualisera data.
Validering av tillvägagångssättet för att bedöma kvaliteten och noggrannheten hos data kan göras med kvantitativ PCR . Detta görs genom att jämföra en sekvens från MeDIP-provet mot en ometylerad kontrollsekvens. Proverna körs sedan på en gel och bandintensiteterna jämförs. Den relativa intensiteten fungerar som vägledning för att hitta berikning. Resultaten kan också jämföras med MeDIP-chipresultat för att avgöra vilken täckning som behövs.
Nedströms bioinformatikanalys
Uppskattningarna av DNA-metyleringsnivån kan förvirras av olika tätheter av metylerade CpG-ställen över genomet när man observerar data genererade av MeDIP. Detta kan vara problematiskt för att analysera CpG-fattiga (lägre densitet) regioner. En anledning till detta densitetsproblem är dess effekt på effektiviteten av immunoutfällning. I sin studie har Down et al. utvecklat ett verktyg för att uppskatta absoluta metyleringsnivåer från data genererad av MeDIP genom att modellera densiteten av metylerade CpG-ställen. Detta verktyg kallas Bayesian tool for methylation analysis (Batman) . Studien rapporterar täckningen av ~90% av alla CpG-ställen i promotorer, genkodande regioner, öar och regulatoriska element där metyleringsnivåer kan uppskattas; detta är nästan 20 gånger bättre täckning än någon tidigare metod.
Studier som använder MeDIP-seq eller MeDIP-chip är båda genomomfattande tillvägagångssätt som har det gemensamma målet att erhålla den funktionella kartläggningen av metylomen. När regioner av DNA-metylering väl har identifierats kan ett antal bioinformatiska analyser tillämpas för att besvara vissa biologiska frågor. Ett uppenbart steg är att undersöka gener som finns i dessa regioner och undersöka den funktionella betydelsen av deras förtryck. Till exempel kan tystnad av tumörsuppressorgener i cancer hänföras till DNA-metylering. Genom att identifiera mutationshändelser som leder till hypermetylering och efterföljande repression av kända tumörsuppressorgener kan man mer specifikt karakterisera de bidragande faktorerna till orsaken till sjukdomen. Alternativt kan man identifiera gener som är kända för att vara normalt metylerade men, som ett resultat av någon mutationshändelse, inte längre tystas.
Man kan också försöka undersöka och identifiera om någon epigenetisk regulator har påverkats såsom DNA-metyltransferas (DNMT); i dessa fall kan anrikningen vara mer begränsad.
Genuppsättningsanalys (till exempel med hjälp av verktyg som DAVID och GoSeq) har visat sig vara allvarligt partisk när den tillämpas på metyleringsdata med hög genomströmning (t.ex. MeDIP-seq och MeDIP-ChIP); det har föreslagits att detta kan korrigeras genom att använda provetikettpermutationer eller använda en statistisk modell för att kontrollera för skillnader i antalet CpG-sonder/CpG-ställen som riktar sig mot varje gen.
Begränsningar för MeDIP
Begränsningar att notera vid användning av MeDIP är typiska experimentella faktorer. Detta inkluderar kvaliteten och korsreaktiviteten hos 5mC-antikroppar som används i proceduren. Vidare involverar DNA-detektionsmetoder (dvs. arrayhybridisering och sekvensering med hög genomströmning) vanligtvis väl etablerade begränsningar. Speciellt för arraybaserade procedurer, som nämnts ovan, är sekvenser som analyseras begränsade till den specifika arraydesign som används.
De flesta typiska begränsningarna för nästa generations sekvensering med hög genomströmning gäller. Problemet med anpassningsnoggrannhet till repetitiva regioner i genomet kommer att resultera i mindre exakt analys av metylering i dessa regioner. Som nämnts ovan representerar korta avläsningar (t.ex. 36-50 bp från en Illumina Genome Analyzer ) en del av ett klippt fragment när det är inriktat med genomet; därför kan det exakta metyleringsstället falla var som helst inom ett fönster som är en funktion av fragmentstorleken. I detta avseende har bisulfitsekvensering mycket högre upplösning (ned till ett enda CpG-ställe; enkel nukleotidnivå). Men denna upplösningsnivå kanske inte krävs för de flesta tillämpningar, eftersom metyleringsstatusen för CpG-ställen inom < 1000 bp har visat sig vara signifikant korrelerad.
Tillämpningar av MeDIP
- Weber et al. 2005 fastställde att den inaktiva X-kromosomen hos kvinnor är hypermetylerad på en kromosom bred nivå med hjälp av MeDIP kopplat med mikroarray.
- Keshet et al. 2006 genomförde en studie på kolon- och prostatacancerceller med hjälp av MeDIP-chip. Resultatet är en genomomfattande analys av gener som ligger i hypermetylerade regioner samt slutsatsen att det finns en instruktiv mekanism för de novo-metylering i cancerceller.
- Zhang et al. 2006 erhöll en högupplöst metylomkartläggning i Arabidopsis med hjälp av MeDIP-chip.
- Novak et al. 2006 använde MeDIP-chipmetod för att undersöka mänsklig bröstcancer för metyleringsassocierad tystnad och observerade inaktiveringen av HOXA-genklustret