Kinemage

En kinemage (förkortning för kinetic image ) är en interaktiv grafisk vetenskaplig illustration. Det används ofta för att visualisera molekyler , särskilt proteiner , även om det också kan representera andra typer av 3-dimensionell data (som geometriska figurer, sociala nätverk eller tetraedrar av RNA-bassammansättning ). Kinemage-systemet är designat för att optimera användarvänlighet, interaktiv prestanda och uppfattningen och kommunikationen av detaljerad 3D-information. Kinemage-informationen lagras i en textfil, läsbar för människor och maskin, som beskriver hierarkin för visningsobjekt och deras egenskaper, och inkluderar valfri förklarande text. Kinemage-formatet är en definierad kemisk MIME-typ av 'chemical/x-kinemage' med filtillägget ' .kin'.
Tidig historia
Kinebilder utvecklades först av David Richardson vid Duke University School of Medicine , för Protein Societys tidskrift Protein Science som hade premiär i januari 1992. Under de första 5 åren (1992–1996) inkluderade varje nummer av Protein Science ett tillägg på diskett med interaktiv, kinemage 3D-datorgrafik för att illustrera många av artiklarna, plus Mage-mjukvaran ( plattformsoberoende , gratis, öppen källkod ) för att visa dem; tilläggsmaterial för kinemage finns fortfarande tillgängligt på tidskriftens webbplats. Mage och RasMol var de första allmänt använda makromolekylära grafikprogrammen som stödde interaktiv visning på persondatorer . Kinebilder används för undervisning och för lärobokstillägg, individuell utforskning och analys av makromolekylära strukturer.

Forskning använder
På senare tid, med tillgången till ett mycket bredare utbud av andra molekylära grafiska verktyg, har presentationsanvändningen av kinemages tagits om av en mängd olika forskningsanvändningar, samtidigt med nya displayfunktioner och med utvecklingen av programvara som producerar kinemage-formatutdata från andra typer av molekylära beräkningar. All-atom kontaktanalys lägger till och optimerar explicita väteatomer och använder sedan fläckar av punktyta för att visa vätebindningen , van der Waals och steriska sammandrabbningar mellan atomer. Resultaten kan användas visuellt (i kinabilder) och kvantitativt för att analysera de detaljerade interaktionerna mellan molekylära ytor, mest omfattande i syfte att validera och förbättra de molekylära modellerna från experimentell röntgenkristallografidata . Både Mage och KiNG (se nedan) har förbättrats för kinematikvisning av data i högre än 3 dimensioner (flytta mellan vyer i olika 3D-projektioner, färglägga och välja kandidatkluster av datapunkter och byta till en parallell koordinatrepresentation) , används till exempel för att definiera kluster av gynnsamma RNA-ryggradskonformationer i det 7-dimensionella utrymmet av ryggradsdiedriska vinklar mellan en ribos och nästa.
Internetanvändning
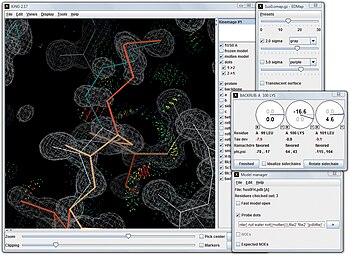
KiNG är en kinemage viewer med öppen källkod, skriven på programmeringsspråket Java av Ian Davis och Vincent Chen, som kan fungera interaktivt antingen fristående på en användarmaskin utan nätverksanslutning eller som en webbtjänst på en webbsida . Den interaktiva karaktären hos kinebilder är deras primära syfte och attribut. För att uppskatta deras natur har demonstrationen KiNG i webbläsaren två exempel som kan flyttas runt i 3D, plus instruktioner för hur man bäddar in en kinebild på en webbsida. Figuren nedan visar KiNG som används för att omforma en lysinsidokedja i en högupplöst kristallstruktur. KiNG är en av tittarna som finns på varje struktursida på Protein Data Bank-webbplatsen och visar valideringsresultat i 3D på MolProbity-webbplatsen. Kinebilder kan också visas i uppslukande virtuell verklighetssystem , med KinImmerse-mjukvaran med öppen källkod. All mjukvara för kinemage display och all-atom kontakt är tillgänglig gratis och öppen källkod på kinemages webbplats .
Se även
externa länkar
- Officiell webbplats , Duke University original, med exempel och programvara
- kinemage exempel i en webbläsare
- RCSB Protein Data Bank
- MolProbity: strukturvalidering, med KiNG on-line kinesbilder