ChIA-PET
Kromatininteraktionsanalys genom paired-end-tag-sekvensering ( ChIA-PET eller ChIA-PETS ) är en teknik som inkluderar kromatinimmunoutfällning (ChIP)-baserad anrikning, kromatin-närhetsligering, paired-end-taggar och högkapacitetssekvensering för att bestämma novocing. långväga kromatininteraktioner över hela genomet.
Gener kan regleras av regioner långt från promotorn såsom regulatoriska element, isolatorer och gränselement och transkriptionsfaktorbindningsställen (TFBS) . Att avslöja samspelet mellan regulatoriska regioner och genkodande regioner är väsentligt för att förstå mekanismerna som styr genreglering vid hälsa och sjukdom (Maston et al., 2006). ChIA-PET kan användas för att identifiera unika, funktionella kromatininteraktioner mellan distala och proximala regulatoriska transkriptionsfaktorbindningsställen och promotorerna för generna de interagerar med.
ChIA-PET kan också användas för att reda ut mekanismerna för genomkontroll under processer som celldifferentiering , proliferation och utveckling . Genom att skapa ChIA-PET- interaktomkartor för DNA-bindande regulatoriska proteiner och promotorregioner kan vi bättre identifiera unika mål för terapeutisk intervention (Fullwood & Yijun, 2009).
Metodik
ChIA-PET-metoden kombinerar ChIP-baserade metoder och kromosomkonformationsinfångning (3C)-baserade metoder, för att utöka kapaciteten för båda metoderna. ChIP-Sequencing (ChIP-Seq) är en populär metod som används för att identifiera TFBS medan 3C har använts för att identifiera långväga kromatininteraktioner. Oberoende lider båda av begränsningar när det gäller att identifiera de-novo långväga interaktioner genomet. Medan ChIP-Seq kan identifiera TFBS-genomfattande, ger den endast linjär information om proteinbindningsställen längs kromosomerna (men inte interaktioner mellan dem), och kan drabbas av högt genomiskt bakgrundsljud (falskt positiva). Medan 3C kan analysera icke-linjära kromatininteraktioner på lång räckvidd, kan den inte användas genomet bred och, liksom ChIP-Seq, lider den också av höga nivåer av bakgrundsljud. Eftersom bruset ökar i förhållande till avståndet mellan interagerande regioner (max 100kb), krävs mödosamma och tråkiga kontroller för noggrann karakterisering av kromatininteraktioner. Till skillnad från 3C som är en lokusspecifik interaktionsprofileringsmetod, har alternativa metoder som Hi-C etablerats för att profilera interaktioner genom hela genomet. Trots helgenomprofileringsmetoder för både TFBS och långdistansinteraktioner möjliggör kombinationen av tillvägagångssätt med ChIA-PET-metoden identifiering av genomiska områden där proteinet av intresse är bundet såväl som den genomiska regionen som den interagerar med.
ChIA-PET-metoden löser framgångsrikt problemen med ospecifikt interaktionsbrus som finns i ChIP-Seq genom att sonikera ChIP-fragmenten för att separera slumpmässiga bilagor från specifika interaktionskomplex. Nästa steg, som kallas anrikning, minskar komplexiteten för genomomfattande analys och lägger till specificitet till kromatininteraktioner bundna av förutbestämda TF: er (transkriptionsfaktorer). Förmågan hos 3C-metoder för att identifiera långväga interaktioner är baserad på teorin om närhetsligering. När det gäller DNA-interligering har fragment som är bundna av vanliga proteinkomplex större kinetiska fördelar under utspädda förhållanden än de som diffunderar fritt i lösning eller förankrade i olika komplex. ChIA-PET drar fördel av detta koncept genom att inkorporera länksekvenser på de fria ändarna av DNA-fragmenten bundna till proteinkomplexen. För att bygga upp anslutningsmöjlighet för fragmenten bundna av regulatoriska komplex ligeras länksekvenserna under nukleär närhetsligering. Därför kan produkterna från linkerkopplad ligering analyseras genom PET-sekvensering med ultrahög genomströmning och kartläggas till referensgenomet . Eftersom ChIA-PET inte är beroende av specifika platser för detektion som 3C och 4C är, tillåter det opartisk, genomomfattande de-novo-detektion av kromatininteraktioner. Jämfört med Hi-C begränsar användningen av en antikroppsneddragning antalet sekvenserade fragment till kromatininteraktioner bundna av proteinet av intresse, vilket också kan underlätta dataanalysen.
Arbetsflöde
Wet-lab-del av arbetsflödet:
- Formaldehyd används för att tvärbinda DNA-proteinkomplexen. Sonikering används för att bryta upp kromatinet och även för att minska ospecifika interaktioner.
- En specifik valfri antikropp används för att berika protein-av intresse-bundna kromatinfragment. ChIP-material bundet av antikroppen används för att konstruera ChIA-PET.
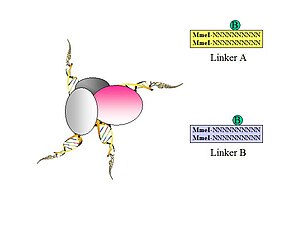
- Figur 1. Biotinylerade oligonukleotidhalvlänkare innehållande flankerande Mmel-ställen används för att koppla ihop närliggande ligerade DNA-fragment. Två olika linkers utformas (A och B) med specifika nukleotidstreckkoder (CG eller AT) för var och en av de två länksekvenserna (detta kommer att möjliggöra identifieringen av den chimära ligeringsprodukten som beskrivs i figur 5.).
- Figur 2. Länkarna ligeras till de bundna DNA-fragmenten.
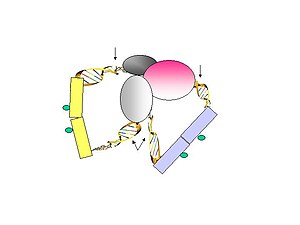
- Figur 3. Linkerfragmenten ligeras på ChIP-pärlorna under utspädda betingelser. Det renade DNA:t spjälkas sedan av Mmel, som skär på ett avstånd från dess igenkänningsställe för att frigöra tag-linker-tag-strukturen.
- Figur 4. De biotinylerade PET:erna immobiliseras sedan på streptavidinkonjugerade magnetiska pärlor.
- Figur 5. PET-sekvenser med AA (CG/CG) och BB (AT/AT) länkstreckkodssammansättning anses vara möjliga intrakomplexa ligeringsprodukter, medan PET-sekvenserna med AB (CG/AT) länksammansättning anses vara härledda från chimära ligeringsprodukter mellan DNA-fragment bundna i olika kromatinkomplex.
Torrlabbdelen av arbetsflödet:
PET-extraktion, kartläggning och statistiska analyser
PET-taggarna extraheras och mappas till det mänskliga referensgenomet i silico .
Identifiering av ChIP-berikade toppar (bindningsställen)
Självligerad PET används för att identifiera ChIP-berikade platser eftersom de ger den mest tillförlitliga kartläggningen (20 + 20 bit/s) till referensgenomet.
Chip-anrikningsalgoritm för att finna topp
En kallad topp anses vara ett bindningsställe om det finns flera överlappande självligerade PET. Den falska upptäcktsfrekvensen (FDR) bestäms med hjälp av statistiska simuleringar för att uppskatta den slumpmässiga bakgrunden för PET-härledda virtuella DNA-överlappningar och det uppskattade bakgrundsbruset.
Filtrering av repetitivt DNA (påverkar ospecifik bindning)
Satellitregioner och bindningsställen som finns i regioner med allvarliga strukturella variationer tas bort.
Antal chip-anrikningar
Antalet självligerings- och interligerings-PET (inom +250 bp-fönster) rapporteras på varje plats. Det totala antalet självligerade och interligerade PET vid en specifik plats kallas för ChIP-anrikningsantalet.
Figur 6. PET-klassificering: Unikt anpassade PET-sekvenser kan klassificeras efter om de härrör från ett DNA-fragment eller två DNA-fragment.
- Självligerande PET
Om de två taggarna av en PET kartläggs på samma kromosom med det genomiska intervallet inom intervallet av ChIP DNA-fragment (mindre än 3 Kb), med förväntad självligeringsorientering och på samma sträng, anses de härröra från en självligering av ett enda ChIP-DNA-fragment och betraktas som en självligerings-PET.
- Inter-ligation PETs
Om en PET inte passar in i dessa kriterier, är PET troligen ett resultat av en ligeringsprodukt mellan två DNA-fragment och hänvisas till som en interligerings-PET. De två taggarna av en interligerings-PET har inte fixerade taggarorienteringar, kanske inte finns på samma strängar, kan ha någon genomisk spännvidd och kanske inte mappas till samma kromosom.
- Intrakromosomala interligerings-PET
Om de två taggarna för en interligerings-PET är kartlagda i samma kromosom men med ett spännvidd > 3 Kb i vilken orientering som helst, så kallas dessa PET:er för intrakromosomala interligerings-PET.
- Interkromosomala interligerings-PET
PETs som är mappade till olika kromosomer kallas interkromosomala interligerings-PET.
Figur 7. Föreslagen mekanism som visar hur distala regulatoriska element kan initiera långväga kromatininteraktioner som involverar promotorregioner av målgener.
Interaktionerna bildar DNA-loopstrukturer med flera TFBS vid förankringscentret. Små slingor kan paketera gener nära förankringscentret i ett tätt underfack, vilket kan öka den lokala koncentrationen av regulatoriska proteiner för förbättrad transkriptionell aktivering. Denna mekanism kan också förbättra transkriptionseffektiviteten, vilket gör det möjligt för RNA pol II att cirkulera de täta cirkulära genmallarna. De stora interaktionsslingorna är mer benägna att länka samman avlägsna gener i vardera änden av slingan som ligger nära ankarställen för samordnad reglering, eller kan separera gener i långa loopar för att förhindra deras aktivering. Anpassad från Fullwood et al. (2009).
Styrkor och svagheter
Fördelar med ChIA-PET-metoden
- ChIA-PET har potential att vara en opartisk, helgenom- och de-novo-metod för långdistansanalys av kromatininteraktion (Fullwood & Yijun, 2009).
- Ett ChIA-PET-experiment kan tillhandahålla två globala datauppsättningar: Proteinfaktorbindningsställena (självligerade PETs); och interaktionerna mellan bindningsställena (interligerade PET).
- ChIA-PET involverar ChIP för att minska komplexiteten för genomomfattande analys och lägger till specificitet till kromatininteraktioner bundna av specifika faktorer av intresse.
- ChIA-PET är kompatibel med taggbaserade nästa generations sekvenseringsmetoder som Roche 454 pyrosequencing, Illumina GA, ABI SOLiD och Helicos.
- ChIA-PET är tillämpbart på många olika proteinfaktorer involverade i transkriptionsreglering eller kromatinstrukturell konformation.
- ChIA-PET-analys kan tillämpas på kromatininteraktioner involverade i en viss nukleär process. Genom att använda allmänna TF: er såsom RNA-polymeras II, kan det vara möjligt att identifiera alla kromatininteraktioner involverade i transkriptionsreglering. Vidare skulle användningen av proteinfaktorer involverade i DNA-replikation eller kromatinstruktur möjliggöra identifiering av alla interaktioner på grund av DNA-replikation och kromatinstrukturell modifiering (Fullwood et al., 2009).
Svagheter
- Det är väl etablerat att cis- och transregulatoriska komplex innehåller unika kombinationer av proteiner baserade på cell- och vävnadsspecifika förhållanden (Dekker et al., 2006). Även om identifiering av enstaka, funktionella TFBS är ett betydande framsteg, skulle användningen av ChIA-PET för att identifiera individuella proteiner i ett komplex kräva gissningsarbete och flera experiment för att identifiera varje interagerande protein. Detta skulle vara ett kostsamt och tidskrävande arbete.
- ChIA-PET begränsas av kvaliteten, renheten och specificiteten hos de antikroppar som används (Fullwood et al., 2009).
- ChIA-PET är beroende av identifiering av sekvenser som kan mappas till referenssekvensen (ref).
- ChIA-PET kräver användning av toppanropande datoralgoritmer för att organisera och kartlägga PET-läsningar till referensgenomet. På grund av variationer mellan programvaruplattformar kan resultaten variera beroende på vilket program som används.
- Även om repetitiva DNA-regioner kan associeras med genreglering (Polak & Domany, 2006), måste de tas bort eftersom de kan påverka data (Fullwood et al., 2009).
- Anrikning från två platser introducerar samtidigt fördomar mot omgivande regioner.
Historia
Fullwood et al. (2009), använde ChIA-PET för att detektera och kartlägga kromatininteraktionsnätverket medierat av östrogenreceptorn alfa (ER-alfa) i humana cancerceller. Den resulterande globala kromatininteraktomkartan avslöjade att avlägsna ER-alfa-bindningsställen också var förankrade till genpromotorer genom långväga kromatininteraktioner, vilket tyder på att ER-alfa fungerar genom omfattande kromatinslingor för att sammanföra gener för koordinerad transkriptionsreglering.
Analys och mjukvara
| programvara | Beskrivning |
|---|---|
| C3PET | En mjukvarusvit för bearbetning av ChIA-PET-data. Använder en icke-parametrisk Bayesiansk metod för att förutsäga kromatininteragerande proteinkomplex. [1] [2] |
| ChIA-PET-verktyg | En mjukvarusvit för bearbetning av ChIA-PET-data. [3] [4] |
| ChIA-PET2 | En mjukvarusvit för bearbetning av ChIA-PET-data. Stöder data från en mängd olika protokoll och ger kvalitetskontroll av dataanalys. [5] [6] |
| ChIA-Sig | En mjukvarusvit för bearbetning av ChIA-PET-data med hjälp av NCHG-modellen. ChIA-Sig webbplats [7] [8] |
| ELAND | Kartlägger ChIP-berikade DNA-fragment till det mänskliga referensgenomet. [9] |
| Mango | En mjukvarusvit för bearbetning av ChIA-PET-data. Slutför alla nödvändiga steg för bearbetning av ChIA-PET-datauppsättningar och tillhandahåller statistiska konfidensuppskattningar för interaktioner. [10] [11] |
| Monte Carlo-simulering | Används för att uppskatta antalet falska upptäckter. |
| Genomiska interaktioner | Ett R-paket för bearbetning av ChIA-PET- eller Hi-C-data. [12] [13] |
| GE | Ett programmeringsbibliotek för att skapa en anpassad genomwebbläsare som är kompatibel med ChIA-PET- eller Hi-C-data. [14] [15] |
| RepeatMasker | In-silico maskering av repetitiva element. [16] |
Alternativ
Kromatinimmunfällning (ChIP):
Den ursprungliga ChIP-metoden är en antikroppsbaserad teknologi som identifierar och binder proteiner selektivt för att ge information om kromatintillstånd och gentranskription . [17]
Genome Architecture Mapping (GAM):
Denna teknik eliminerar ett antal nackdelar förknippade med 3C-baserade tekniker genom att samla in tredimensionella närheter mellan valfritt antal genomiska loci .
Delad pool igenkänning av interaktioner med taggtillägg (SPRITE)
SPRITE är en teknik för att kartlägga interaktioner av högre ordning i kärnan över genomet. Detta tillvägagångssätt detekterar interaktioner som inträffar över större rumsliga avstånd och det möjliggör för genomtäckande detektion av många RNA- och DNA -interaktioner som inträffar samtidigt.
ChIA-Drop
ChIA-Drop är en enkel metod för att analysera multiplexkromatininteraktioner med hjälp av droppbaserad och streckkodslänkad sekvensering vid enmolekylär noggrannhet. Tidigare parvisa tillvägagångssätt på populationsnivå som Hi-C och ChIA-PET skiljer sig från denna teknik.
- Barski et al., (2007). Högupplöst profilering av histonmetyleringar i det mänskliga genomet. Cell. (129); 823–37.
- Dekker, (2002). Fångar kromosomkonformation. Vetenskap. (295); 1306–1311.
- Dekker, (2006). De tre "C":en för kromosomkonformation fångar: kontroller, kontroller, kontroller. Nat. Metoder. (3); 17–21.
- Fullwood et al., (2009). En östrogen-receptor-α-bunden human kromatininteraktom. Natur. (462); 58–64.
- Fullwood & Yijun, (2009). ChIP-baserade metoder för identifiering av långväga kromatininteraktioner. J Cell Biochem. 107(1); 30–39.
- Johnson et al., (2007). Genomomfattande kartläggning av in vivo protein-DNA-interaktioner. Vetenskap. (316); 1497–502.
- Kuo & Allis, (1999). In vivo tvärbindning och immunfällning för att studera dynamiskt protein: DNA-föreningar i en kromatinmiljö. Metoder. (19); 425–33.
- Li, G., Fullwood, MJ, Xu, H., Mulawadi, FH, Velkov, S., Vega, V., Ariyaratne, PN, Mohamed, YB, Ooi, HS, Tennakoon, C., Wei, CL, Ruan , Y. och Sung, WK ChIA-PET-verktyg för omfattande kromatininteraktionsanalys med parad-änd-tag-sekvensering. Genome Biol, 11 (2). R22.
- Maston et al., (2006). Transkriptionsreglerande element i det mänskliga genomet. Annu. Rev: Genomik. Hum Genet. (7); 29–59.
- Polak & Domany, (2006). Alu-element innehåller många bindningsställen för transkriptionsfaktorer och kan spela en roll vid reglering av utvecklingsprocesser. BMC Genomics. (7); 133.
- Wei et al., (2006). En global karta över p53-transkriptionsfaktorbindningsställen i det mänskliga genomet. Cell. (124); 207–19.
externa länkar
- ChIA-PET Genome Browser - Denna webbläsare är till för att visa data från Fullwood et al. (2009), och inkluderar en anpassad Whole Genome Interaction Viewer som ger en makroskopisk bild av bindningsställen och interaktioner tillsammans med ett helt genomlandskap.