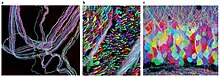
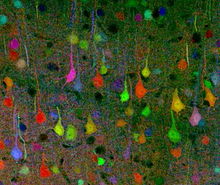
Hjärnbåge
Brainbow är en process genom vilken enskilda neuroner i hjärnan kan särskiljas från närliggande neuroner med hjälp av fluorescerande proteiner. Genom att slumpmässigt uttrycka olika förhållanden av röda, gröna och blå derivat av grönt fluorescerande protein i individuella neuroner, är det möjligt att flagga varje neuron med en distinkt färg. Denna process har varit ett stort bidrag till området för neural connectomics .
Tekniken utvecklades ursprungligen 2007 av ett team ledd av Jeff W. Lichtman och Joshua R. Sanes , båda vid Harvard University . Den ursprungliga tekniken har nyligen anpassats för användning med andra modellforskningsorganismer inklusive fruktflugan ( melanogaster ) Drosophila , zebrafisk ( Danio rerio ) och Arabidopsis thaliana .
Medan tidigare märkningstekniker möjliggjorde kartläggning av endast ett fåtal neuroner, tillåter denna nya metod att mer än 100 olika kartlagda neuroner kan belysas samtidigt och differentiellt på detta sätt. Detta leder till dess karakteristiska mångfärgade utseende på bildbehandling, förtjänar sitt namn och vinner priser i vetenskapliga fotografitävlingar. [ citat behövs ]
Historia och utveckling
Brainbow utvecklades ursprungligen av Jeff W. Lichtman och Joshua R. Sanes vid Washington University i St. Louis , även om de har flyttat till Harvard University sedan dess. Teamet konstruerade Brainbow med hjälp av en tvåstegsprocess: först genererades en specifik genetisk konstruktion som kunde kombineras i flera arrangemang för att producera en av antingen tre eller fyra färger baserat på de särskilda fluorescerande proteinerna (XFP) som implementeras. Därefter infogades flera kopior av samma transgena konstruktion i genomet av målarten, vilket resulterade i det slumpmässiga uttrycket av olika XFP-förhållanden och som sedan fick olika celler att uppvisa en mängd olika färgglada nyanser.
Brainbow skapades ursprungligen som en förbättring jämfört med mer traditionella neuroimaging- tekniker, såsom Golgi-färgning och färginjektion, som båda presenterade allvarliga begränsningar för forskare i deras förmåga att visualisera den invecklade arkitekturen hos neurala kretsar i hjärnan . Medan äldre tekniker endast kunde färga celler med ett begränsat spektrum av färger, ofta med hjälp av två- och trefärgade transgena möss för att avslöja begränsad information om neuronala strukturer, är Brainbow mycket mer flexibel genom att den har kapaciteten att fluorescerande märka individuella neuroner med upp till cirka 100 olika nyanser så att forskare kan identifiera och till och med skilja mellan dendritiska och axonala processer. Genom att avslöja sådan detaljerad information om neuronala anslutningar och mönster, ibland även in vivo, kan forskare ofta dra slutsatser om information om neuronala interaktioner och deras efterföljande inverkan på beteende och funktion. Således fyllde Brainbow tomrummet efter tidigare neuroimagingmetoder.
Med den senaste tillkomsten av Brainbow inom neurovetenskap kan forskare nu konstruera specifika kartor över neurala kretsar och bättre undersöka hur dessa relaterar till olika mentala aktiviteter och deras relaterade beteenden (dvs. Brainbow avslöjar information om sammankopplingarna mellan neuroner och deras efterföljande interaktioner som påverkar övergripande hjärnfunktionalitet). Som en ytterligare extrapolering av denna metod kan Brainbow därför också användas för att studera både neurologiska och psykologiska störningar genom att analysera skillnader i neurala kartor.
Metoder
Brainbow-tekniker förlitar sig på Cre-Lox-rekombinationen , där proteinet Cre-rekombinas driver inversion eller excision av DNA mellan loxP-ställen. Den ursprungliga Brainbow-metoden inkluderar både Brainbow-1 och Brainbow-2, som använder olika former av cre/lox-rekombination. Brainbow-3, en modifierad version av Brainbow-1, utvecklades 2013. För alla Brainbow-subtyper är uttrycket av en given XFP en stokastisk eller slumpmässig händelse.
Brainbow-1 använder DNA-konstruktioner med olika fluorescerande proteingener (XFP) separerade av mutanta och kanoniska former av loxP. Detta skapar en uppsättning ömsesidigt exklusiva excisionsmöjligheter, eftersom cre-medierad rekombination endast sker mellan identiska loxP-ställen. Efter att rekombination inträffat uttrycks det fluorescerande proteinet som finns kvar direkt efter promotorn unikt. Således kan en konstruktion med fyra XFP: er separerade av tre olika loxP-ställen, tre excisionshändelser och den ursprungliga konstruktionen producera fyra olika fluorescerande proteiner.
Brainbow-2 använder Cre-excision och inversion för att tillåta flera uttrycksmöjligheter i en given konstruktion. I ett DNA-segment med två motsatt orienterade XFP:er kommer Cre att inducera en slumpmässig inversionshändelse som lämnar ett fluorescerande protein i rätt orientering för uttryck. Om två av dessa inverterbara sekvenser är inriktade, är tre olika inversionshändelser möjliga. När excisionshändelser också beaktas, kommer ett av fyra fluorescerande proteiner att uttryckas för en given kombination av Cre-excisioner och inversioner.
Brainbow-3 behåller Brainbow-1 loxP-formatet, men ersätter RFP-, YFP- och CFP-generna med mOrange2, EGFP och mKate2. mO2, EGFP och mK2 valdes både för att deras fluorescerande excitations- och emissionsspektra överlappar minimalt, och för att de delar minimal sekvenshomologi, vilket möjliggör design av selektiva antikroppar som kan användas för att detektera dem i immunhistokemiska protokoll . Brainbow-3 tar också upp frågan om ojämn fyllning av neuroner med XFP:er genom att använda farnesylerade derivat av XFP:er, som transporteras mer jämnt till neuronala membran.
Brainbow implementeras in vivo genom att korsa två transgena organismstammar: en som uttrycker Cre-proteinet och en annan som har transfekterats med flera versioner av en loxP/XFP-konstruktion. Genom att använda flera kopior av transgenen kan XFP:erna kombineras på ett sätt som kan ge en av cirka 100 olika färger. Således är varje neuron märkt med en annan nyans baserat på dess givna kombinatoriska och stokastiska uttryck av fluorescerande proteiner.
För att belysa differentiella XFP-uttrycksmönster till en synlig form, avbildas hjärnskivor med konfokalmikroskopi . När den exponeras för en foton med dess speciella excitationsvåglängd avger varje fluorofor en signal som samlas in i en röd, grön eller blå kanal, och den resulterande ljuskombinationen analyseras med dataanalysmjukvara. Överlagring av differentiellt färgade neuroner möjliggör visuell lösgöring av komplicerade neurala kretsar.
Brainbow har övervägande testats på möss hittills; Men den grundläggande tekniken som beskrivs ovan har också modifierats för användning i nyare studier sedan tillkomsten av den ursprungliga metoden som introducerades 2007.
Möss

Mushjärnan har 75 000 000 neuroner och är mer lik en mänsklig hjärna än drosophila och andra vanliga organismer för att modellera denna teknik , såsom C. elegans . Möss var de första organismerna där Brainbow-metoden för neuroimaging framgångsrikt användes. Livet et al. (2007) utvecklade två versioner av Brainbow-möss med hjälp av Brainbow-1 och Brainbow-2, som beskrivs ovan. Genom att använda dessa metoder för att skapa en komplett karta och spåra axonerna i en musmuskel, är det nödvändigt att samla tiotusentals bilder och sammanställa dem i staplar för att skapa en komplett schematisk. Det är sedan möjligt att spåra varje motoraxon och dess synaptiska kontakter för att konstruera en komplett konnektor av muskeln.
Fler exempel på neuroner som undersökts med hjälp av Brainbow-tekniken i transgena möss finns i den motoriska nerven som innerverar öronmusklerna, axonkanalerna i hjärnstammen och hippocampus dentate gyrus .
Drosophila
Komplexiteten hos Drosophila-hjärnan, som består av cirka 100 000 neuroner, gör den till en utmärkt kandidat för att implementera neurofysiologi och neurovetenskapliga tekniker som Brainbow. Faktum är att Stefanie Hampel et al. (2011) kombinerade Brainbow i kombination med genetiska inriktningsverktyg för att identifiera individuella neuroner inom Drosophila-hjärnan och olika neuronala linjer. Ett av de genetiska inriktningsverktygen var ett GAL4/UAS binärt uttryckssystem som kontrollerar uttrycket av UAS-Brainbow och riktar uttrycket till små grupper av neuroner. Att använda "Flip Out"-metoder ökade den cellulära upplösningen av reporterkonstruktionen. Uttrycket av fluorescerande proteiner, som med den ursprungliga Brainbow, berodde på Cre-rekombination motsvarande matchade lox-ställen. Hampel et al. (2011) utvecklade också sin egen variant av Brainbow (dBrainbow), baserad på antikroppsmärkning av epitoper snarare än endogen fluorescens. Två kopior av deras konstruktion ger sex ljusa, separerbara färger. Detta, tillsammans med förenklingar i färgtilldelning, gjorde det möjligt för dem att observera banorna för varje neuron över långa avstånd. Specifikt spårade de motorneuroner från antennloben till neuromuskulära korsningar, vilket gjorde att de kunde identifiera de specifika muskelmålen för individuella neuroner.
I slutändan ger denna teknik möjligheten att effektivt kartlägga neuronkretsar i Drosophila så att forskare kan avslöja mer information om hjärnstrukturen hos detta ryggradslösa djur och hur det relaterar till dess efterföljande beteende.
Begränsningar
Som med alla neuroimaging tekniker har Brainbow ett antal begränsningar som härrör från de metoder som krävs för att utföra den. Till exempel är processen att föda upp minst två stammar av transgena djur från embryonala stamceller både tidskrävande och komplex. Även om två transgena arter skapas framgångsrikt, kommer inte alla deras avkommor att visa rekombinationen. Detta kräver alltså omfattande planering innan ett experiment utförs.
Dessutom, på grund av den slumpmässiga naturen i uttrycket av de fluorescerande proteinerna, kan forskare inte exakt kontrollera märkningen av neurala kretsar, vilket kan resultera i dålig identifiering av specifika neuroner.
Användningen av hjärnbåge i däggdjurspopulationer hämmas också av den otroliga mångfalden av nervceller i det centrala nervsystemet . Den stora tätheten av neuroner i kombination med närvaron av långa axoner gör det svårt att se större regioner av CNS med hög upplösning. Brainbow är mest användbar när man undersöker encellsupplösning mot bakgrunden av en komplex flercellig miljö. Men på grund av upplösningsgränserna för optisk mikroskopi , är det inte lätt att göra en avgörande identifiering av synaptiska kopplingar mellan neuroner. Detta problem undviks något genom att använda synaptiska markörer för att komplettera användningen av optisk mikroskopi för att se synaptiska anslutningar.